-
Notifications
You must be signed in to change notification settings - Fork 6
Commit
This commit does not belong to any branch on this repository, and may belong to a fork outside of the repository.
Merge pull request #144 from ScienceCore/batch4
Batch 4 translations
- Loading branch information
Showing
4 changed files
with
1,563 additions
and
0 deletions.
There are no files selected for viewing
343 changes: 343 additions & 0 deletions
343
...s-419/02_Software_Tools_Techniques/01_Loading_Raster_Data_from_GeoTIFF_Files.md
This file contains bidirectional Unicode text that may be interpreted or compiled differently than what appears below. To review, open the file in an editor that reveals hidden Unicode characters.
Learn more about bidirectional Unicode characters
| Original file line number | Diff line number | Diff line change |
|---|---|---|
| @@ -0,0 +1,343 @@ | ||
| --- | ||
| jupyter: | ||
| jupytext: | ||
| text_representation: | ||
| extension: .md | ||
| format_name: markdown | ||
| format_version: "1.3" | ||
| jupytext_version: 1.16.2 | ||
| kernelspec: | ||
| display_name: Python 3 (ipykernel) | ||
| language: python | ||
| name: python3 | ||
| --- | ||
|
|
||
| # Carga de datos ráster desde archivos GeoTIFF | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| Dado que la mayoría de los datos geoespaciales con los que trabajaremos en este tutorial están almacenados en archivos GeoTIFF, debemos saber cómo trabajar con esos archivos. La solución más sencilla es utilizar [rioxarray](https://corteva.github.io/rioxarray/html/index.html). Esta solución se encarga de muchos detalles complicados de forma transparente. También podemos utilizar [Rasterio](https://rasterio.readthedocs.io/en/stable) como herramienta para leer datos o metadatos de archivos GeoTIFF. Un uso adecuado de Rasterio puede marcar una gran diferencia a la hora de trabajar con archivos remotos en la nube. | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| import numpy as np | ||
| import rasterio | ||
| import rioxarray as rio | ||
| from pathlib import Path | ||
| FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book' | ||
| ``` | ||
|
|
||
| *** | ||
|
|
||
| ## [rioxarray](https://corteva.github.io/rioxarray/html/index.html) | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| `rioxarray` es un paquete que _extiende_ el paquete Xarray (hablaremos al respecto más adelante). Las principales funciones de `rioxarray` que utilizaremos en este tutorial son: | ||
|
|
||
| - `rioxarray.open_rasterio` para cargar archivos GeoTIFF directamente en estructuras Xarray `DataArray`, y | ||
| - `xarray.DataArray.rio` para proporcionar usos útiles (por ejemplo, para especificar información CRS). | ||
|
|
||
| Para acostumbrarnos a trabajar con archivos GeoTIFF, utilizaremos algunos ejemplos específicos en este cuaderno computacional y en otros posteriores. Más adelante explicaremos qué tipo de datos contiene el archivo, por el momento, solo queremos acostumbrarnos a cargar datos. | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| ### Carga de archivos en un DataArray | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| Observa en primer lugar que `open_rasterio` funciona con direcciones de archivos locales y URL remotas. | ||
|
|
||
| - Como era de esperarse, el acceso local es más rápido que el remoto. | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| %%time | ||
| LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif') | ||
| data = rio.open_rasterio(LOCAL_PATH) | ||
| ``` | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| %%time | ||
| REMOTE_URL ='https://opera-provisional-products.s3.us-west-2.amazonaws.com/DIST/DIST_HLS/WG/DIST-ALERT/McKinney_Wildfire/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif' | ||
| data_remote = rio.open_rasterio(REMOTE_URL) | ||
| ``` | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| La siguiente operación compara elementos de un Xarray `DataArray` elemento a elemento (el uso del método `.all` es similar a lo que haríamos para comparar arrays NumPy). Por lo general, esta no es una forma recomendable de comparar matrices, series, dataframes u otras estructuras de datos grandes que contengan datos de punto flotante. Sin embargo, en este caso concreto, como las dos estructuras de datos se leyeron del mismo archivo almacenado en dos ubicaciones diferentes, la comparación elemento a elemento tiene sentido. Confirma que los datos cargados en la memoria desde dos fuentes distintas son idénticos en cada bit. | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| (data_remote == data).all() # Verify that the data is identical from both sources | ||
| ``` | ||
|
|
||
| *** | ||
|
|
||
| ## [rasterio](https://rasterio.readthedocs.io/en/stable) | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| Esta sección puede omitirse si `rioxarray` funciona adecuadamente para nuestros análisis, es decir, si la carga de datos en la memoria no es prohibitiva. Cuando _no_ sea el caso, `rasterio` proporciona estrategias alternativas para explorar los archivos GeoTIFF. Es decir, el paquete `rasterio` ofrece formas de bajo nivel para cargar datos que `rioxarray` cuando sea necesario. | ||
|
|
||
| De la [documentación de Rasterio](https://rasterio.readthedocs.io/en/stable): | ||
|
|
||
| > Antes de Rasterio había una opción en Python para acceder a los diferentes tipos de archivos de datos ráster utilizados en el campo de los SIG: los enlaces de Python distribuidos con la [Biblioteca de Abstracción de Datos Geoespaciales](http://gdal.org/) (GDAL, por sus siglas en inglés de _Geospatial Data Abstraction Library_). Estos enlaces extienden Python, pero proporcionan poca abstracción para la Interface de programación de aplicaciones C (API C, por sus siglas en inglés de _Application Programming Interface_) de GDAL. Esto significa que los programas Python que los utilizan tienden a leerse y ejecutarse como programas de C. Por ejemplo, los enlaces a Python de GDAL obligan a los usuarios a tener cuidado con los punteros de C incorrectos, que pueden bloquear los programas. Esto es malo: entre otras consideraciones hemos elegido Python en vez de C para evitar problemas con los punteros. | ||
| > | ||
| > ¿Cómo sería tener una abstracción de datos geoespaciales en la biblioteca estándar de Python? ¿Una que utilizara características y modismos modernos del lenguaje Python? ¿Una que liberara a los usuarios de la preocupación por los punteros incorrectos y otras trampas de la programación en C? El objetivo de Rasterio es ser este tipo de biblioteca de datos ráster, que exprese el modelo de datos de GDAL utilizando menos clases de extensión no idiomáticas y tipos y protocolos de Python más idiomáticos, a la vez que funciona tan rápido como los enlaces de Python de GDAL. | ||
| > | ||
| > Alto rendimiento, menor carga cognitiva, código más limpio y transparente. Eso es Rasterio. | ||
| <!-- #endregion --> | ||
|
|
||
| *** | ||
|
|
||
| ### Abrir archivos con rasterio.open | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| # Show rasterio.open works using context manager | ||
| LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif') | ||
| print(LOCAL_PATH) | ||
| ``` | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| Dada una fuente de datos (por ejemplo, un archivo GeoTIFF en el contexto actual), podemos abrir un objeto `DatasetReader` asociado utilizando `rasterio.open`. Técnicamente, debemos recordar cerrar el objeto después. Es decir, nuestro código quedaría así: | ||
|
|
||
| ```{code-cell} python | ||
| ds = rasterio.open(LOCAL_PATH) | ||
| # .. | ||
| # do some computation | ||
| # ... | ||
| ds.close() | ||
| ``` | ||
|
|
||
| Al igual que con el manejo de archivos en Python, podemos utilizar un _administrador de contexto_ (es decir, una cláusula `with`) en su lugar. | ||
|
|
||
| ```python | ||
| with rasterio.open(LOCAL_PATH) as ds: | ||
| # ... | ||
| # do some computation | ||
| # ... | ||
|
|
||
| # more code outside the scope of the with block. | ||
| ``` | ||
|
|
||
| El conjunto de datos se cerrará automáticamente fuera del bloque `with`. | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| with rasterio.open(LOCAL_PATH) as ds: | ||
| print(f'{type(ds)=}') | ||
| assert not ds.closed | ||
| # outside the scope of the with block | ||
| assert ds.closed | ||
| ``` | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| La principal ventaja al utilizar `rasterio.open` en vez de `rioxarray.open_rasterio` para abrir un archivo es que este último método abre el archivo y carga inmediatamente su contenido en un `DataDarray` en la memoria. | ||
|
|
||
| Por el contrario, al utilizar `rasterio.open` se abre el archivo en su lugar y su contenido _no_ se carga inmediatamente en la memoria. Los datos del archivo _pueden_ leerse, pero esto debe hacerse explícitamente. Esto representa una gran diferencia cuando se trabaja con datos remotos. Transferir todo el contenido a través de una red de datos implica ciertos costos. Por ejemplo, si examinamos los metadatos, que suelen ser mucho más pequeños y pueden transferirse rápidamente, podemos descubrir, por ejemplo, que no está justificado mover todo un _array_ de datos a través de la red. | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| *** | ||
|
|
||
| ### Análisis de los atributos DatasetReader | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| Cuando se abre un archivo utilizando `rasterio.open`, el objeto instanciado es de la clase `DatasetReader`. Esta clase tiene una serie de atributos y métodos de interés para nosotros: | ||
|
|
||
| | | | | | ||
| | --------- | ----------- | -------- | | ||
| | `profile` | `height` | `width` | | ||
| | `shape` | `count` | `nodata` | | ||
| | `crs` | `transform` | `bounds` | | ||
| | `xy` | `index` | `read` | | ||
|
|
||
| En primer lugar, dado un `DatasetReader` `ds` asociado a una fuente de datos, el análisis de `ds.profile` devuelve cierta información de diagnóstico. | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| with rasterio.open(LOCAL_PATH) as ds: | ||
| print(f'{ds.profile=}') | ||
| ``` | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| Los atributos `ds.height`, `ds.width`, `ds.shape`, `ds.count`, `ds.nodata` y `ds.transform` se incluyen en la salida de `ds.profile`, pero también son accesibles individualmente. | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| with rasterio.open(LOCAL_PATH) as ds: | ||
| print(f'{ds.height=}') | ||
| print(f'{ds.width=}') | ||
| print(f'{ds.shape=}') | ||
| print(f'{ds.count=}') | ||
| print(f'{ds.nodata=}') | ||
| print(f'{ds.crs=}') | ||
| print(f'{ds.transform=}') | ||
| ``` | ||
|
|
||
| *** | ||
|
|
||
| ### Lectura de datos en la memoria | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| El método `ds.read` carga un _array_ del archivo de datos en la memoria. Ten en cuenta que esto se puede hacer en archivos locales o remotos. | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| %%time | ||
| with rasterio.open(LOCAL_PATH) as ds: | ||
| array = ds.read() | ||
| print(f'{array.shape=}') | ||
| ``` | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| %%time | ||
| with rasterio.open(REMOTE_URL) as ds: | ||
| array = ds.read() | ||
| print(f'{array.shape=}') | ||
| ``` | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| print(f'{type(array)=}') | ||
| ``` | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| El _array_ cargado en la memoria con `ds.read` es una matriz NumPy. Este puede ser encapsulado por un Xarray `DataArray` si proporcionamos código adicional para especificar las etiquetas de las coordenadas y demás. | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| *** | ||
|
|
||
| ### Mapeo de coordenadas | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| Anteriormente, cargamos los datos de un archivo local en un `DataArray` llamado `da` utilizando `rioxarray.open_rasterio`. | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| da = rio.open_rasterio(LOCAL_PATH) | ||
| da | ||
| ``` | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| De este modo se simplificó la carga de datos ráster de un archivo GeoTIFF en un Xarray `DataArray` a la vez que cargaban los metadatos automáticamente. En particular, las coordenadas asociadas a los píxeles se almacenaron en `da.coords` (los ejes de coordenadas predeterminados son `band`, `x` y `y` para este _array_ tridimensional). | ||
|
|
||
| Si ignoramos la dimensión extra de `band`, los píxeles de los datos ráster se asocian con coordenadas de píxel (enteros) y coordenadas espaciales (valores reales, típicamente un punto en el centro de cada píxel). | ||
|
|
||
| 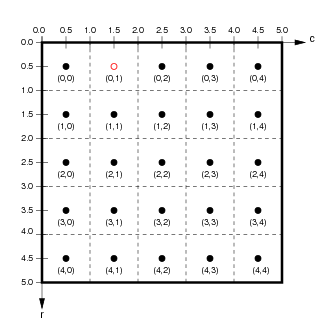 | ||
| 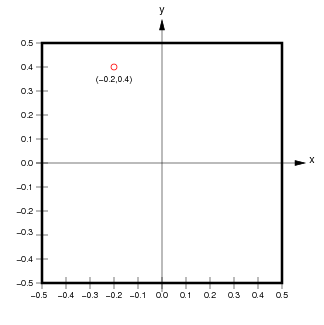 | ||
| (de `http://ioam.github.io/topographica`) | ||
|
|
||
| Los accesores `da.isel` y `da.sel` nos permiten extraer porciones del _array_ utilizando coordenadas de píxel o coordenadas espaciales, respectivamente. | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| Si utilizamos `rasterio.open` para abrir un archivo, el atributo `transform` de `DatasetReader` proporciona los detalles sobre cómo realizar la conversión entre coordenadas de píxel y espaciales. Utilizaremos esta propiedad en algunos de los casos prácticos más adelante. | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| with rasterio.open(LOCAL_PATH) as ds: | ||
| print(f'{ds.transform=}') | ||
| print(f'{np.abs(ds.transform[0])=}') | ||
| print(f'{np.abs(ds.transform[4])=}') | ||
| ``` | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| El atributo `ds.transform` es un objeto que describe una [_transformación afín_](https://es.wikipedia.org/wiki/Transformaci%C3%B3n_af%C3%ADn) (representada anteriormente como una matriz $2\times3$). Observa que los valores absolutos de las entradas diagonales de la matriz `ds.transform` dan las dimensiones espaciales de los píxeles ($30\mathrm{m}\times30\mathrm{m}$ en este caso). | ||
|
|
||
| También podemos utilizar este objeto para convertir las coordenadas de los píxeles en las coordenadas espaciales correspondientes. | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| with rasterio.open(LOCAL_PATH) as ds: | ||
| print(f'{ds.transform * (0,0)=}') # top-left pixel | ||
| print(f'{ds.transform * (0,3660)=}') # bottom-left pixel | ||
| print(f'{ds.transform * (3660,0)=}') # top-right pixel | ||
| print(f'{ds.transform * (3660,3660)=}') # bottom-right pixel | ||
| ``` | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| El atributo `ds.bounds` muestra los límites de la región espacial (izquierda, abajo, derecha, arriba). | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| with rasterio.open(LOCAL_PATH) as ds: | ||
| print(f'coordinate bounds: {ds.bounds=}') | ||
| ``` | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| El método `ds.xy` también convierte coordenadas de índice entero en coordenadas continuas. Observa que `ds.xy` asigna enteros al centro de los píxeles. Los bucles siguientes imprimen la primera esquina superior izquierda de las coordenadas en coordenadas de píxel y, después, las coordenadas espaciales correspondientes. | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| with rasterio.open(LOCAL_PATH) as ds: | ||
| for k in range(3): | ||
| for l in range(4): | ||
| print(f'({k:2d},{l:2d})','\t', end='') | ||
| print() | ||
| print() | ||
| for k in range(3): | ||
| for l in range(4): | ||
| e,n = ds.xy(k,l) | ||
| print(f'({e},{n})','\t', end='') | ||
| print() | ||
| print() | ||
| ``` | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| `ds.index` hace lo contrario: dadas las coordenadas espaciales `(x,y)`, devuelve los índices enteros del píxel que contiene ese punto. | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| with rasterio.open(LOCAL_PATH) as ds: | ||
| print(ds.index(500000, 4700015)) | ||
| ``` | ||
|
|
||
| <!-- #region jupyter={"source_hidden": false} --> | ||
|
|
||
| Estas conversiones pueden ser complicadas, sobre todo porque las coordenadas de píxel corresponden a los centros de los píxeles y también porque la segunda coordenada espacial `y` _disminuye_ a medida que la segunda coordenada de píxel _aumenta_. Hacer un seguimiento de detalles tediosos como este es en parte la razón por la que resulta útil cargar desde `rioxarray`, es decir, que nosotros lo hagamos. Pero vale la pena saber que podemos reconstruir este mapeo si es necesario a partir de los metadatos en el archivo GeoTIFF (utilizaremos este hecho más adelante). | ||
|
|
||
| <!-- #endregion --> | ||
|
|
||
| ```{code-cell} python jupyter={"source_hidden": false} | ||
| with rasterio.open(LOCAL_PATH) as ds: | ||
| print(ds.bounds) | ||
| print(ds.transform * (0.5,0.5)) # Maps to centre of top left pixel | ||
| print(ds.xy(0,0)) # Same as above | ||
| print(ds.transform * (0,0)) # Maps to top left corner of top left pixel | ||
| print(ds.xy(-0.5,-0.5)) # Same as above | ||
| print(ds.transform[0], ds.transform[4]) | ||
| ``` | ||
|
|
||
| *** |
Oops, something went wrong.