| title | authors | authorLinks | affiliations | date | dataset | abstract |
|---|---|---|---|---|---|---|
Análise genômica do espalhamento da COVID-19. Relatório da situação até 05/03/2020. |
Trevor Bedford, Richard Neher, James Hadfield, Emma Hodcroft, Misja Ilcisin, Nicola Müller |
Fred Hutch, Seattle, USA and Biozentrum, Basel, Switzerland |
05 de Março de 2020 |
Este relatório usa dados genômicos sobre a COVID-19 publicamente compartilhados pela GISAID e Genbank, para estimar taxas e padrões de espalhamento de epidemia viral. Nós planejamos liberar relatórios atualizados assim que novos dados sejam produzidos e compartilhados. Esse site é otimizado para visualização em navegadores de computadores.
Tradução por: Glaucio Santos & Anderson Brito. |
## Resumo
Examinamos a diversidade genética dos 169</tag> genomas do SARS-CoV-2 compartilhados publicamente, a fim de caracterizar o espalhamento da doença COVID-19 em diferentes áreas, e inferir a data do ancestral comum dos vírus. Concluímos que:
* O vírus da COVID-19 foi introduzido na Itália ao menos duas vezes, seguido por espalhamento comunitário a partir disso ([saiba mais](https://nextstrain.org/narratives/ncov/sit-rep/pt/2020-03-05?n=7)).
* Isso inclui um grupo de sequências de 6 diferentes países, onde casos parecem ter sido exportados da Itália ([saiba mais](https://nextstrain.org/narratives/ncov/sit-rep/pt/2020-03-05?n=9)).
* Dados de sequências genéticas sustentam a hipótese de espalhamento sustentado do vírus da COVID-19 na Região Metropolitana de Seattle desde meados de Janeiro ([saiba mais](https://nextstrain.org/narratives/ncov/sit-rep/pt/2020-03-05?n=10)).
* Todos os casos sequenciados nesta análise provavelmente compartilham um ancestral comum de algum momento entre meados de Novembro e meados de Dezembro de 2019 ([saiba mais](https://nextstrain.org/narratives/ncov/sit-rep/pt/2020-03-05?n=11)).
- Resumo do surto de nCov na Wikipédia 30/01/2020
- Material disponibilizado pelo Centro de Controle e Prevenção de Doenças dos Estados Unidos (CDC) 29/01/2020
## Fontes sobre COVID-19
Abaixo preparamos alguns materiais (em Inglês) que valem a pena ler para se familiarizar com a COVID-19 e o vírus que o causa, conhecido como SARS-CoV-2.
Essa informação tornará mais fácil a interpretação dos dados apresentados aqui.
<div>
<a href="https://nextstrain.org/help/coronavirus/human-CoV"><img alt="microscopy image of coronaviruses" width="100" src="https://nextstrain.org/static/ncov_narrative-76cfd610d11ef708d213a3170de9519f.png"/> Sobre os Coronavírus </a>
<a href="https://nextstrain.org/help/coronavirus/SARS-CoV-2"><img alt="illustration of a coronavirus" width="100" src="http://data.nextstrain.org/img_nCoV-CDC.jpg"/> Sobre o Recente Surto de COVID-19 </a>
<a href="https://nextstrain.org/help/general/how-to-read-a-tree"><img alt="cartoon of a phylogenetic tree" width="100" src="http://data.nextstrain.org/img_toy_alignment_mini.png"/> Como interpretar filogenias</a>
</div>
## Narrativas Nextstrain
As páginas seguintes contém análises realizadas usando o [Nextstrain](https://nextstrain.org). Rolando a página no menu a esquerda, textos correspondentes aos dados genômicos visualizados na direita surgirão na tela.
Ter acesso a genomas completos de um novo vírus de RNA longo, e tão rapidamente, é uma conquista notável. Essas análises têm sido possíveis devido ao rápido compartilhamento público de dados genômicos e interpretações por cientistas de todo o mundo (veja a última página para conhecer os autores do sequenciamento).
-
"Não acredite nas teorias da conspiração que você ouve sobre coronavírus & HIV (em Inglês)" artigo 31/01/2020
-
"Teorias da conspiração infundadas afirmam que o novo coronavírus foi criado por bioengenharia (em Inglês)" artigo 07/02/2020
-
"Não, o coronavírus de Wuhan não foi geneticamente modificado para colocar pedaços de HIV nele (em Inglês)" artigo 02/02/2020
-
"Derrubando mitos envolvendo coronavírus (em Inglês)" AFP Checamos 19/02/2020
## Dúvidas & Equívocos Frequentes
### Dúvidas Frequentes
Sabemos que muitas pessoas têm dúvidas sobre a COVID-19. Criamos um guia para tentar responder algumas das perguntas mais frequentes:
<div>
<a href="https://nextstrain.org/help/coronavirus/FAQ"><img alt="picture of a question mark" width="100" src="http://data.nextstrain.org/img_question-mark.jpg"/> Dúvidas Frequentes sobre a COVID-19 (em Inglês) </a>
</div>
### Equívocos
Vários conceitos errados estão em circulação acerca das origens do novo coronavírus. Durante surtos como este, a disseminação de informações claramente incorretas pode levar a mais pânico, e fazer com que as pessoas não confiem em cientistas e governos, o que significa que estarão menos propensos a seguir orientações e tomar as devidas precauções.
Num esforço conjunto para tentar explicar por que essas visões estão incorretas, cientistas abordaram essas teorias nas páginas abaixo (em Inglês):
<div>
<a href="http://virological.org/t/ncovs-relationship-to-bat-coronaviruses-recombination-signals-no-snakes-no-evidence-the-2019-ncov-lineage-is-recombinant/331"><img alt="picture of a snake" width="100" src="http://data.nextstrain.org/img_snake-freeToUse.jpg"/> SARS-CoV-2 tem origem em "cobras"? (texto técnico) </a>
<a href="https://twitter.com/trvrb/status/1223666856923291648"><img alt="illustration of HIV" width="100" src="http://data.nextstrain.org/img_HIV-wiki.jpg"/> "A suposta engenharia do HIV" (Tópico no Twitter)</a>
</div>
Aqui apresentamos uma filogenia de 169 cepas de SARS-CoV-2 (o vírus que causa a COVID-19) que foram compartilhadas publicamente. Informações sobre como a análise foi realizada estão disponíveis em nosso repositório no GitHub.
As cores representam a região ou estado onde foram coletadas as amostras, com o eixo X representando a data da amostra. O eixo Y mostra como as sequências estão conectadas e não possui nenhuma unidade de medida.
As datas das amostras são úteis, mas nem sempre mostram exatamente como duas sequências estão relacionadas geneticamente - duas sequências idênticas podem ter datas de amostragem diferentes e, portanto, podem parecer distantes.
Podemos mudar a forma de visualização para que o eixo x agora mostre 'divergência'.
Perceba quantas sequências que pareciam diferentes antes, agora estão alinhadas verticalmente. Você pode rolar para cima e para baixo entre a página anterior e esta, para ver como a árvore muda.
A divergência é medida conforme o número de mudanças (mutações) no genoma. Muitas sequências têm 0 mutações, o que significa que são todas idênticas até a raíz (base) da árvore. Outros vírus têm entre uma e onze mutações.
Sequenciar o genoma de um novo vírus de RNA longo durante um surto é desafiante. Algumas das diferenças observadas nessas sequências podem ser resultado de erros de sequenciamento, e não mutações reais. Inserções, deleções, e diferenças nas extremidades do genoma têm grandes chances de serem erros, por isso as ignoramos visando cumprir os propósitos dessa análise.
Algumas vezes mostraremos a árvore em modo "cronológico" (tempo) e, às vezes no modo "divergência", dependendo do que estamos tentando destacar.
Atualmente temos sequências de amostras colhidas em cinco continentes diferentes. Enquanto os primeiros casos estavam todos diretamente ligados aos casos em Wuhan (associados com o surto do mercado de frutos do mar), agora observamos vários casos diferentes que mostram evidências de transmissão comunitária (ou seja, no próprio local de detecção do vírus) ou importados de outros lugares fora da China.
Geralmente, quando as introduções via animais reservatórios são múltiplas, é possível observar diversidade genética significativa (como tem sido com a febre de Lassa, o Ebola, a MERS-CoV e a gripe aviária). O fato de podermos observar um agrupamento tão forte em infecções humanas pode indicar que o surto descende de uma única introdução zoonótica (vinda de um animal) para a população humana, seguida do espalhamento epidêmico de pessoa para pessoa.
Atualmente, temos três sequências da Itália, duas das quais são da área de Roma e uma da Lombardia (norte do país).
Essas três sequências compartilham um ancestral comum no início da epidemia (próximo à base da árvore, à esquerda), o que sugere fortemente que houve pelo menos duas introduções com transmissão comunitária na Itália.
Dr. Nuno Faria e colaboradores publicaram uma excelente análise disponível aqui (em Inglês), explicando como as sequências brasileiras e outras sequências globais mostram que "o surto no norte da Itália foi provavelmente resultado de várias introduções na região, e não de uma única fonte".
As duas sequências de Roma (29 de Janeiro de 2020) estão diretamente conectadas entre si e ambas têm um histórico de viagens para a China.
Mudamos agora para a visualização de "divergências" para exibir como as duas sequências italianas são idênticas, enquanto outras sequências próximas (da Inglaterra, Brasil, Suíça, EUA e China) são na verdade separadas das sequências italianas por 2 a 4 mutações.
Contudo, a sequência brasileira (SPBR-02) tem um histórico de viagem para Milão (Lombardia), e acredita-se que a sequência suíça desse grupo também tenha viajado recentemente para a Itália. Não sabemos nada sobre o histórico de viagens da amostra dos Estados Unidos. O caso da Inglaterra é uma importação direta da China.
A amostra da Inglaterra (oriunda da China) fica entre as amostras italianas mais antigas (de Roma) e os casos com viagens confirmadas para a Itália (Suíça e Brasil) ou viagens possíveis (EUA). Isso significa que não devemos supor que as sequências italianas mais antigas e as as sequências mais recentes associadas às italianas estão diretamente ligadas. As amostras mais recentes podem ser resultado de outra introdução independente na Itália.
A sequência da Lombardia (Italy/CDG1/2020) se agrupa junto das sequências de pacientes com histórico de viagens para a Itália, os quais foram provavelmente infectados naquele país. São elas: as sequências do México, Alemanha, Brasil & Finlândia.
A sequência alemã "BavPat1" faz parte de uma introdução mais antiga proveniente da China. Sua semelhança com a outra sequência no grupo (elas são separadas por apenas uma mutação), pode indicar uma transmissão não detectada ("silenciosa") na Europa, decorrente daquele antigo grupo de sequências alemãs.
Esse grupo de sequências pode também ser resultado de duas introduções separadas na Europa - por exemplo, uma sequência ainda não amostrada de outro lugar poderia se posicionar entre "BavPat1" e o resto do grupo mostrado ao lado, levantando uma hipótese alternativa. Nesse sentido, no momento não podemos dizer com certeza qual cenário está correto.
No momento há vários casos de COVID-19 relatados na área metropolitana de Seattle e nos Estados Unidos em geral. Os casos recém-descobertos e sequenciados são geneticamente relacionados a um caso amostrado no meio de Janeiro na mesma região.
Há duas possíveis explicações para isso. O vírus pode ter sido introduzido ao menos duas vezes na área da grande Seattle de uma fonte comum na China. No entanto, outra explicação é que o vírus circulava sem ser detectado na área por algum tempo.
Trevor Bedford (co-fundador da Nextstrain) escreveu um ótimo artigo sobre essas possibilidades, o qual você pode ler aqui (em Inglês).
As sequências recentes de Washington nos dizem outra coisa: essas sequências da região de Seattle estão proximamente agrupadas. Isso sugere fortemente uma transmissão comunitária, e que o vírus SARS-CoV-2 já estava circulando na área há algum tempo.
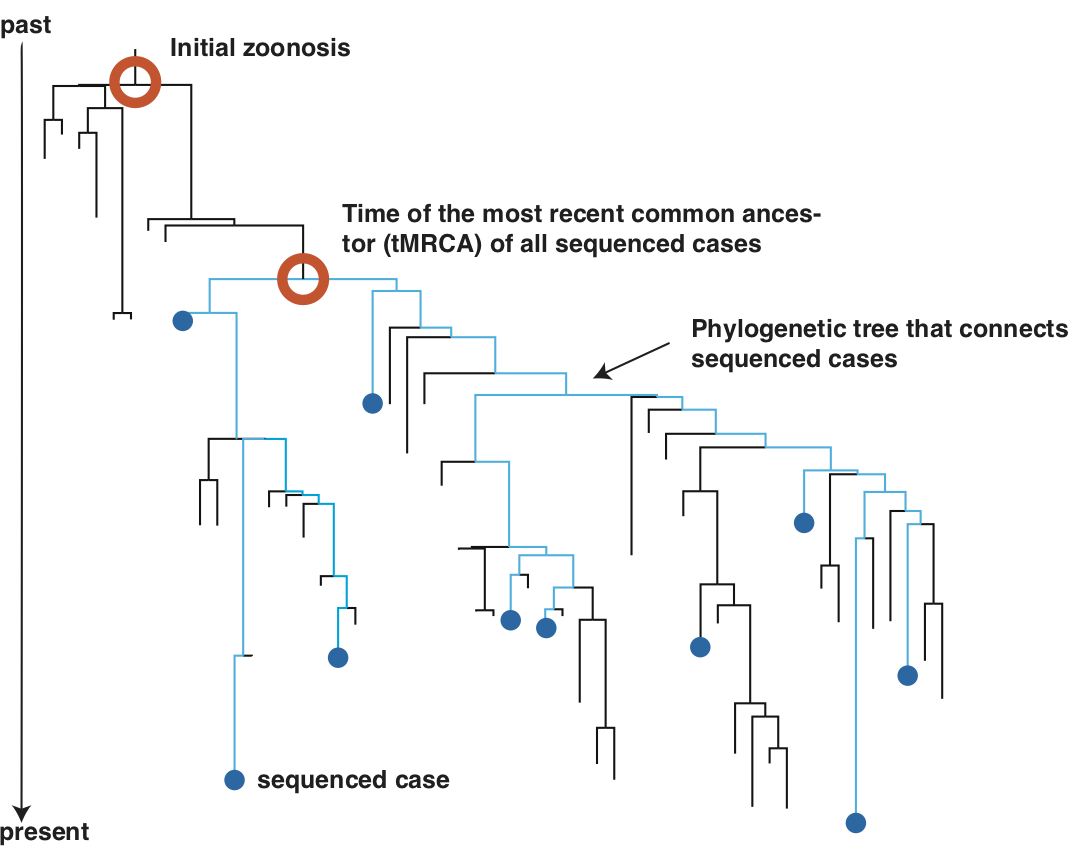
O tMRCA (sigla em Inglês para 'tempo até o ancestral comum mais recente') de um conjunto de casos conhecidos indica quando esses casos sequenciados compartilharam pela última vez um ancestral comum. Esse momento pode ser tão cedo quanto no exato momento em que um vírus entrou na população humana, mas também pode ser substancialmente mais tarde, como mostra a figura abaixo.
## Provável data do ancestral comum dos vírus causadores do surto
Inúmeros grupos de pesquisa e pessoas têm estimado a data do ancestral comum mais recente - confira [esta postagem de A. Rambaut (em Inglês)](http://virological.org/t/phylodynamic-analysis-of-sars-cov-2-update-2020-03-06/420) ou [esta postagem de T. Stadler (em Inglês)](http://virological.org/t/evolutionary-epidemiological-analysis-of-93-genomes).
O ancestral comum de todas as sequências provavelmente existiu entre meados de Novembro e meados de Dezembro. Isso seria consistente com todos os casos atualmente sequenciados, descendentes do [conjunto inicial de casos no mercado de frutos do mar de Wuhan](http://virological.org/t/phylodynamic-analysis-of-sars-cov-2-update-2020-03-06/420).
<div>
<img alt="estimate of the tMRCA using Bayesian phylogenetics" width="500" src="https://raw.githubusercontent.com/nicfel/nCov-Nicola/master/figures/beast_coal-tmrca_2020303.png"/>
</div>
Gostaríamos de reconhecer o trabalho incrível e oportuno realizado por todos os cientistas envolvidos nesse surto, sobretudo os que estão trabalhando na China. Somente com o compartilhamento rápido de dados genômicos e de metadados que tais análises foram possíveis.
Também agradecemos a GISAID por fornecer a plataforma pela qual esses dados podem ser inseridos e compartilhados.
Somos gratos pelos dados coletados pelos seguintes laboratórios:
* Centre for Infectious Diseases and Microbiology Laboratory Services
* Pathology Queensland
* Monash Medical Centre
* National Institute for Viral Disease Control and Prevention, China CDC
* KU Leuven, Clinical and Epidemiological Virology
* Hospital Israelita Albert Einstein
* Virology Unit, Institut Pasteur du Cambodge.
* BCCDC Public Health Laboratory
* Yongchuan District Center for Disease Control and Prevention
* Zhongxian Center for Disease Control and Prevention
* Respiratory Virus Unit, Microbiology Services Colindale, Public Health England
* Lapland Central Hospital
* HUS Diagnostiikkakeskus, Hallinto
* Guangdong Provincial Center for Diseases Control and Prevention; Guangdong Provincial Public Health
* Department of Infectious and Tropical Diseases, Bichat Claude Bernard Hospital, Paris
* Sorbonne Universite, Inserm et Assistance Publique-Hopitaux de Paris (Pitie Salpetriere)
* CNR Virus des Infections Respiratoires - France SUD
* Fujian Center for Disease Control and Prevention
* State Health Office Baden-Wuerttemberg
* Charite Universitatsmedizin Berlin, Institute of Virology; Institut fur Mikrobiologie der Bundeswehr, Munich
* Guangdong Provincial Center for Diseases Control and Prevention; Guangdong Provinical Public Health
* Guangdong Provincial Center for Diseases Control and Prevention;Guangdong Provincial Institute of Public Health
* Hangzhou Center for Disease and Control Microbiology Lab
* Hangzhou Center for Disease Control and Prevention
* Second Hospital of Anhui Medical University
* Hong Kong Department of Health
* Department of Infectious Diseases, Istituto Superiore di Sanita, Roma , Italy
* INMI Lazzaro Spallanzani IRCCS
* Department of Infectious Diseases, Istituto Superiore di Sanita, Rome, Italy
* Department of Virology III, National Institute of Infectious Diseases
* Dept. of Virology III, National Institute of Infectious Diseases
* Dept. of Pathology, National Institute of Infectious Diseases
* NHC Key laboratory of Enteric Pathogenic Microbiology, Institute of Pathogenic Microbiology
* Jingzhou Center for Disease Control and Prevention
* Division of Viral Diseases, Center for Laboratory Control of Infectious Diseases, Korea Centers for Diseases Control and Prevention
* Instituto Nacional de Enfermedades Respiratorias
* National Influenza Centre, National Public Health Laboratory, Kathmandu, Nepal
* Bamrasnaradura Hospital
* The University of Hong Kong - Shenzhen Hospital
* Shenzhen Third People's Hospital
* Shenzhen Key Laboratory of Pathogen and Immunity, National Clinical Research Center for Infectious Disease, Shenzhen Third People's Hospital
* Singapore General Hospital
* National Public Health Laboratory, National Centre for Infectious Diseases
* National Public Health Laboratory
* National Centre for Infectious Diseases
* Singapore General Hospital, Molecular Laboratory, Division of Pathology
* Korea Centers for Disease Control & Prevention (KCDC) Center for Laboratory Control of Infectious Diseases Division of Viral Diseases
* Serology, Virology and OTDS Laboratories (SAViD), NSW Health Pathology Randwick
* Centers for Disease Control, R.O.C. (Taiwan)
* Taiwan Centers for Disease Control
* Laboratory Medicine
* Department of Laboratory Medicine, National Taiwan University Hospital
* Tianmen Center for Disease Control and Prevention
* Arizona Department of Health Services
* California Department of Public Health
* California Department of Health
* IL Department of Public Health Chicago Laboratory
* Massachusetts Department of Public Health
* Texas Department of State Health Services
* WA State Department of Health
* Washington State Department of Health
* Providence Regional Medical Center
* Wisconsin Department of Health Services
* National Influenza Center - National Institute of Hygiene and Epidemiology (NIHE)
* Wuhan Jinyintan Hospital
* The Central Hospital Of Wuhan
* Union Hospital of Tongji Medical College, Huazhong University of Science and Technology
* CR & Wisco General Hospital
* Wuhan Lung Hospital
* Institute of Pathogen Biology, Chinese Academy of Medical Sciences & Peking Union Medical College
* Institute of Viral Disease Control and Prevention, China CDC
* General Hospital of Central Theater Command of People's Liberation Army of China
* Wuhan Fourth Hospital
* Zhejiang Provincial Center for Disease Control and Prevention
* Wuhan Institute of Virology, Chinese Academy of Sciences
* Shandong First Medical University & Shandong Academy of Medical Sciences
* South China Agricultural University
* Beijing Institute of Microbiology and Epidemiology
Esses dados foram compartilhados através da GISAID. Agradecemos as suas contribuições com profunda gratidão.
A direita, especificamos quais sequências foram compartilhadas por quais laboratórios.
Os genomas de SARS-CoV-2 foram generosamente cedidos por cientistas desses laboratórios:
* NSW Health Pathology - Institute of Clinical Pathology and Medical Research; Westmead Hospital; University of Sydney
* Australia/NSW01/2020
* Australia/NSW05/2020
* Sydney/2/2020
* Public Health Virology Laboratory
* Australia/QLD01/2020
* Australia/QLD02/2020
* Australia/QLD03/2020
* Australia/QLD04/2020
* Collaboration between the University of Melbourne at The Peter Doherty Institute for Infection and Immunity, and the Victorian Infectious Disease Reference Laboratory
* Australia/VIC01/2020
* National Institute for Viral Disease Control & Prevention, CCDC
* Beijing/IVDC-BJ-005/2020
* Chongqing/IVDC-CQ-001/2020
* Jiangsu/IVDC-JS-001/2020
* Jiangxi/IVDC-JX-002/2020
* Shandong/IVDC-SD-001/2020
* Shanghai/IVDC-SH-001/2020
* Sichuan/IVDC-SC-001/2020
* Yunnan/IVDC-YN-003/2020
* KU Leuven, Clinical and Epidemiological Virology
* Belgium/GHB-03021/2020
* Instituto Adolfo Lutz Interdisciplinary Procedures Center Strategic Laboratory
* Brazil/SPBR-01/2020
* Brazil/SPBR-02/2020
* Virology Unit, Institut Pasteur du Cambodge (Sequencing done by: Jessica E Manning/Jennifer A Bohl at Malaria and Vector Research Research Laboratory, National Institute of Allergy and Infectious Diseases and Vida Ahyong from Chan-Zuckerberg Biohub)
* Cambodia/0012/2020
* BCCDC Public Health Laboratory
* Canada/BC_37_0-2/2020
* Technology Centre, Guangzhou Customs
* China/IQTC01/2020
* China/IQTC02/2020
* Key Laboratory of Human Diseases, Comparative Medicine, Institute of Laboratory Animal Science
* China/WH-09/2020
* State Key Laboratory of Virology, Wuhan University
* China/WHU01/2020
* China/WHU02/2020
* Chongqing Municipal Center for Disease Control and Prevention
* Chongqing/YC01/2020
* Chongqing/ZX01/2020
* Respiratory Virus Unit, Microbiology Services Colindale, Public Health England
* England/01/2020
* England/02/2020
* England/09c/2020
* Department of Virology, University of Helsinki and Helsinki University Hospital, Helsinki, Finland
* Finland/1/2020
* Department of Virology Faculty of Medicine, Medicum University of Helsinki
* Finland/FIN-25/2020
* Guangdong Provincial Center for Diseases Control and Prevention
* Foshan/20SF207/2020
* Foshan/20SF210/2020
* Foshan/20SF211/2020
* Guangdong/20SF201/2020
* Guangzhou/20SF206/2020
* National Reference Center for Viruses of Respiratory Infections, Institut Pasteur, Paris
* France/IDF0372-isl/2020
* France/IDF0372/2020
* France/IDF0373/2020
* France/IDF0515-isl/2020
* France/IDF0515/2020
* France/IDF0626/2020
* Laboratoire Virpath, CIRI U111, UCBL1, INSERM, CNRS, ENS Lyon
* France/IDF0386-islP1/2020
* France/IDF0386-islP3/2020
* France/IDF0571/2020
* CNR Virus des Infections Respiratoires - France SUD
* France/RA739/2020
* Fujian Center for Disease Control and Prevention
* Fujian/13/2020
* Fujian/8/2020
* Charite Universitatsmedizin Berlin, Institute of Virology
* Germany/Baden-Wuerttemberg-1/2020
* Germany/BavPat1/2020
* Department of Microbiology, Guangdong Provincial Center for Diseases Control and Prevention
* Guangdong/20SF012/2020
* Guangdong/20SF013/2020
* Guangdong/20SF014/2020
* Guangdong/20SF025/2020
* Guangdong/20SF028/2020
* Guangdong/20SF040/2020
* Guangdong Provincial Center for Disease Control and Prevention
* Guangdong/20SF174/2020
* Hangzhou Center for Disease and Control Microbiology Lab
* Hangzhou/HZ-1/2020
* Hangzhou Center for Disease Control and Prevention
* Hangzhou/HZCDC0001/2020
* Second Hospital of Anhui Medical University
* Hefei/2/2020
* National Institute for Viral Disease Control & Prevention, China CDC
* Henan/IVDC-HeN-002/2020
* School of Public Health, The University of Hon g Kong
* HongKong/VB20026565/2020
* HongKong/VM20001061/2020
* The University of Hong Kong
* HongKong/VM20001988/2020
* Nepal/61/2020
* Virology Laboratory, Scientific Department, Army Medical Center
* Italy/CDG1/2020
* Italy/SPL1/2020
* Laboratory of Virology, INMI Lazzaro Spallanzani IRCCS
* Italy/INMI1-cs/2020
* Italy/INMI1-isl/2020
* Pathogen Genomics Center, National Institute of Infectious Diseases
* Japan/AI/I-004/2020
* Japan/KY-V-029/2020
* Japan/NA-20-05-1/2020
* Japan/OS-20-07-1/2020
* Japan/TY-WK-012/2020
* Japan/TY-WK-501/2020
* Japan/TY-WK-521/2020
* Takayuki Hishiki Kanagawa Prefectural Institute of Public Health, Department of Microbiology
* Japan/Hu_DP_Kng_19-020/2020
* Japan/Hu_DP_Kng_19-027/2020
* Jiangsu Provincial Center for Disease Control & Prevention
* Jiangsu/JS01/2020
* Jiangsu/JS02/2020
* Jiangsu/JS03/2020
* Hubei Provincial Center for Disease Control and Prevention
* Jingzhou/HBCDC-HB-01/2020
* Tianmen/HBCDC-HB-07/2020
* Wuhan/HBCDC-HB-01/2019
* Wuhan/HBCDC-HB-02/2019
* Wuhan/HBCDC-HB-02/2020
* Wuhan/HBCDC-HB-03/2019
* Wuhan/HBCDC-HB-03/2020
* Wuhan/HBCDC-HB-04/2019
* Wuhan/HBCDC-HB-04/2020
* Wuhan/HBCDC-HB-05/2020
* Wuhan/HBCDC-HB-06/2020
* Division of Viral Diseases, Center for Laboratory Control of Infectious Diseases, Korea Centers for Diseases Control and Prevention
* Korea/KCDC05/2020
* Korea/KCDC06/2020
* Korea/KCDC07/2020
* Korea/KCDC12/2020
* Korea/KCDC24/2020
* Instituto de Diagnostico y Referencia Epidemiologicos (INDRE)
* Mexico/CDMX/InDRE_01/2020
* 1. Department of Medical Sciences, Ministry of Public Health, Thailand 2. Thai Red Cross Emerging Infectious Diseases - Health Science Centre 3. Department of Disease Control, Ministry of Public Health, Thailand
* Nonthaburi/61/2020
* Nonthaburi/74/2020
* Li Ka Shing Faculty of Medicine, The University of Hong Kong
* Shenzhen/HKU-SZ-002/2020
* Shenzhen/HKU-SZ-005/2020
* Shenzhen Key Laboratory of Pathogen and Immunity, National Clinical Research Center for Infectious Disease, Shenzhen Third People's Hospital
* Shenzhen/SZTH-001/2020
* Shenzhen/SZTH-002/2020
* Shenzhen/SZTH-003/2020
* Shenzhen/SZTH-004/2020
* National Public Health Laboratory
* Singapore/1/2020
* Singapore/11/2020
* National Centre for Infectious Diseases, National Centre for Infectious Diseases
* Singapore/10/2020
* Programme in Emerging Infectious Diseases, Duke-NUS Medical School
* Singapore/2/2020
* Singapore/3/2020
* Singapore/4/2020
* Singapore/5/2020
* Singapore/6/2020
* National Public Health Laboratory, National Centre for Infectious Diseases
* Singapore/7/2020
* Singapore/8/2020
* Singapore/9/2020
* Korea Centers for Disease Control & Prevention (KCDC) Center for Laboratory Control of Infectious Diseases Division of Viral Diseases
* SouthKorea/KCDC03/2020
* Department of Clinical Diagnostics
* SouthKorea/SNU01/2020
* Unit for Laboratory Development and Technology Transfer, Public Health Agency of Sweden
* Sweden/01/2020
* NSW Health Pathology - Institute of Clinical Pathology and Medical Research; Centre for Infectious Diseases and Microbiology Laboratory Services; Westmead Hospital; University of Sydney
* Sydney/3/2020
* Centers for Disease Control, R.O.C. (Taiwan)
* Taiwan/2/2020
* Taiwan Centers for Disease Control
* Taiwan/3/2020
* Taiwan/4/2020
* Department of Laboratory Medicine, Lin-Kou Chang Gung Memorial Hospital, Taoyuan, Taiwan.
* Taiwan/CGMH-CGU-01/2020
* Microbial Genomics Core Lab, National Taiwan University Centers of Genomic and Precision Medicine
* Taiwan/NTU01/2020
* Taiwan/NTU02/2020
* Pathogen Discovery, Respiratory Viruses Branch, Division of Viral Diseases, Centers for Disease Control and Prevention
* USA/AZ1/2020
* USA/CA1/2020
* USA/CA2/2020
* USA/CA3/2020
* USA/CA4/2020
* USA/CA5/2020
* USA/CA6/2020
* USA/CA7/2020
* USA/CA8/2020
* USA/CA9/2020
* USA/IL1/2020
* USA/IL2/2020
* USA/MA1/2020
* USA/TX1/2020
* USA/WA1-A12/2020
* USA/WA1-F6/2020
* USA/WI1/2020
* Division of Viral Diseases, Centers for Disease Control and Prevention
* USA/WA1/2020
* Seattle Flu Study
* USA/WA2/2020
* National Influenza Center - National Institute of Hygiene and Epidemiology (NIHE)
* Vietnam/VR03-38142/2020
* National Institute for Communicable Disease Control and Prevention (ICDC) Chinese Center for Disease Control and Prevention (China CDC)
* Wuhan-Hu-1/2019
* Institute of Pathogen Biology, Chinese Academy of Medical Sciences & Peking Union Medical College
* Wuhan/IPBCAMS-WH-01/2019
* Wuhan/IPBCAMS-WH-02/2019
* Wuhan/IPBCAMS-WH-03/2019
* Wuhan/IPBCAMS-WH-04/2019
* Wuhan/IPBCAMS-WH-05/2020
* National Institute for Viral Disease Control and Prevention, China CDC
* Wuhan/IVDC-HB-01/2019
* Wuhan/IVDC-HB-04/2020
* Wuhan/IVDC-HB-05/2019
* Institute of Viral Disease Control and Prevention, China CDC
* Wuhan/IVDC-HB-envF13-20/2020
* Wuhan/IVDC-HB-envF13-21/2020
* Wuhan/IVDC-HB-envF13/2020
* Wuhan/IVDC-HB-envF54/2020
* BGI & Institute of Microbiology, Chinese Academy of Sciences & Shandong First Medical University & Shandong Academy of Medical Sciences & General Hospital of Central Theater Command of People's Liberation Army of China
* Wuhan/WH01/2019
* Wuhan/WH02/2019
* Wuhan/WH03/2020
* Wuhan/WH04/2020
* Beijing Genomics Institute (BGI)
* Wuhan/WH05/2020
* Wuhan Institute of Virology, Chinese Academy of Sciences
* Wuhan/WIV02/2019
* Wuhan/WIV04/2019
* Wuhan/WIV05/2019
* Wuhan/WIV06/2019
* Wuhan/WIV07/2019
* Department of Microbiology, Zhejiang Provincial Center for Disease Control and Prevention
* Zhejiang/WZ-01/2020
* Zhejiang/WZ-02/2020