| title | authors | authorLinks | affiliations | date | dataset | abstract |
|---|---|---|---|---|---|---|
新型コロナウイルス拡散の遺伝的解析と状況報告 2020-03-05. |
Trevor Bedford, Richard Neher, James Hadfield, Emma Hodcroft, Misja Ilcisin, Nicola Müller, Takeshi Sato, Fengjun Zhang, Tomoyuki Nezu |
Fred Hutch, Seattle, USA and Biozentrum, Basel, Switzerland |
2020 March 05 |
このレポートでは、GISAID および Genbank から公開されている COVID-19 遺伝子情報を使用して、ウイルスの流行の広がりの割合とパターンを推定します。 新しいデータが作成および共有次第、このレポートを更新する予定です。 この Web サイトは、デスクトップブラウザーでの表示用に最適化されています。 |
## 概要
公開共有された169系統の COVID-19 遺伝子情報を使用して遺伝的多様性を調べることで、さまざまな地域での COVID-19 の広がりを特徴付け、共通の祖先の日付を推測しました。
私たちが発見したこととしては、
* COVID-19 はイタリアに少なくとも2度持ち込まれ、その後コミュニティで拡散した ([ページを見る](https://nextstrain.org/narratives/ncov/sit-rep/ja/2020-03-05?n=7))
* これには、イタリアから症例が輸出されたと思われる6か国の遺伝子情報のクラスターが含まれます ([これを見る](https://nextstrain.org/narratives/ncov/sit-rep/ja/2020-03-05?n=9))
* 遺伝子配列データは、1月中旬以降にシアトル地域で COVID-19 が検出されずに持続的に拡散するという仮説を裏付けています ([これを見る](https://nextstrain.org/narratives/ncov/sit-rep/ja/2020-03-05?n=10))
* この解析で用いられたものも含み、遺伝子解析が行われたすべての症例は、2019年11月中頃から12月中頃までの期間に共通祖先を持つ可能性があります ([これを見る](https://nextstrain.org/narratives/ncov/sit-rep/ja/2020-03-05?n=11))
## COVID-19 に関する資料
以下に、COVID-19 とその症例を引き起こすウイルスである SARS-CoV-2 を理解するための参考文献を用意しました。この情報により、このレポートで提示するデータの解釈が容易になります。
<div>
<a href="https://nextstrain.org/help/coronavirus/human-CoV"><img alt="microscopy image of coronaviruses" width="100" src="https://nextstrain.org/static/ncov_narrative-76cfd610d11ef708d213a3170de9519f.png"/> コロナウイルスの背景 </a>
<a href="https://nextstrain.org/help/coronavirus/SARS-CoV-2"><img alt="illustration of a coronavirus" width="100" src="http://data.nextstrain.org/img_nCoV-CDC.jpg"/> 最近の COVID-19 流行の背景 </a>
<a href="https://nextstrain.org/help/general/how-to-read-a-tree"><img alt="cartoon of a phylogenetic tree" width="100" src="http://data.nextstrain.org/img_toy_alignment_mini.png"/> 系統発生の読み方 </a>
</div>
## Nextstrain からの状況報告
このあとのページが[Nextstrain](https://nextstrain.org) を使用して行われた解析です。左側のサイドバーをスクロールすると項目ごとのテキストが表示され、右側に対応するゲノム情報が表示されます。
新規で大型な RNA ウイルスの完全なゲノム情報を早い段階でこのような形にするのは驚くべき成果です。これらの解析は、世界中の科学者によるゲノム情報の迅速かつオープンな共有、解釈によって可能になりました(遺伝子配列の提供者については最後のスライドを参照してください)
-
"Baseless Conspiracy Theories Claim New Coronavirus Was Bioengineered" 記事 2020-02-07
-
"No, The Wuhan Coronavirus Was Not Genetically Engineered To Put Pieces Of HIV In It" 記事 2020-02-02
-
"Busting coronavirus myths" AFP ファクトチェック 2020-02-19
## よくある質問と誤解
### よくある質問
多くの方々が COVID-19 について疑問をお持ちでしょう。よくある質問について[こちら](https://nextstrain.org/help/coronavirus/FAQ)にガイドを用意しました。
<div>
<a href="https://nextstrain.org/help/coronavirus/FAQ"><img alt="picture of a question mark" width="100" src="http://data.nextstrain.org/img_question-mark.jpg"/> COVID-19 FAQ </a>
</div>
### 誤解
新規コロナウイルスの起源について、多くの誤解が広まっています。このような大きな感染症流行において、不適切であるとわかる情報の拡散はより大きなパニックを引き起こし、人々が科学者と政府に対して不信を抱かせることに繋がります。つまり、助言に従い、適切な予防措置を取る可能性が低くなります。
次のような見方がなぜ適切でないかを説明しようと、科学者は以下のページでこれらの理論に取り組んでいます。
<div>
<a href="http://virological.org/t/ncovs-relationship-to-bat-coronaviruses-recombination-signals-no-snakes-no-evidence-the-2019-ncov-lineage-is-recombinant/331"><img alt="picture of a snake" width="100" src="http://data.nextstrain.org/img_snake-freeToUse.jpg"/> SARS-CoV-2 の起源 "蛇" (専門的) </a>
<a href="https://twitter.com/trvrb/status/1223666856923291648"><img alt="illustration of HIV" width="100" src="http://data.nextstrain.org/img_HIV-wiki.jpg"/> "HIV エンジニアリング"という考え(Twitter)</a>
</div>
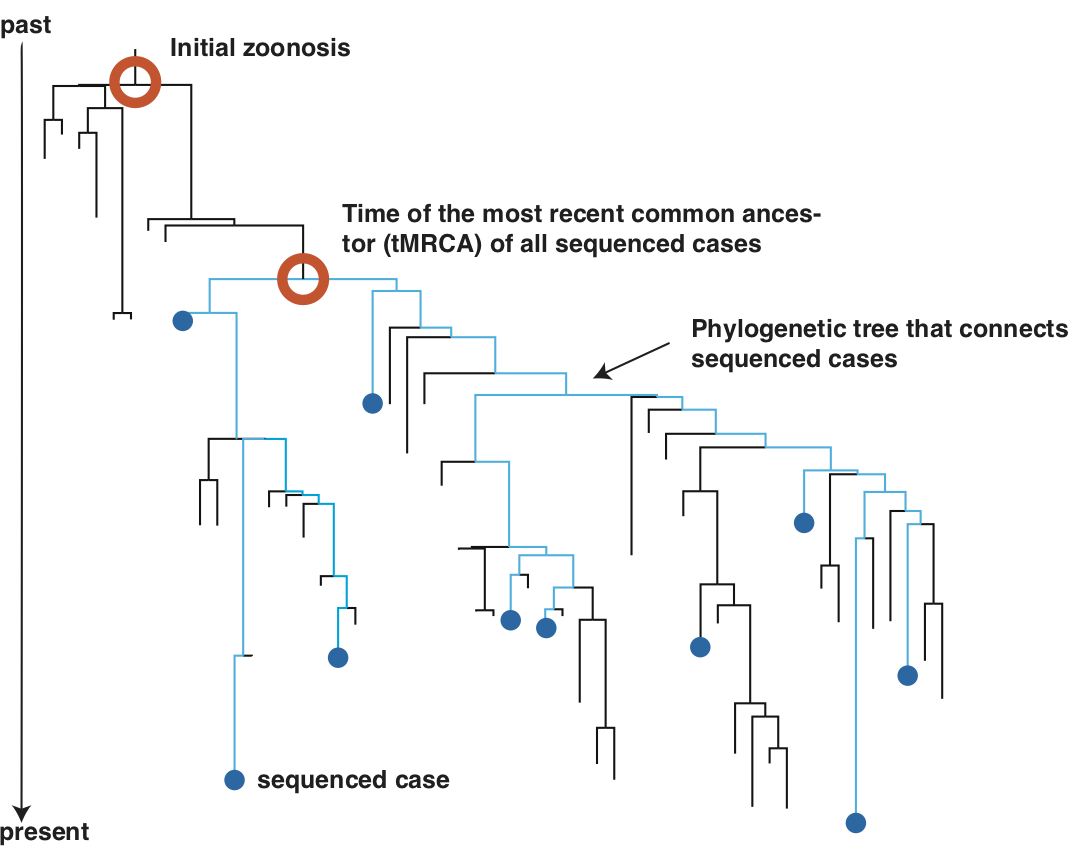
ここでは、公に共有されている SARS-CoV-2(COVID-19を引き起こすウイルス)の169系統の系統発生を示します。 解析の実行方法に関する情報は、このGitHubリポジトリで閲覧可能です。
色は、国内の地域または米国の州別を表し、x軸はサンプルの日付を、y軸は遺伝子情報の関係性を示します。測定単位はありません。
サンプル取得日は有益ですが、2つの系統が遺伝的にどのように関連するかを常に正確に示すわけではありません。2つの遺伝子配列はサンプル取得日が違ったとしても同一ですが、この表示方法では離れて見えてしまっています。
サンプル取得日の代わりに x軸に"多様性"を表すように表示を変えました
さきほどの表示とは違った見え方をする系統の多さに注意してください、この画面では垂直方向に一列に並んでいます。前のスライドとこのスライドの間をスクロールで行き来して、系統樹の変化を見ることができます。
多様性は遺伝子情報の変化 (突然変異) の数で計測しています。系統によっては、突然変異がゼロの場合があります。つまり、それらはすべてツリーのルート(中心)と同一です。 他のウイルスには1から11個の変異があります。
今まさに流行が起きている状況下で、新規の大規模な RNA ウイルスの遺伝子情報に対して系統解析を行う事は困難です。これらの系統解析で観察される違いの一部は、実際の突然変異ではなく、遺伝子配列解析のエラーである可能性があります。 遺伝子配列の両端での挿入、削除、および変異はエラーである可能性が高いため、この解析の目的に従い除去しました。
私たちが何を強調しようとしているかにより、系統樹を"時間" ビューで表示する場合もありますし、 "多様性" ビューで表示する場合もあります。
私たちは現在5つの異なる大陸で採取されたサンプル由来の遺伝子配列を所有しています。早期の症例は、海産食品市場での感染流行と関連した、武漢での症例と直接的に結びついていましたが、現在では、コミュニティでの拡散を示すものや、中国国外の感染源から持ち込まれた、様々な症例が見られます。
一般的に、動物を宿主とし何度も持ち込まれるウイルスは大きな多様性を示します (ラサ、エボラ、MERSや鳥インフルエンザで当てはまります) このような強いヒト感染のクラスター化の観察は、動物からヒトへのウイルスの持ち込みが一度きりの感染流行で、ヒトからヒトへの疫学的拡散が続いたと説明することができます。
現時点でイタリアからの遺伝子情報が3つあり、そのうち2つはローマ (Rome) 地域からのもので、1つは北イタリアのロンバルディア (Lombardy) からのものです。
これらの3つの遺伝子配列は感染流行の初期に共通の祖先を持ちます (左側、系統樹の元の近く) このことはイタリアには少なくとも 2 度ウイルスが持ち込まれたことを強く示唆します。
Nuno Faria 博士らは ここでブラジルや世界の他の遺伝子配列が "北イタリアの感染流行が一つの感染源由来ではなく、複数回当該の地域に持ち込まれた結果である可能性が高い" という優れた解析を提示しています。
ローマ由来の2つの遺伝子情報 (2020/01/29) は互いに直接的に結びついていて、どちらの患者の方も中国への渡航歴がありました。
"多様性" ビューに切り替えて、イタリアの2つの遺伝子情報が同一であることを示しました。他の近くの遺伝子情報 (イングランド、ブラジル、スイス、アメリカ、中国由来の) は実際そのイタリアの遺伝子情報とは2から4つの変異の差があることを示しました。
しかしながら、ブラジルの遺伝子配列 (SPBR-02) はロンバルディア (Lombardy) 州のミラノ (Milan) への渡航歴があり、このクラスタ内でのスイスの遺伝子配列もイタリアへの渡航歴があったと考えられています。アメリカのサンプルの渡航歴については不明です。England/09c の1件は中国からの直接的な持ち込みです。
(中国からの) イングランドのサンプルは早期のローマからのイタリアのサンプルと、イタリアへの渡航が既知のサンプル (スイス、ブラジル) またはその可能性がある (アメリカ) のサンプルの間にあります。これは、これらの早期のイタリアの遺伝子配列が、最近のイタリアに関係する遺伝子配列と直接結びついていると想定すべきではないということを意味します。最近のサンプルはイタリアに独立して別に持ち込まれた持ち込まれた可能性があります。
ロンバルディア (Lombardy) (Italy / CDG1 / 2020) 由来の遺伝子配列はイタリアへの渡航歴があり、そこで感染した可能性が高い既知の遺伝子配列とクラスタを形成しています。メキシコ、ドイツ、ブラジルそしてフィンランドからの遺伝子配列です。
ドイツの "BavPat1" 遺伝子配列は、感染流行のかなり早期に中国から持ち込まれたものの一部です。もう一方の遺伝子配列に対するその類似度 (1つだけの突然変異によって分離されている) は早期のドイツのクラスタに由来する、ヨーロッパでの未検出 ("cryptic"な) 伝染を示唆しえます。
また、ヨーロッパに2度独立して持ち込まれた結果である可能性もあります。まだ採取されていないどこかの遺伝子配列が、"BavPat1 と他方のクラスタの間に入る可能性があります。現時点では、私たちはどちらのシナリオが正しいかを明言できません。
COVID-19のいくつかの症例がシアトル大都市圏 (Greater Seattle area) とアメリカ全域で報告されています。新たに分離され解析が行われた症例は、1月半ばに同じ地域で単離されたケースと遺伝的に強く関連します。
これには 2 つの説明の可能性があります。ウイルスは少なくとも2度シアトル大都市圏 (Greater Seattle area) に中国の共通の感染源から持ち込まれた可能性があります。他方の説明としては、一定期間の間、ウイルスは未検出の状態で伝搬していたというものです。
Trevor Bedford (Nextstrainの共同設立者) はこれらの可能性については素晴らしい投稿をしています。ここで閲覧することができます。
残ったワシントン (Washington) の解析からは別なことがわかります。これらの遺伝子配列は、シアトル大都市圏 (Greater Seattle area) 由来の遺伝子配列とともにクラスタを形成します。このことはコミュニティでの拡散があること、SARS-CoV-2 ウイルスが当該のエリアですでにある一定の期間伝搬していることを強く示唆します。
遺伝子の配列解析が行われた症例の集合の、最新の共通祖先 (つまり tMRCA) の時間は、これらの配列解析された症例が、最後に共通の祖先を持ったのがいつかを示します。下の図に示すように、この時間は、ウイルスが最初に人間の集団に侵入したときと同じくらい早い場合もありますが、下の図に示すように、かなり遅い場合もあります。
## 感染が流行したウイルスの共通祖先の出現日
いくつかの研究グループや個人が、最近の共通の祖先の時間を推定しました [Rambautによるこの投稿](http://virological.org/t/phylodynamic-analysis-of-sars-cov-2-update-2020-03-06/420)か[T Stadlerによるこの投稿](http://virological.org/t/evolutionary-epidemiological-analysis-of-93-genomes)を参照してください。
全遺伝系統の共通の祖先は、11月中頃と12月中頃の間に現れた可能性が最も高いです。これは[武漢海産食品市場での初期のクラスタ](http://virological.org/t/phylodynamic-analysis-of-sars-cov-2-update-2020-03-06/420) から派生した、現在遺伝子解析されているすべての事例と一致します。
<div>
<img alt="estimate of the tMRCA using Bayesian phylogenetics" width="500" src="https://raw.githubusercontent.com/nicfel/nCov-Nicola/master/figures/beast_coal-tmrca_2020303.png"/>
</div>
私たちはこの感染症流行の中において、関係したすべての科学者、中国で働いている科学者による驚くべきタイムリーな研究に謝意を表します。これらのような解析は、ゲノム情報とメタデータの迅速な共有を通じてのみ可能です。
また、これらのデータをアップロードおよび共有できるプラットフォームを提供してくれた GISAID にも感謝します。
私たちはこれらの研修室によって集積されたデータに感謝します:
* Centre for Infectious Diseases and Microbiology Laboratory Services
* Pathology Queensland
* Monash Medical Centre
* National Institute for Viral Disease Control and Prevention, China CDC
* KU Leuven, Clinical and Epidemiological Virology
* Hospital Israelita Albert Einstein
* Virology Unit, Institut Pasteur du Cambodge.
* BCCDC Public Health Laboratory
* Yongchuan District Center for Disease Control and Prevention
* Zhongxian Center for Disease Control and Prevention
* Respiratory Virus Unit, Microbiology Services Colindale, Public Health England
* Lapland Central Hospital
* HUS Diagnostiikkakeskus, Hallinto
* Guangdong Provincial Center for Diseases Control and Prevention; Guangdong Provincial Public Health
* Department of Infectious and Tropical Diseases, Bichat Claude Bernard Hospital, Paris
* Sorbonne Universite, Inserm et Assistance Publique-Hopitaux de Paris (Pitie Salpetriere)
* CNR Virus des Infections Respiratoires - France SUD
* Fujian Center for Disease Control and Prevention
* State Health Office Baden-Wuerttemberg
* Charite Universitatsmedizin Berlin, Institute of Virology; Institut fur Mikrobiologie der Bundeswehr, Munich
* Guangdong Provincial Center for Diseases Control and Prevention; Guangdong Provinical Public Health
* Guangdong Provincial Center for Diseases Control and Prevention;Guangdong Provincial Institute of Public Health
* Hangzhou Center for Disease and Control Microbiology Lab
* Hangzhou Center for Disease Control and Prevention
* Second Hospital of Anhui Medical University
* Hong Kong Department of Health
* Department of Infectious Diseases, Istituto Superiore di Sanita, Roma , Italy
* INMI Lazzaro Spallanzani IRCCS
* Department of Infectious Diseases, Istituto Superiore di Sanita, Rome, Italy
* Department of Virology III, National Institute of Infectious Diseases
* Dept. of Virology III, National Institute of Infectious Diseases
* Dept. of Pathology, National Institute of Infectious Diseases
* NHC Key laboratory of Enteric Pathogenic Microbiology, Institute of Pathogenic Microbiology
* Jingzhou Center for Disease Control and Prevention
* Division of Viral Diseases, Center for Laboratory Control of Infectious Diseases, Korea Centers for Diseases Control and Prevention
* Instituto Nacional de Enfermedades Respiratorias
* National Influenza Centre, National Public Health Laboratory, Kathmandu, Nepal
* Bamrasnaradura Hospital
* The University of Hong Kong - Shenzhen Hospital
* Shenzhen Third People's Hospital
* Shenzhen Key Laboratory of Pathogen and Immunity, National Clinical Research Center for Infectious Disease, Shenzhen Third People's Hospital
* Singapore General Hospital
* National Public Health Laboratory, National Centre for Infectious Diseases
* National Public Health Laboratory
* National Centre for Infectious Diseases
* Singapore General Hospital, Molecular Laboratory, Division of Pathology
* Korea Centers for Disease Control & Prevention (KCDC) Center for Laboratory Control of Infectious Diseases Division of Viral Diseases
* Serology, Virology and OTDS Laboratories (SAViD), NSW Health Pathology Randwick
* Centers for Disease Control, R.O.C. (Taiwan)
* Taiwan Centers for Disease Control
* Laboratory Medicine
* Department of Laboratory Medicine, National Taiwan University Hospital
* Tianmen Center for Disease Control and Prevention
* Arizona Department of Health Services
* California Department of Public Health
* California Department of Health
* IL Department of Public Health Chicago Laboratory
* Massachusetts Department of Public Health
* Texas Department of State Health Services
* WA State Department of Health
* Washington State Department of Health
* Providence Regional Medical Center
* Wisconsin Department of Health Services
* National Influenza Center - National Institute of Hygiene and Epidemiology (NIHE)
* Wuhan Jinyintan Hospital
* The Central Hospital Of Wuhan
* Union Hospital of Tongji Medical College, Huazhong University of Science and Technology
* CR & Wisco General Hospital
* Wuhan Lung Hospital
* Institute of Pathogen Biology, Chinese Academy of Medical Sciences & Peking Union Medical College
* Institute of Viral Disease Control and Prevention, China CDC
* General Hospital of Central Theater Command of People's Liberation Army of China
* Wuhan Fourth Hospital
* Zhejiang Provincial Center for Disease Control and Prevention
* Wuhan Institute of Virology, Chinese Academy of Sciences
* Shandong First Medical University & Shandong Academy of Medical Sciences
* South China Agricultural University
* Beijing Institute of Microbiology and Epidemiology
これらのデータは GISAID を介して共有されました。 彼らの貢献に感謝します。
右側に、各ラボで共有される特定の遺伝子配列を示します。
The SARS-CoV-2 genomes were generously shared by scientists at these submitting labs:
* NSW Health Pathology - Institute of Clinical Pathology and Medical Research; Westmead Hospital; University of Sydney
* Australia/NSW01/2020
* Australia/NSW05/2020
* Sydney/2/2020
* Public Health Virology Laboratory
* Australia/QLD01/2020
* Australia/QLD02/2020
* Australia/QLD03/2020
* Australia/QLD04/2020
* Collaboration between the University of Melbourne at The Peter Doherty Institute for Infection and Immunity, and the Victorian Infectious Disease Reference Laboratory
* Australia/VIC01/2020
* National Institute for Viral Disease Control & Prevention, CCDC
* Beijing/IVDC-BJ-005/2020
* Chongqing/IVDC-CQ-001/2020
* Jiangsu/IVDC-JS-001/2020
* Jiangxi/IVDC-JX-002/2020
* Shandong/IVDC-SD-001/2020
* Shanghai/IVDC-SH-001/2020
* Sichuan/IVDC-SC-001/2020
* Yunnan/IVDC-YN-003/2020
* KU Leuven, Clinical and Epidemiological Virology
* Belgium/GHB-03021/2020
* Instituto Adolfo Lutz Interdisciplinary Procedures Center Strategic Laboratory
* Brazil/SPBR-01/2020
* Virology Unit, Institut Pasteur du Cambodge (Sequencing done by: Jessica E Manning/Jennifer A Bohl at Malaria and Vector Research Research Laboratory, National Institute of Allergy and Infectious Diseases and Vida Ahyong from Chan-Zuckerberg Biohub)
* Cambodia/0012/2020
* BCCDC Public Health Laboratory
* Canada/BC_37_0-2/2020
* Technology Centre, Guangzhou Customs
* China/IQTC01/2020
* China/IQTC02/2020
* Key Laboratory of Human Diseases, Comparative Medicine, Institute of Laboratory Animal Science
* China/WH-09/2020
* State Key Laboratory of Virology, Wuhan University
* China/WHU01/2020
* China/WHU02/2020
* Chongqing Municipal Center for Disease Control and Prevention
* Chongqing/YC01/2020
* Chongqing/ZX01/2020
* Respiratory Virus Unit, Microbiology Services Colindale, Public Health England
* England/01/2020
* England/02/2020
* England/09c/2020
* Department of Virology, University of Helsinki and Helsinki University Hospital, Helsinki, Finland
* Finland/1/2020
* Department of Virology Faculty of Medicine, Medicum University of Helsinki
* Finland/FIN-25/2020
* Guangdong Provincial Center for Diseases Control and Prevention
* Foshan/20SF207/2020
* Foshan/20SF210/2020
* Foshan/20SF211/2020
* Guangdong/20SF201/2020
* Guangzhou/20SF206/2020
* National Reference Center for Viruses of Respiratory Infections, Institut Pasteur, Paris
* France/IDF0372-isl/2020
* France/IDF0372/2020
* France/IDF0373/2020
* France/IDF0515-isl/2020
* France/IDF0515/2020
* France/IDF0626/2020
* Laboratoire Virpath, CIRI U111, UCBL1, INSERM, CNRS, ENS Lyon
* France/IDF0386-islP1/2020
* France/IDF0386-islP3/2020
* France/IDF0571/2020
* CNR Virus des Infections Respiratoires - France SUD
* France/RA739/2020
* Fujian Center for Disease Control and Prevention
* Fujian/13/2020
* Fujian/8/2020
* Charite Universitatsmedizin Berlin, Institute of Virology
* Germany/Baden-Wuerttemberg-1/2020
* Germany/BavPat1/2020
* Department of Microbiology, Guangdong Provincial Center for Diseases Control and Prevention
* Guangdong/20SF012/2020
* Guangdong/20SF013/2020
* Guangdong/20SF014/2020
* Guangdong/20SF025/2020
* Guangdong/20SF028/2020
* Guangdong/20SF040/2020
* Guangdong Provincial Center for Disease Control and Prevention
* Guangdong/20SF174/2020
* Hangzhou Center for Disease and Control Microbiology Lab
* Hangzhou/HZ-1/2020
* Hangzhou Center for Disease Control and Prevention
* Hangzhou/HZCDC0001/2020
* Second Hospital of Anhui Medical University
* Hefei/2/2020
* National Institute for Viral Disease Control & Prevention, China CDC
* Henan/IVDC-HeN-002/2020
* School of Public Health, The University of Hon g Kong
* HongKong/VB20026565/2020
* HongKong/VM20001061/2020
* The University of Hong Kong
* HongKong/VM20001988/2020
* Nepal/61/2020
* Virology Laboratory, Scientific Department, Army Medical Center
* Italy/CDG1/2020
* Italy/SPL1/2020
* Laboratory of Virology, INMI Lazzaro Spallanzani IRCCS
* Italy/INMI1-cs/2020
* Italy/INMI1-isl/2020
* Pathogen Genomics Center, National Institute of Infectious Diseases
* Japan/AI/I-004/2020
* Japan/KY-V-029/2020
* Japan/NA-20-05-1/2020
* Japan/OS-20-07-1/2020
* Japan/TY-WK-012/2020
* Japan/TY-WK-501/2020
* Japan/TY-WK-521/2020
* Takayuki Hishiki Kanagawa Prefectural Institute of Public Health, Department of Microbiology
* Japan/Hu_DP_Kng_19-020/2020
* Japan/Hu_DP_Kng_19-027/2020
* Jiangsu Provincial Center for Disease Control & Prevention
* Jiangsu/JS01/2020
* Jiangsu/JS02/2020
* Jiangsu/JS03/2020
* Hubei Provincial Center for Disease Control and Prevention
* Jingzhou/HBCDC-HB-01/2020
* Tianmen/HBCDC-HB-07/2020
* Wuhan/HBCDC-HB-01/2019
* Wuhan/HBCDC-HB-02/2019
* Wuhan/HBCDC-HB-02/2020
* Wuhan/HBCDC-HB-03/2019
* Wuhan/HBCDC-HB-03/2020
* Wuhan/HBCDC-HB-04/2019
* Wuhan/HBCDC-HB-04/2020
* Wuhan/HBCDC-HB-05/2020
* Wuhan/HBCDC-HB-06/2020
* Division of Viral Diseases, Center for Laboratory Control of Infectious Diseases, Korea Centers for Diseases Control and Prevention
* Korea/KCDC05/2020
* Korea/KCDC06/2020
* Korea/KCDC07/2020
* Korea/KCDC12/2020
* Korea/KCDC24/2020
* Instituto de Diagnostico y Referencia Epidemiologicos (INDRE)
* Mexico/CDMX/InDRE_01/2020
* 1. Department of Medical Sciences, Ministry of Public Health, Thailand 2. Thai Red Cross Emerging Infectious Diseases - Health Science Centre 3. Department of Disease Control, Ministry of Public Health, Thailand
* Nonthaburi/61/2020
* Nonthaburi/74/2020
* Li Ka Shing Faculty of Medicine, The University of Hong Kong
* Shenzhen/HKU-SZ-002/2020
* Shenzhen/HKU-SZ-005/2020
* Shenzhen Key Laboratory of Pathogen and Immunity, National Clinical Research Center for Infectious Disease, Shenzhen Third People's Hospital
* Shenzhen/SZTH-001/2020
* Shenzhen/SZTH-002/2020
* Shenzhen/SZTH-003/2020
* Shenzhen/SZTH-004/2020
* National Public Health Laboratory
* Singapore/1/2020
* Singapore/11/2020
* National Centre for Infectious Diseases, National Centre for Infectious Diseases
* Singapore/10/2020
* Programme in Emerging Infectious Diseases, Duke-NUS Medical School
* Singapore/2/2020
* Singapore/3/2020
* Singapore/4/2020
* Singapore/5/2020
* Singapore/6/2020
* National Public Health Laboratory, National Centre for Infectious Diseases
* Singapore/7/2020
* Singapore/8/2020
* Singapore/9/2020
* Korea Centers for Disease Control & Prevention (KCDC) Center for Laboratory Control of Infectious Diseases Division of Viral Diseases
* SouthKorea/KCDC03/2020
* Department of Clinical Diagnostics
* SouthKorea/SNU01/2020
* Unit for Laboratory Development and Technology Transfer, Public Health Agency of Sweden
* Sweden/01/2020
* NSW Health Pathology - Institute of Clinical Pathology and Medical Research; Centre for Infectious Diseases and Microbiology Laboratory Services; Westmead Hospital; University of Sydney
* Sydney/3/2020
* Centers for Disease Control, R.O.C. (Taiwan)
* Taiwan/2/2020
* Taiwan Centers for Disease Control
* Taiwan/3/2020
* Taiwan/4/2020
* Department of Laboratory Medicine, Lin-Kou Chang Gung Memorial Hospital, Taoyuan, Taiwan.
* Taiwan/CGMH-CGU-01/2020
* Microbial Genomics Core Lab, National Taiwan University Centers of Genomic and Precision Medicine
* Taiwan/NTU01/2020
* Taiwan/NTU02/2020
* Pathogen Discovery, Respiratory Viruses Branch, Division of Viral Diseases, Centers for Disease Control and Prevention
* USA/AZ1/2020
* USA/CA1/2020
* USA/CA2/2020
* USA/CA3/2020
* USA/CA4/2020
* USA/CA5/2020
* USA/CA6/2020
* USA/CA7/2020
* USA/CA8/2020
* USA/CA9/2020
* USA/IL1/2020
* USA/IL2/2020
* USA/MA1/2020
* USA/TX1/2020
* USA/WA1-A12/2020
* USA/WA1-F6/2020
* USA/WI1/2020
* Division of Viral Diseases, Centers for Disease Control and Prevention
* USA/WA1/2020
* Seattle Flu Study
* USA/WA2/2020
* National Influenza Center - National Institute of Hygiene and Epidemiology (NIHE)
* Vietnam/VR03-38142/2020
* National Institute for Communicable Disease Control and Prevention (ICDC) Chinese Center for Disease Control and Prevention (China CDC)
* Wuhan-Hu-1/2019
* Institute of Pathogen Biology, Chinese Academy of Medical Sciences & Peking Union Medical College
* Wuhan/IPBCAMS-WH-01/2019
* Wuhan/IPBCAMS-WH-02/2019
* Wuhan/IPBCAMS-WH-03/2019
* Wuhan/IPBCAMS-WH-04/2019
* Wuhan/IPBCAMS-WH-05/2020
* National Institute for Viral Disease Control and Prevention, China CDC
* Wuhan/IVDC-HB-01/2019
* Wuhan/IVDC-HB-04/2020
* Wuhan/IVDC-HB-05/2019
* Institute of Viral Disease Control and Prevention, China CDC
* Wuhan/IVDC-HB-envF13-20/2020
* Wuhan/IVDC-HB-envF13-21/2020
* Wuhan/IVDC-HB-envF13/2020
* Wuhan/IVDC-HB-envF54/2020
* BGI & Institute of Microbiology, Chinese Academy of Sciences & Shandong First Medical University & Shandong Academy of Medical Sciences & General Hospital of Central Theater Command of People's Liberation Army of China
* Wuhan/WH01/2019
* Wuhan/WH02/2019
* Wuhan/WH03/2020
* Wuhan/WH04/2020
* Beijing Genomics Institute (BGI)
* Wuhan/WH05/2020
* Wuhan Institute of Virology, Chinese Academy of Sciences
* Wuhan/WIV02/2019
* Wuhan/WIV04/2019
* Wuhan/WIV05/2019
* Wuhan/WIV06/2019
* Wuhan/WIV07/2019
* Department of Microbiology, Zhejiang Provincial Center for Disease Control and Prevention
* Zhejiang/WZ-01/2020
* Zhejiang/WZ-02/2020