| title | authors | authorLinks | affiliations | date | dataset | abstract |
|---|---|---|---|---|---|---|
Analisi genomica della propagazione del COVID-19. Rapporto sulla situazione al 2020-03-05. |
Trevor Bedford, Richard Neher, James Hadfield, Emma Hodcroft, Misja Ilcisin, Nicola Müller |
Fred Hutch, Seattle, USA and Biozentrum, Basel, Switzerland |
2020 March 05 |
Questo rapporto utilizza i dati genomici del COVID-19 pubblicati su GISAID e Genbank. I dati sono utilizzati per stimare il tasso e le modalità di diffusione dell'epidemia virale. Prevediamo di pubblicare rapporti aggiornati non appena saranno prodotti e diffusi nuovi dati. Questo sito web è ottimizzato per la visualizzazione su browser desktop. |
## Sintesi
Utilizzando i 169</tag> genomi pubblicati del COVID-19, abbiamo esaminato la diversità genetica che caratterizza la propagazione del COVID-19 in diverse aree geografiche e determinato la data del loro antenato comune.
Risultati principali:
* Il COVID-19 è stato introdotto in Italia almeno due volte, con successiva propagazione nella popolazione. ([Leggi](https://nextstrain.org/narratives/ncov/sit-rep/it/2020-03-05?n=7)).
* Questo include un gruppo di sequenze genetiche da 6 nazioni differenti dove i casi sembrano essere stati esportati dall'Italia. ([Leggi](https://nextstrain.org/narratives/ncov/sit-rep/it/2020-03-05?n=9)).
* I dati delle sequenze genetiche supportano l'ipotesi di una diffusione inosservata e sostenuta del COVID-19 nell'area di Seattle dalla metà di gennaio. ([Leggi](https://nextstrain.org/narratives/ncov/sit-rep/it/2020-03-05?n=10)).
* Tutti i casi sequenziati inclusi in questa analisi condividono probabilmente un antenato comune tra la metà di novembre e quella di dicembre 2019. ([Leggi](https://nextstrain.org/narratives/ncov/sit-rep/it/2020-03-05?n=11)).
- Informazioni generali sull'epidemia di COVID-19 Wikipedia 2020-03-04
- Materiali forniti dal Centro per il Controllo e la Prevenzione delle Malattie degli Stati Uniti US CDC 2020-01-29
## Risorse sul COVID-19
Di seguito elenchiamo alcune letture utili per familiarizzare con i concetti generali relativi al COVID-19 e al virus che ne è la causa, SARS-CoV-2.
Queste informazioni renderanno più semplice interpretare i dati che presenteremo più avanti.
<div>
<a href="https://nextstrain.org/help/coronavirus/human-CoV"><img alt="immagine al microscopio di coronavirus" width="100" src="https://nextstrain.org/static/ncov_narrative-76cfd610d11ef708d213a3170de9519f.png"/> Informazioni generali sui Coronavirus (in inglese) </a>
<a href="https://nextstrain.org/help/coronavirus/SARS-CoV-2"><img alt="illustrazione di un coronavirus" width="100" src="http://data.nextstrain.org/img_nCoV-CDC.jpg"/> Informazioni sulla recente epidemia di COVID-19 (in inglese) </a>
<a href="https://nextstrain.org/help/general/how-to-read-a-tree"><img alt="disegno di un albero filogenetico" width="100" src="http://data.nextstrain.org/img_toy_alignment_mini.png"/> Come leggere una filogenesi (in inglese) </a>
</div>
## Analisi Nextstrain
Le pagine che seguono contengono un'analisi eseguita utilizzando [Nextstrain](https://nextstrain.org).
Scorrendo lungo la barra laterale sinistra sarà possibile leggere il testo illustrativo relativo alla visualizzazione dei dati genomici nel lato destro della pagina.
Avere a disposizione così rapidamente i genomi completi per un nuovo virus RNA così velocemente è un risultato notevole.
Queste analisi sono state rese possibili grazie alla rapida e libera diffusione dei dati genomici e alla loro interpretazione da parte degli scienziati di tutto il mondo (nella slide finale è possibile leggere i nomi degli autori del sequenziamento).
-
"Don't believe the conspiracy theories you hear about coronavirus & HIV" article 2020-01-31
-
"Baseless Conspiracy Theories Claim New Coronavirus Was Bioengineered" article 2020-02-07
-
"No, The Wuhan Coronavirus Was Not Genetically Engineered To Put Pieces Of HIV In It" article 2020-02-02
-
"Busting coronavirus myths" AFP Fact Check 2020-02-19
## Domande frequenti e convinzioni errate
### Domande frequenti
Sappiamo che molte persone hanno domande sul COVID-19.
Per questo abbiamo creato una guida per provare a rispondere ad alcune delle domande più frequenti [qui](https://nextstrain.org/help/coronavirus/FAQ):
<div>
<a href="https://nextstrain.org/help/coronavirus/FAQ"><img alt="foto di un punto interrogativo" width="100" src="http://data.nextstrain.org/img_question-mark.jpg"/> FAQ sul COVID-19 (in inglese) </a>
</div>
### Convinzioni errate
Stanno circolando diverse idee errate sulle origini del nuovo coronavirus.
Durante epidemie come questa, la diffusione incontrollata di informazioni che sappiamo errate può generare panico e diminuire la fiducia della popolazione negli scienziati e nel governo. Questo può portare a non seguire i consigli più sensati e a non prendere le appropriate precauzioni per evitare il contagio.
Nel tentativo di spiegare perché queste convinzioni non sono corrette, gli scienziati hanno affrontato queste teorie nelle pagine che seguono (in inglese):
<div>
<a href="http://virological.org/t/ncovs-relationship-to-bat-coronaviruses-recombination-signals-no-snakes-no-evidence-the-2019-ncov-lineage-is-recombinant/331"><img alt="foto di un serpente" width="100" src="http://data.nextstrain.org/img_snake-freeToUse.jpg"/> Il virus SARS-CoV-2 ha origine nei serpenti? (tecnico) </a>
<a href="https://twitter.com/trvrb/status/1223666856923291648"><img alt="illustrazione dell'HIV" width="100" src="http://data.nextstrain.org/img_HIV-wiki.jpg"/> L'idea dell'HIV 'manipolato' (thread su Twitter) </a>
</div>
Qui presentiamo la filogenesi dei 161 ceppi di SARS-CoV-2 (il virus che causa COVID-19) che sono stati pubblicati. Informazioni su come l'analisi è stata condotta sono disponibili in questo repository GitHub.
I colori rappresentano la regione all'interno del paese o lo stato in cui il virus è stato isolato, con l'asse x che rappresenta la data del campionamento. L'asse y mostra come le sequenze sono collegate e non ha unità di misura.
Le date del campionamento sono utili, ma non sempre mostrano esattamente come due sequenze sono correlate geneticamente - due sequenze identiche potrebbero avere differenti date di campionamento, e risultare così molto distanti in questa visualizzazione.
Possiamo cambiare la visualizzazione in modo che l'asse x mostri invece la 'divergenza'.
Si noti che molte sequenze genetiche che prima sembravano differenti, ora sono nella stessa linea verticale. È possibile scorrere su e giù tra la slide precedente e quella attuale per vedere come l'albero filogenetico cambia.
La divergenza è misurata come il numero di cambiamenti (mutazioni) nel genoma. Alcune sequenze possono avere zero mutazioni; in tal caso sono identiche alla radice (al centro) dell'albero. Altri virus hanno tra una e undici mutazioni.
Sequenziare il genoma di un nuovo virus RNA nel corso di un'epidemia è arduo. Alcune delle differenze osservate in queste sequenze possono essere errori di sequenziamento e non mutazioni vere e proprie. Inserzioni, delezioni e differenze nelle parti terminali del genoma sono molto probabilmente errori e dunque sono state mascherate in questa analisi.
In base a ciò che cerchiamo di evidenziare, a volte mostreremo l'albero filogenetico in una visualizzazione temporale, altre volte in modalità 'divergenza'.
Al momento abbiamo sequenze da campioni presi in cinque diversi continenti. Mentre i primi casi erano tutti collegati direttamente a casi di Wuhan, associati al focolaio originato al mercato del pesce, ora osserviamo molti casi differenti. Questi sono correlati a una diffusione nella popolazione o a un'importazione da regioni al di fuori della Cina.
In generale, introduzioni ripetute da una riserva virale animale mostreranno una diversità significativa (questo si è verificato per Lassa, Ebola, MERS-CoV e influenza aviaria). L'osservazione di un cluster di infezioni umane così importante può essere spiegata da un'epidemia che deriva da un singolo evento di introduzione zoonotica nella popolazione umana, seguito da una diffusione uomo-uomo.
Al momento abbiamo a disposizione 3 sequenze dall'Italia, due delle quali provengono da Roma e una dalla Lombardia, nel nord Italia.
Queste 3 sequenze condividono un antenato comune nelle fasi iniziali dell'epidemia (vicino alla base dell'albero, sulla sinistra). Questo suggerisce fortemente che vi siano state almeno due introduzioni con diffusione nella popolazione in Italia.
Il Dr. Nuno Faria et al. propone qui (in inglese) un'eccellente analisi di come sequenze globali, tra cui quella brasiliana, mostrano che "l'epidemia nel nord Italia è stata probabilmente il risultato di introduzioni multiple nella regione e non da un'unica fonte."
Le due sequenze da Roma (29 gennaio 2020) sono direttamente connesse tra loro e sono entrambe correlate a viaggi in Cina.
Siamo passati alla visualizzazione in modalità 'divergenza' per mostrare che le due sequenze italiane sono identiche, mentre altre sequenze vicine (da Inghilterra, Brasile, Svizzera, USA e Cina) sono separate da quella italiana da 2-4 mutazioni.
In ogni caso la sequenza brasiliana (SPBR-02) è correlata ad un viaggio a Milano, in Lombardia, e si pensa che la sequenza svizzera sia anch'essa correlata ad un recente viaggio in Italia. Non sappiamo nulla circa gli spostamenti del campione statunitense. Il campione England/09c isolato è di diretta importazione dalla Cina.
Il campione inglese (dalla Cina) si trova tra i precedenti campioni italiani da Roma ed i casi con legami certi (Svizzera, Brasile) o ipotizzati (USA) ad un viaggio in Italia. Ciò significa che non è possibile dare per certo che le prime sequenze italiane e qulle più recenti associate all'Italia siano direttamente collegate. I campioni più recenti potrebbero derivare da un'ulteriore introduzione indipendente in Italia.
La sequenza dalla Lombardia (Italy/CDG1/2020) è nel gruppo con sequenze da Messico, Germania, Brasile e Finlandia, con legami noti a viaggi in Italia e che molto probabilmente hanno contratto l'infezione nello stesso paese.
La sequenza tedesca "BavPat1" è parte di una introduzione dalla Cina in una fase molto precedente dell'epidemia. La sua somiglianza con le altre sequenze del gruppo (sono separate da una singola mutazione) potrebbe indicare una trasmissione inosservata ("criptica") in Europa, nata da questo gruppo tedesco.
Potrebbe anche essere il risultato di due separate introduzioni in Europa, una sequenza non campionata di provenienza sconosciuta potrebbe collocarsi tra 'BavPat1' e il resto del gruppo. Al momento non possiamo affermare con certezza quale dei due scenari sia quello corretto.
Al momento sono segnalati numerosi casi di COVID-19 nell'area di Seattle e negli USA in generale. Il caso recentemente isolato e sequenziato dalla Snohomish County (a nord di Seattle), è geneticamente strettamente correlato ad un caso isolato a metà gennaio nella stessa area.
Ci sono due possibili spiegazioni per questo fenomeno. Il virus potrebbe essere stato introdotto almeno due volte nell'area di Seattle da una fonte comune in Cina. Tuttavia, un'altra spiegazione plausibile è che il virus stesse circolando nell'area inosservato da diverso tempo.
Trevor Bedford (co-fondatore di Nextstrain) ha scritto un ottimo blog post su queste ipotesi, che è possibile leggere qui.
Le restanti sequenze dello stato di Washington ci dicono qualcosa di diverso: queste sequenze formano un gruppo unico. Questo fatto suggerisce che ci sia un focolaio attivo all’interno della comunita’, e che quindi il virus SARS-CoV2 sia in giro nell’area già da un po’ di tempo.
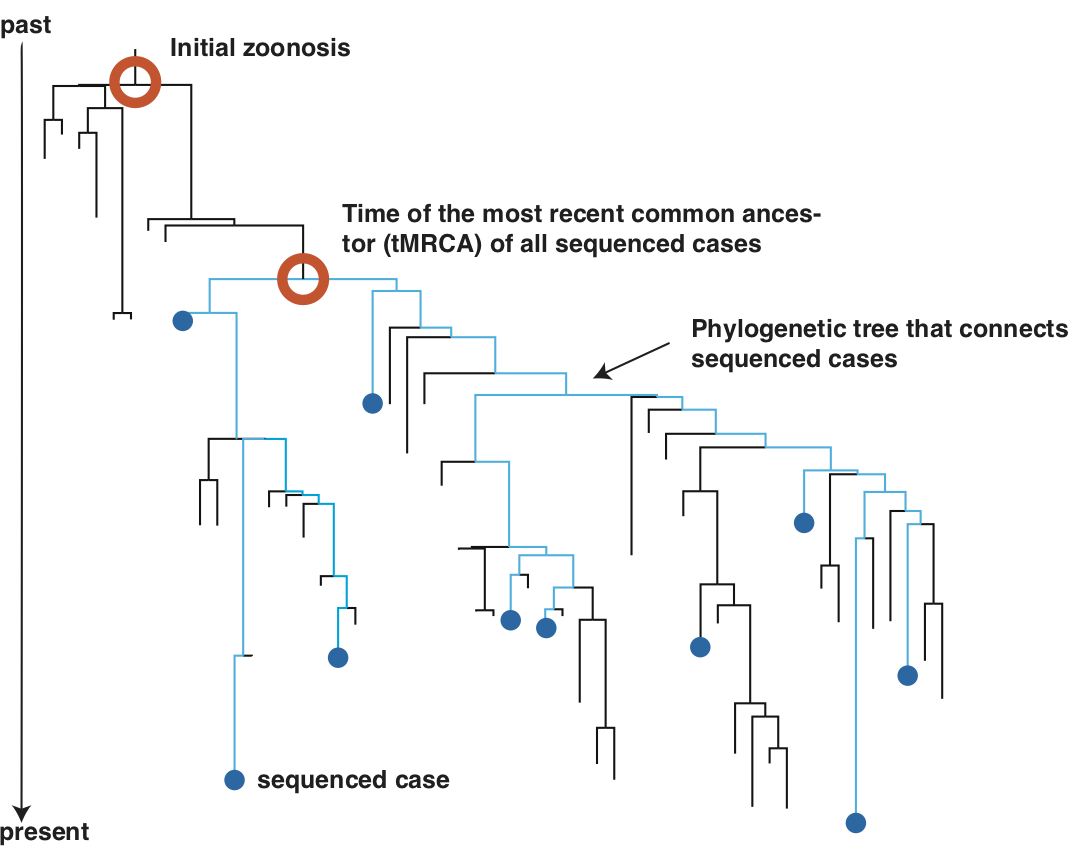
La datazione del più recente antenato comune (detta tMRCA) di un insieme di casi sequenziati indica quando questi casi hanno condiviso per l'ultima volta un comune antenato. Ciò può coincidere con il momento in cui un virus è entrato per la prima volta nella popolazione umana, ma può collocarsi anche considerevolmente più tardi, come mostrato nella figura successiva.
## Datazione dell'antenato comune dei virus epidemici
Molti scienziati e gruppi di ricerca hanno stimato la data del più recente antenato comune, vedi [questo post di A. Rambaut (in inglese)](http://virological.org/t/phylodynamic-analysis-of-sars-cov-2-update-2020-03-06/420) oppure [questo post di T. Stadler (in inglese)](http://virological.org/t/evolutionary-epidemiological-analysis-of-93-genomes).
L'antenato comune a tutte le sequenze si trova probabilmente tra metà novembre e metà dicembre.
Questo sarebbe coerente con l'ipotesi che tutti i casi attualmente sequenziati discendano dal
[gruppo iniziale di casi al mercato del pesce di Wuhan (in inglese)](http://virological.org/t/phylodynamic-analysis-of-sars-cov-2-update-2020-03-06/420).
<div>
<img alt="estimate of the tMRCA using Bayesian phylogenetics" width="500" src="https://raw.githubusercontent.com/nicfel/nCov-Nicola/master/figures/beast_coal-tmrca_2020303.png"/>
</div>
Ringraziamo tutti gli scienziati al lavoro su questa epidemia, e in particolare quelli che lavorano in Cina, per lo straordinario e tempestivo lavoro fatto. È solo attraverso la rapida condivisione di dati e metadati genomici che analisi come questa sono possibili.
Siamo anche grati a GISAID per aver fornito la piattaforma su cui questi dati sono stati caricati e condivisi.
Ringraziamo i seguenti laboratori per i dati raccolti:
* Centre for Infectious Diseases and Microbiology Laboratory Services
* Pathology Queensland
* Monash Medical Centre
* National Institute for Viral Disease Control and Prevention, China CDC
* KU Leuven, Clinical and Epidemiological Virology
* Hospital Israelita Albert Einstein
* Virology Unit, Institut Pasteur du Cambodge.
* BCCDC Public Health Laboratory
* Yongchuan District Center for Disease Control and Prevention
* Zhongxian Center for Disease Control and Prevention
* Respiratory Virus Unit, Microbiology Services Colindale, Public Health England
* Lapland Central Hospital
* HUS Diagnostiikkakeskus, Hallinto
* Guangdong Provincial Center for Diseases Control and Prevention; Guangdong Provincial Public Health
* Department of Infectious and Tropical Diseases, Bichat Claude Bernard Hospital, Paris
* Sorbonne Universite, Inserm et Assistance Publique-Hopitaux de Paris (Pitie Salpetriere)
* CNR Virus des Infections Respiratoires - France SUD
* Fujian Center for Disease Control and Prevention
* State Health Office Baden-Wuerttemberg
* Charite Universitatsmedizin Berlin, Institute of Virology; Institut fur Mikrobiologie der Bundeswehr, Munich
* Guangdong Provincial Center for Diseases Control and Prevention; Guangdong Provinical Public Health
* Guangdong Provincial Center for Diseases Control and Prevention;Guangdong Provincial Institute of Public Health
* Hangzhou Center for Disease and Control Microbiology Lab
* Hangzhou Center for Disease Control and Prevention
* Second Hospital of Anhui Medical University
* Hong Kong Department of Health
* Department of Infectious Diseases, Istituto Superiore di Sanita, Roma , Italy
* INMI Lazzaro Spallanzani IRCCS
* Department of Infectious Diseases, Istituto Superiore di Sanita, Rome, Italy
* Department of Virology III, National Institute of Infectious Diseases
* Dept. of Virology III, National Institute of Infectious Diseases
* Dept. of Pathology, National Institute of Infectious Diseases
* NHC Key laboratory of Enteric Pathogenic Microbiology, Institute of Pathogenic Microbiology
* Jingzhou Center for Disease Control and Prevention
* Division of Viral Diseases, Center for Laboratory Control of Infectious Diseases, Korea Centers for Diseases Control and Prevention
* Instituto Nacional de Enfermedades Respiratorias
* National Influenza Centre, National Public Health Laboratory, Kathmandu, Nepal
* Bamrasnaradura Hospital
* The University of Hong Kong - Shenzhen Hospital
* Shenzhen Third People's Hospital
* Shenzhen Key Laboratory of Pathogen and Immunity, National Clinical Research Center for Infectious Disease, Shenzhen Third People's Hospital
* Singapore General Hospital
* National Public Health Laboratory, National Centre for Infectious Diseases
* National Public Health Laboratory
* National Centre for Infectious Diseases
* Singapore General Hospital, Molecular Laboratory, Division of Pathology
* Korea Centers for Disease Control & Prevention (KCDC) Center for Laboratory Control of Infectious Diseases Division of Viral Diseases
* Serology, Virology and OTDS Laboratories (SAViD), NSW Health Pathology Randwick
* Centers for Disease Control, R.O.C. (Taiwan)
* Taiwan Centers for Disease Control
* Laboratory Medicine
* Department of Laboratory Medicine, National Taiwan University Hospital
* Tianmen Center for Disease Control and Prevention
* Arizona Department of Health Services
* California Department of Public Health
* California Department of Health
* IL Department of Public Health Chicago Laboratory
* Massachusetts Department of Public Health
* Texas Department of State Health Services
* WA State Department of Health
* Washington State Department of Health
* Providence Regional Medical Center
* Wisconsin Department of Health Services
* National Influenza Center - National Institute of Hygiene and Epidemiology (NIHE)
* Wuhan Jinyintan Hospital
* The Central Hospital Of Wuhan
* Union Hospital of Tongji Medical College, Huazhong University of Science and Technology
* CR & Wisco General Hospital
* Wuhan Lung Hospital
* Institute of Pathogen Biology, Chinese Academy of Medical Sciences & Peking Union Medical College
* Institute of Viral Disease Control and Prevention, China CDC
* General Hospital of Central Theater Command of People's Liberation Army of China
* Wuhan Fourth Hospital
* Zhejiang Provincial Center for Disease Control and Prevention
* Wuhan Institute of Virology, Chinese Academy of Sciences
* Shandong First Medical University & Shandong Academy of Medical Sciences
* South China Agricultural University
* Beijing Institute of Microbiology and Epidemiology
Questi dati sono stati condivisi attraverso GISAID, a cui siamo grati per il contributo.
A destra forniamo le sequenze specifiche condivise da ciascun laboratorio.
I genomi di SARS-CoV-2 sono stati generosamente condivisi dagli scienziati di questi laboratori:
* NSW Health Pathology - Institute of Clinical Pathology and Medical Research; Westmead Hospital; University of Sydney
* Australia/NSW01/2020
* Australia/NSW05/2020
* Sydney/2/2020
* Public Health Virology Laboratory
* Australia/QLD01/2020
* Australia/QLD02/2020
* Australia/QLD03/2020
* Australia/QLD04/2020
* Collaboration between the University of Melbourne at The Peter Doherty Institute for Infection and Immunity, and the Victorian Infectious Disease Reference Laboratory
* Australia/VIC01/2020
* National Institute for Viral Disease Control & Prevention, CCDC
* Beijing/IVDC-BJ-005/2020
* Chongqing/IVDC-CQ-001/2020
* Jiangsu/IVDC-JS-001/2020
* Jiangxi/IVDC-JX-002/2020
* Shandong/IVDC-SD-001/2020
* Shanghai/IVDC-SH-001/2020
* Sichuan/IVDC-SC-001/2020
* Yunnan/IVDC-YN-003/2020
* KU Leuven, Clinical and Epidemiological Virology
* Belgium/GHB-03021/2020
* Instituto Adolfo Lutz Interdisciplinary Procedures Center Strategic Laboratory
* Brazil/SPBR-01/2020
* Virology Unit, Institut Pasteur du Cambodge (Sequencing done by: Jessica E Manning/Jennifer A Bohl at Malaria and Vector Research Research Laboratory, National Institute of Allergy and Infectious Diseases and Vida Ahyong from Chan-Zuckerberg Biohub)
* Cambodia/0012/2020
* BCCDC Public Health Laboratory
* Canada/BC_37_0-2/2020
* Technology Centre, Guangzhou Customs
* China/IQTC01/2020
* China/IQTC02/2020
* Key Laboratory of Human Diseases, Comparative Medicine, Institute of Laboratory Animal Science
* China/WH-09/2020
* State Key Laboratory of Virology, Wuhan University
* China/WHU01/2020
* China/WHU02/2020
* Chongqing Municipal Center for Disease Control and Prevention
* Chongqing/YC01/2020
* Chongqing/ZX01/2020
* Respiratory Virus Unit, Microbiology Services Colindale, Public Health England
* England/01/2020
* England/02/2020
* England/09c/2020
* Department of Virology, University of Helsinki and Helsinki University Hospital, Helsinki, Finland
* Finland/1/2020
* Department of Virology Faculty of Medicine, Medicum University of Helsinki
* Finland/FIN-25/2020
* Guangdong Provincial Center for Diseases Control and Prevention
* Foshan/20SF207/2020
* Foshan/20SF210/2020
* Foshan/20SF211/2020
* Guangdong/20SF201/2020
* Guangzhou/20SF206/2020
* National Reference Center for Viruses of Respiratory Infections, Institut Pasteur, Paris
* France/IDF0372-isl/2020
* France/IDF0372/2020
* France/IDF0373/2020
* France/IDF0515-isl/2020
* France/IDF0515/2020
* France/IDF0626/2020
* Laboratoire Virpath, CIRI U111, UCBL1, INSERM, CNRS, ENS Lyon
* France/IDF0386-islP1/2020
* France/IDF0386-islP3/2020
* France/IDF0571/2020
* CNR Virus des Infections Respiratoires - France SUD
* France/RA739/2020
* Fujian Center for Disease Control and Prevention
* Fujian/13/2020
* Fujian/8/2020
* Charite Universitatsmedizin Berlin, Institute of Virology
* Germany/Baden-Wuerttemberg-1/2020
* Germany/BavPat1/2020
* Department of Microbiology, Guangdong Provincial Center for Diseases Control and Prevention
* Guangdong/20SF012/2020
* Guangdong/20SF013/2020
* Guangdong/20SF014/2020
* Guangdong/20SF025/2020
* Guangdong/20SF028/2020
* Guangdong/20SF040/2020
* Guangdong Provincial Center for Disease Control and Prevention
* Guangdong/20SF174/2020
* Hangzhou Center for Disease and Control Microbiology Lab
* Hangzhou/HZ-1/2020
* Hangzhou Center for Disease Control and Prevention
* Hangzhou/HZCDC0001/2020
* Second Hospital of Anhui Medical University
* Hefei/2/2020
* National Institute for Viral Disease Control & Prevention, China CDC
* Henan/IVDC-HeN-002/2020
* School of Public Health, The University of Hon g Kong
* HongKong/VB20026565/2020
* HongKong/VM20001061/2020
* The University of Hong Kong
* HongKong/VM20001988/2020
* Nepal/61/2020
* Virology Laboratory, Scientific Department, Army Medical Center
* Italy/CDG1/2020
* Italy/SPL1/2020
* Laboratory of Virology, INMI Lazzaro Spallanzani IRCCS
* Italy/INMI1-cs/2020
* Italy/INMI1-isl/2020
* Pathogen Genomics Center, National Institute of Infectious Diseases
* Japan/AI/I-004/2020
* Japan/KY-V-029/2020
* Japan/NA-20-05-1/2020
* Japan/OS-20-07-1/2020
* Japan/TY-WK-012/2020
* Japan/TY-WK-501/2020
* Japan/TY-WK-521/2020
* Takayuki Hishiki Kanagawa Prefectural Institute of Public Health, Department of Microbiology
* Japan/Hu_DP_Kng_19-020/2020
* Japan/Hu_DP_Kng_19-027/2020
* Jiangsu Provincial Center for Disease Control & Prevention
* Jiangsu/JS01/2020
* Jiangsu/JS02/2020
* Jiangsu/JS03/2020
* Hubei Provincial Center for Disease Control and Prevention
* Jingzhou/HBCDC-HB-01/2020
* Tianmen/HBCDC-HB-07/2020
* Wuhan/HBCDC-HB-01/2019
* Wuhan/HBCDC-HB-02/2019
* Wuhan/HBCDC-HB-02/2020
* Wuhan/HBCDC-HB-03/2019
* Wuhan/HBCDC-HB-03/2020
* Wuhan/HBCDC-HB-04/2019
* Wuhan/HBCDC-HB-04/2020
* Wuhan/HBCDC-HB-05/2020
* Wuhan/HBCDC-HB-06/2020
* Division of Viral Diseases, Center for Laboratory Control of Infectious Diseases, Korea Centers for Diseases Control and Prevention
* Korea/KCDC05/2020
* Korea/KCDC06/2020
* Korea/KCDC07/2020
* Korea/KCDC12/2020
* Korea/KCDC24/2020
* Instituto de Diagnostico y Referencia Epidemiologicos (INDRE)
* Mexico/CDMX/InDRE_01/2020
* 1. Department of Medical Sciences, Ministry of Public Health, Thailand 2. Thai Red Cross Emerging Infectious Diseases - Health Science Centre 3. Department of Disease Control, Ministry of Public Health, Thailand
* Nonthaburi/61/2020
* Nonthaburi/74/2020
* Li Ka Shing Faculty of Medicine, The University of Hong Kong
* Shenzhen/HKU-SZ-002/2020
* Shenzhen/HKU-SZ-005/2020
* Shenzhen Key Laboratory of Pathogen and Immunity, National Clinical Research Center for Infectious Disease, Shenzhen Third People's Hospital
* Shenzhen/SZTH-001/2020
* Shenzhen/SZTH-002/2020
* Shenzhen/SZTH-003/2020
* Shenzhen/SZTH-004/2020
* National Public Health Laboratory
* Singapore/1/2020
* Singapore/11/2020
* National Centre for Infectious Diseases, National Centre for Infectious Diseases
* Singapore/10/2020
* Programme in Emerging Infectious Diseases, Duke-NUS Medical School
* Singapore/2/2020
* Singapore/3/2020
* Singapore/4/2020
* Singapore/5/2020
* Singapore/6/2020
* National Public Health Laboratory, National Centre for Infectious Diseases
* Singapore/7/2020
* Singapore/8/2020
* Singapore/9/2020
* Korea Centers for Disease Control & Prevention (KCDC) Center for Laboratory Control of Infectious Diseases Division of Viral Diseases
* SouthKorea/KCDC03/2020
* Department of Clinical Diagnostics
* SouthKorea/SNU01/2020
* Unit for Laboratory Development and Technology Transfer, Public Health Agency of Sweden
* Sweden/01/2020
* NSW Health Pathology - Institute of Clinical Pathology and Medical Research; Centre for Infectious Diseases and Microbiology Laboratory Services; Westmead Hospital; University of Sydney
* Sydney/3/2020
* Centers for Disease Control, R.O.C. (Taiwan)
* Taiwan/2/2020
* Taiwan Centers for Disease Control
* Taiwan/3/2020
* Taiwan/4/2020
* Department of Laboratory Medicine, Lin-Kou Chang Gung Memorial Hospital, Taoyuan, Taiwan.
* Taiwan/CGMH-CGU-01/2020
* Microbial Genomics Core Lab, National Taiwan University Centers of Genomic and Precision Medicine
* Taiwan/NTU01/2020
* Taiwan/NTU02/2020
* Pathogen Discovery, Respiratory Viruses Branch, Division of Viral Diseases, Centers for Disease Control and Prevention
* USA/AZ1/2020
* USA/CA1/2020
* USA/CA2/2020
* USA/CA3/2020
* USA/CA4/2020
* USA/CA5/2020
* USA/CA6/2020
* USA/CA7/2020
* USA/CA8/2020
* USA/CA9/2020
* USA/IL1/2020
* USA/IL2/2020
* USA/MA1/2020
* USA/TX1/2020
* USA/WA1-A12/2020
* USA/WA1-F6/2020
* USA/WI1/2020
* Division of Viral Diseases, Centers for Disease Control and Prevention
* USA/WA1/2020
* Seattle Flu Study
* USA/WA2/2020
* National Influenza Center - National Institute of Hygiene and Epidemiology (NIHE)
* Vietnam/VR03-38142/2020
* National Institute for Communicable Disease Control and Prevention (ICDC) Chinese Center for Disease Control and Prevention (China CDC)
* Wuhan-Hu-1/2019
* Institute of Pathogen Biology, Chinese Academy of Medical Sciences & Peking Union Medical College
* Wuhan/IPBCAMS-WH-01/2019
* Wuhan/IPBCAMS-WH-02/2019
* Wuhan/IPBCAMS-WH-03/2019
* Wuhan/IPBCAMS-WH-04/2019
* Wuhan/IPBCAMS-WH-05/2020
* National Institute for Viral Disease Control and Prevention, China CDC
* Wuhan/IVDC-HB-01/2019
* Wuhan/IVDC-HB-04/2020
* Wuhan/IVDC-HB-05/2019
* Institute of Viral Disease Control and Prevention, China CDC
* Wuhan/IVDC-HB-envF13-20/2020
* Wuhan/IVDC-HB-envF13-21/2020
* Wuhan/IVDC-HB-envF13/2020
* Wuhan/IVDC-HB-envF54/2020
* BGI & Institute of Microbiology, Chinese Academy of Sciences & Shandong First Medical University & Shandong Academy of Medical Sciences & General Hospital of Central Theater Command of People's Liberation Army of China
* Wuhan/WH01/2019
* Wuhan/WH02/2019
* Wuhan/WH03/2020
* Wuhan/WH04/2020
* Beijing Genomics Institute (BGI)
* Wuhan/WH05/2020
* Wuhan Institute of Virology, Chinese Academy of Sciences
* Wuhan/WIV02/2019
* Wuhan/WIV04/2019
* Wuhan/WIV05/2019
* Wuhan/WIV06/2019
* Wuhan/WIV07/2019
* Department of Microbiology, Zhejiang Provincial Center for Disease Control and Prevention
* Zhejiang/WZ-01/2020
* Zhejiang/WZ-02/2020