| title | authors | authorLinks | affiliations | date | dataset | abstract |
|---|---|---|---|---|---|---|
Analyse génomique de la propagation du nCoV. Rapport de situation au 2020-03-05. |
Trevor Bedford, Richard Neher, James Hadfield, Emma Hodcroft, Misja Ilcisin, Nicola Müller |
États-Unis, Seattle, USA et Biozentrum, Bâle, Suisse |
5 mars 2020 |
Ce rapport utilise des données génomiques du nouveau coronavirus (nCoV) rendues publiques sur GISAID et Genbank afin d'estimer la vitesse et les modalités de propagation de l'épidémie virale. Nous prévoyons d'émettre des rapports de situation à mesure que de nouvelles données sont produites et partagées. Ce site web est optimisé pour être affiché sur un ordinateur de bureau. |
## Résumé exécutif
À l'aide de 169</tag> génomes COVID-19 partagés publiquement, nous avons examiné la diversité génétique pour caractériser la propagation de COVID-19 dans différentes régions et en déduire la date de leur ancêtre commun.
Principaux résultats :
* COVID-19 a été introduit en Italie au moins deux fois avec par la suite de la transmission au sein de la communauté ([lien](https://nextstrain.org/narratives/ncov/sit-rep/fr/2020-03-05?n=7)).
* Cela inclut un regroupement de séquences de 6 pays différents où il semble que des cas ont été exportés d’Italie ([lien](https://nextstrain.org/narratives/ncov/sit-rep/fr/2020-03-05?n=9)).
* L’analyse des séquences génétiques supporte l’hypothèse d’une propagation non détectée de COVID-19 dans la région de Seattle depuis la mi-janvier ([lien](https://nextstrain.org/narratives/ncov/sit-rep/fr/2020-03-05?n=10)).
* Tous les cas séquencés inclus dans cette analyse partagent vraisemblablement un ancêtre commun apparu entre la mi-novembre et la mi-décembre 2019. ([lien](https://nextstrain.org/narratives/ncov/sit-rep/fr/2020-03-05?n=11)).
- Informations générales sur les coronavirus sur Wikipedia 2020-01-30
- Documents fournis par l'US CDC 2020-01-29
## Ressources sur COVID-19
Ci-dessous, nous avons préparé quelques ressources (en anglais) pour vous familiariser avec COVID-19 et le virus qui la provoque, SARS-CoV-2.
Ces informations faciliteront l'interprétation des données que nous présentons dans ce récit.
<div>
<a href="https://nextstrain.org/help/coronavirus/human-CoV"><img alt="image de microscopie des coronavirus" width="100" src="https://nextstrain.org/static/ncov_narrative-76cfd610d11ef708d213a3170de9519f.png"/> Informations sur les coronavirus </a>
<a href="https://nextstrain.org/help/coronavirus/SARS-CoV-2"><img alt="illustration d'un coronavirus" width="100" src="http://data.nextstrain.org/img_nCoV-CDC.jpg"/> Contexte de l'émergence de COVID-19 </a>
<a href="https://nextstrain.org/help/general/how-to-read-a-tree"><img alt="illustration d'un arbre phylogénétique" width="100" src="http://data.nextstrain.org/img_toy_alignment_mini.png"/> Comment lire les phylogénies </a>
</div>
## Récits Nextstrain
Les pages suivantes contiennent des analyses effectuées à l'aide de [Nextstrain](https://nextstrain.org).
En faisant défiler la barre latérale gauche, vous découvrirez des paragraphes de texte avec sur le côté droit une visualisation correspondant aux données génomiques.
Obtenir des génomes complets d'un nouveau et grand virus à ARN si rapidement est une réalisation remarquable. Ces analyses ont été rendues possibles grâce au partage rapide et ouvert de données génomiques et aux interprétations de scientifiques du monde entier (voir la diapositive finale pour une visualisation des crédits des séquençages).
-
"Don't believe the conspiracy theories you hear about coronavirus & HIV" article 2020-01-31
-
"Baseless Conspiracy Theories Claim New Coronavirus Was Bioengineered" article 2020-02-07
-
"No, The Wuhan Coronavirus Was Not Genetically Engineered To Put Pieces Of HIV In It" article 2020-02-02
-
"Busting coronavirus myths" AFP Fact Check 2020-02-19
## FAQs & Idées reçues
### FAQs
Nous savons que beaucoup de gens ont des questions sur COVID-19.
Nous avons mis en place un guide pour essayer de répondre à certaines des questions les plus fréquemment posées [ici](https://nextstrain.org/help/coronavirus/FAQ):
<div>
<a href="https://nextstrain.org/help/coronavirus/FAQ"><img alt="image d'un point d'interrogation" width="100" src="http://data.nextstrain.org/img_question-mark.jpg"/> COVID-19 FAQ (en anglais) </a>
</div>
### Idées reçues
Un certain nombre d'idées fausses ont été diffusées sur les origines du nouveau coronavirus.
Lors d'épidémies comme celle-ci, la diffusion d'informations connues pour être incorrectes peut conduire à davantage de panique et amener les gens à ne pas faire confiance aux scientifiques et aux gouvernements, ce qui signifie qu'ils sont moins susceptibles de suivre les avis et de prendre les précautions appropriées.
Afin d'essayer d'expliquer pourquoi ces vues sont incorrectes, les scientifiques ont abordé ces théories dans les pages ci-dessous (en anglais):
<div>
<a href="http://virological.org/t/ncovs-relationship-to-bat-coronaviruses-recombination-signals-no-snakes-no-evidence-the-2019-ncov-lineage-is-recombinant/331"><img alt="photo d'un serpent" width="100" src="http://data.nextstrain.org/img_snake-freeToUse.jpg"/> 'Snake' Origins of SARS-CoV-2 (Technical) </a>
<a href="https://twitter.com/trvrb/status/1223666856923291648"><img alt="illustration du VIH" width="100" src="http://data.nextstrain.org/img_HIV-wiki.jpg"/> 'HIV Engineering' Idea (Twitter thread)</a>
</div>
Nous présentons ici une phylogénie de 169 souches de SARS-CoV-2 (le virus responsable de COVID-19) qui ont été partagées publiquement. Des informations sur la façon dont l'analyse a été effectuée sont disponibles dans ce répertoire GitHub.
Les couleurs représentent la région d'isolement à l'intérieur d’un pays ou d’un état des États-Unis, avec l'axe des x représentant la date d'échantillonnage.
L'axe des y montre comment les séquences sont connectées et n'a aucune unité de mesure.
Les dates d'échantillonnage sont utiles, mais elles ne montrent pas toujours exactement comment deux séquences sont génétiquement liées - deux séquences qui sont identiques peuvent avoir des dates d'échantillonnage différentes et peuvent avoir l'air très éloignées dans cette visualisation.
Il est possible de changer la représentation graphique pour que l'axe des 'x' représente la divergence nucléotidique.
Vous pouvez remarquer combien de séquences qui avaient l'air différentes avant sont maintenant sur une ligne verticale. En faisant défiler la diapo précédente et celle-ci, vous pouvez observer comment l'arbre change.
La divergence correspond au nombre de changements (mutations) dans le génome. Certaines séquences peuvent ne pas avoir de mutations, ce qui veut dire qu'elles sont toutes identiques à la racine (centre) de l'arbre. Certains virus peuvent avoir entre une et onze mutations.
Le séquençage du génome d'un nouveau et grand virus à ARN dans une situation d'épidémie est difficile. Certaines des différences observées dans ces séquences peuvent être des erreurs de séquençage plutôt que des mutations réelles. Les insertions, les suppressions et les différences aux extrémités du génome sont plus susceptibles d'être des erreurs et nous les avons donc masquées pour cette analyse.
Nous représenterons l'arbre en utilisant parfois le temps sur l'axe de 'x' ou parfois la divergence nucléotidique, selon ce que nous cherchons à mettre en lumière.
Nous possédons actuellement des séquences d'échantillons prélevés sur cinq continents différents. Alors que les premiers cas étaient tous directement liés à des cas à Wuhan, associés à l'épidémie du marché de fruits de mer, nous observons maintenant divers cas différents qui montrent des preuves de propagation communautaire ou qui ont été importés de sources extérieures à la Chine.
En général, des introductions répétées à partir d'un réservoir animal montreront une diversité significative (comme pour Lassa, Ebola, MERS-CoV et la grippe aviaire). L'observation d'une telle forte concentration d'infections humaines peut s'expliquer par une épidémie qui découle d'un seul événement d'introduction zoonotique dans la population humaine suivi d'une propagation d'épidémie interhumaine.
Nous avons actuellement 3 séquences en provenance d'Italie, dont deux de la région de Rome et une de Lombardie, dans le nord de l'Italie.
Ces trois séquences ont un ancêtre commun au début de l'épidémie (près de la base de l'arbre, à gauche), ce qui suggère fortement qu'il y a eu au moins deux introductions séparées en Italie avec ensuite avec de la diffusion dans la communauté.
L'article du Dr Nuno Faria et coll. donne une excellente explication ici (en anglais) de comment les séquences brésiliennes et d'autres séquences observées au niveau mondial montrent que "l'épidémie en Italie du Nord est probablement le résultat de plusieurs introductions dans la région et non d'une source unique".
Les deux séquences de Rome (29 janvier 2020) sont directement connectées et ont toutes les deux un historique de voyage en Chine.
Nous avons basculé sur la visualisation avec la divergence pour montrer que les deux séquences italiennes sont identiques alors que les autres séquences les plus proches (virus isolés en Angleterre, au Brésil, en Suisse, aux États-Unis et en <chine) sont séparées des séquences italiennes par 2-4 mutations.
Il faut noter cependant que la séquence brésilienne (SPBR-02) a un historique de voyage à Milan en Lombardie et il semble que la séquence suisse dans ce regroupement a aussi voyagé en Italie. Nous ne connaissons pas du tout l’historique de voyage de l’échantillon des États-Unis. L’isolat England/09c est un cas direct d’importation de Chine.
L’échantillon d’Angleterre (venant de Chine) se trouve entre les échantillons italiens isolés précédemment à Rome et les cas avec un historique de voyage connu (Suisse, Brésil) ou suspecté (États-Unis) en Italie. Cela veut dire que nous ne devrions pas faire l’hypothèse que les séquences italiennes précédentes et les nouvelles séquences associées à l’Italie sont directement liées. Les échantillons plus récents pourraient provenir d’une introduction indépendante en Italie.
La séquence provenant de Lombardie (Italy/CDG1/2020) se groupe avec des séquences qui ont des historiques de voyage en Italie connus et qui y ont tres probablement été infectés: les séquences du Mexique, de l'Allemagne, du Brésil et de la Finlande.
La séquence d'Allemagne « BavPat1 » fait partie d’une introduction provenant de la Chine bien plus tôt au cours de l’épidémie. Sa similarité avec les autres séquences du regroupement (elles ne sont séparées que par une seule mutation) pourrait indiquer de la transmission non détectée (« cryptique ») en Europe venant de ce cluster allemand plus ancien.
Cela pourrait aussi être le résultat de deux introductions séparées en Europe: une séquence pas encore échantillonnée pourrait se situer entre « bavPat1' et le reste du cluster. Pour l’instant, nous ne pouvons pas savoir avec certitude quel scénario est correct.
Il y a maintenant plusieurs cas de COVID-19 signalés dans la région autour de Seattle et aux États-Unis en général. Les nouveaux cas isolés et séquencés sont génétiquement très proches d'un cas isolé à la mi-janvier dans la même zone.
Il y a deux explications possibles à cela. Le virus aurait pu être introduit au moins deux fois dans la région de Seattle à partir d'une source commune en Chine. Cependant, une autre explication est que le virus a circulé sans être détecté dans la région pendant un certain temps.
Trevor Bedford (co-fondateur de Nextstrain) a écrit un excellent article de blog sur ces possibilités, que vous pouvez lire ici (en anglais).
Les autres séquences récentes de Washington nous disent autre chose: ces séquences de la région de Seattle se groupent ensemble. Cela suggère fortement une propagation intracommunautaire et une circulation du virus SRAS-CoV-2 dans la région depuis un certain temps.
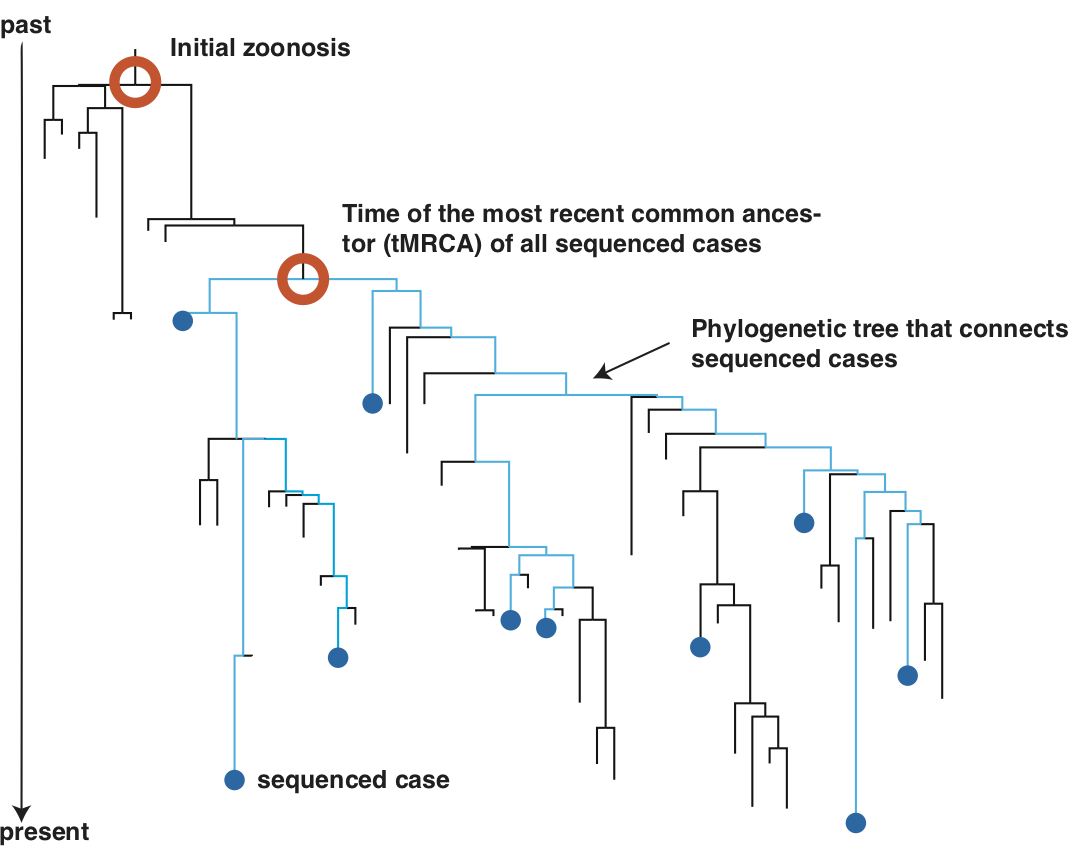
La date de l'ancêtre commun le plus récent d'un ensemble de cas séquencés correspond au dernier moment où ces cas ont partagé un ancêtre commun. Cette date peut correspondre au moment où un virus est entré pour la première fois dans la population humaine, mais elle peut aussi être bien plus tardive que la date d'introduction comme cela est montré dans la figure ci-dessous.
## Datation de l'ancêtre commun des virus épidémiques
Plusieurs groupes de recherche et personnes ont estimé une datation de l'ancêtre commun le plus récent - voir [cet article de A. Rambaut (en anglais)](http://virological.org/t/phylodynamic-analysis-of-sars-cov-2-update-2020-03-06/420) or [cet article de T. Stadler (en anglais)](http://virological.org/t/evolutionary-epidemiological-analysis-of-93-genomes).
L'ancêtre commun de toutes les séquences se situe probablement entre la mi-novembre et la mi-décembre.
Cela serait cohérent avec tous les cas actuellement séquencés issus du [groupe initial de cas sur un marché de fruits de mer de Wuhan](http://virological.org/t/phylodynamic-analysis-of-sars-cov-2-update-2020-03-06/420).
<div>
<img alt="estimation de la DAC en utilisant la phylogénétique bayésienne" width="500" src="https://raw.githubusercontent.com/nicfel/nCov-Nicola/master/figures/beast_coal-tmrca_2020303.png"/>
</div>
Nous tenons à souligner le superbe travail accompli si rapidement par tous les scientifiques impliqués dans cette épidémie, et en particulier ceux qui travaillent en Chine. Ce n'est que par le partage rapide des données génomiques et des métadonnées que de telles analyses sont possibles.
Les génomes nCoV ont été généreusement partagés par les scientifiques de:
* Centre for Infectious Diseases and Microbiology Laboratory Services
* Pathology Queensland
* Monash Medical Centre
* National Institute for Viral Disease Control and Prevention, China CDC
* KU Leuven, Clinical and Epidemiological Virology
* Hospital Israelita Albert Einstein
* Virology Unit, Institut Pasteur du Cambodge.
* BCCDC Public Health Laboratory
* Yongchuan District Center for Disease Control and Prevention
* Zhongxian Center for Disease Control and Prevention
* Respiratory Virus Unit, Microbiology Services Colindale, Public Health England
* Lapland Central Hospital
* HUS Diagnostiikkakeskus, Hallinto
* Guangdong Provincial Center for Diseases Control and Prevention; Guangdong Provincial Public Health
* Department of Infectious and Tropical Diseases, Bichat Claude Bernard Hospital, Paris
* Sorbonne Universite, Inserm et Assistance Publique-Hopitaux de Paris (Pitie Salpetriere)
* CNR Virus des Infections Respiratoires - France SUD
* Fujian Center for Disease Control and Prevention
* State Health Office Baden-Wuerttemberg
* Charite Universitatsmedizin Berlin, Institute of Virology; Institut fur Mikrobiologie der Bundeswehr, Munich
* Guangdong Provincial Center for Diseases Control and Prevention; Guangdong Provinical Public Health
* Guangdong Provincial Center for Diseases Control and Prevention;Guangdong Provincial Institute of Public Health
* Hangzhou Center for Disease and Control Microbiology Lab
* Hangzhou Center for Disease Control and Prevention
* Second Hospital of Anhui Medical University
* Hong Kong Department of Health
* Department of Infectious Diseases, Istituto Superiore di Sanita, Roma , Italy
* INMI Lazzaro Spallanzani IRCCS
* Department of Infectious Diseases, Istituto Superiore di Sanita, Rome, Italy
* Department of Virology III, National Institute of Infectious Diseases
* Dept. of Virology III, National Institute of Infectious Diseases
* Dept. of Pathology, National Institute of Infectious Diseases
* NHC Key laboratory of Enteric Pathogenic Microbiology, Institute of Pathogenic Microbiology
* Jingzhou Center for Disease Control and Prevention
* Division of Viral Diseases, Center for Laboratory Control of Infectious Diseases, Korea Centers for Diseases Control and Prevention
* Instituto Nacional de Enfermedades Respiratorias
* National Influenza Centre, National Public Health Laboratory, Kathmandu, Nepal
* Bamrasnaradura Hospital
* The University of Hong Kong - Shenzhen Hospital
* Shenzhen Third People's Hospital
* Shenzhen Key Laboratory of Pathogen and Immunity, National Clinical Research Center for Infectious Disease, Shenzhen Third People's Hospital
* Singapore General Hospital
* National Public Health Laboratory, National Centre for Infectious Diseases
* National Public Health Laboratory
* National Centre for Infectious Diseases
* Singapore General Hospital, Molecular Laboratory, Division of Pathology
* Korea Centers for Disease Control & Prevention (KCDC) Center for Laboratory Control of Infectious Diseases Division of Viral Diseases
* Serology, Virology and OTDS Laboratories (SAViD), NSW Health Pathology Randwick
* Centers for Disease Control, R.O.C. (Taiwan)
* Taiwan Centers for Disease Control
* Laboratory Medicine
* Department of Laboratory Medicine, National Taiwan University Hospital
* Tianmen Center for Disease Control and Prevention
* Arizona Department of Health Services
* California Department of Public Health
* California Department of Health
* IL Department of Public Health Chicago Laboratory
* Massachusetts Department of Public Health
* Texas Department of State Health Services
* WA State Department of Health
* Washington State Department of Health
* Providence Regional Medical Center
* Wisconsin Department of Health Services
* National Influenza Center - National Institute of Hygiene and Epidemiology (NIHE)
* Wuhan Jinyintan Hospital
* The Central Hospital Of Wuhan
* Union Hospital of Tongji Medical College, Huazhong University of Science and Technology
* CR & Wisco General Hospital
* Wuhan Lung Hospital
* Institute of Pathogen Biology, Chinese Academy of Medical Sciences & Peking Union Medical College
* Institute of Viral Disease Control and Prevention, China CDC
* General Hospital of Central Theater Command of People's Liberation Army of China
* Wuhan Fourth Hospital
* Zhejiang Provincial Center for Disease Control and Prevention
* Wuhan Institute of Virology, Chinese Academy of Sciences
* Shandong First Medical University & Shandong Academy of Medical Sciences
* South China Agricultural University
* Beijing Institute of Microbiology and Epidemiology
Ces données ont été partagées par GISAID. Nous remercions chaleureusement leurs contributions.
Sur la droite nous indiquons les séquences partagées par chaque laboratoire.
Les génomes du SRAS-CoV-2 ont été généreusement partagés par les scientifiques de ces laboratoires:
* NSW Health Pathology - Institute of Clinical Pathology and Medical Research; Westmead Hospital; University of Sydney
* Australia/NSW01/2020
* Australia/NSW05/2020
* Sydney/2/2020
* Public Health Virology Laboratory
* Australia/QLD01/2020
* Australia/QLD02/2020
* Australia/QLD03/2020
* Australia/QLD04/2020
* Collaboration between the University of Melbourne at The Peter Doherty Institute for Infection and Immunity, and the Victorian Infectious Disease Reference Laboratory
* Australia/VIC01/2020
* National Institute for Viral Disease Control & Prevention, CCDC
* Beijing/IVDC-BJ-005/2020
* Chongqing/IVDC-CQ-001/2020
* Jiangsu/IVDC-JS-001/2020
* Jiangxi/IVDC-JX-002/2020
* Shandong/IVDC-SD-001/2020
* Shanghai/IVDC-SH-001/2020
* Sichuan/IVDC-SC-001/2020
* Yunnan/IVDC-YN-003/2020
* KU Leuven, Clinical and Epidemiological Virology
* Belgium/GHB-03021/2020
* Instituto Adolfo Lutz Interdisciplinary Procedures Center Strategic Laboratory
* Brazil/SPBR-01/2020
* Virology Unit, Institut Pasteur du Cambodge (Sequencing done by: Jessica E Manning/Jennifer A Bohl at Malaria and Vector Research Research Laboratory, National Institute of Allergy and Infectious Diseases and Vida Ahyong from Chan-Zuckerberg Biohub)
* Cambodia/0012/2020
* BCCDC Public Health Laboratory
* Canada/BC_37_0-2/2020
* Technology Centre, Guangzhou Customs
* China/IQTC01/2020
* China/IQTC02/2020
* Key Laboratory of Human Diseases, Comparative Medicine, Institute of Laboratory Animal Science
* China/WH-09/2020
* State Key Laboratory of Virology, Wuhan University
* China/WHU01/2020
* China/WHU02/2020
* Chongqing Municipal Center for Disease Control and Prevention
* Chongqing/YC01/2020
* Chongqing/ZX01/2020
* Respiratory Virus Unit, Microbiology Services Colindale, Public Health England
* England/01/2020
* England/02/2020
* England/09c/2020
* Department of Virology, University of Helsinki and Helsinki University Hospital, Helsinki, Finland
* Finland/1/2020
* Department of Virology Faculty of Medicine, Medicum University of Helsinki
* Finland/FIN-25/2020
* Guangdong Provincial Center for Diseases Control and Prevention
* Foshan/20SF207/2020
* Foshan/20SF210/2020
* Foshan/20SF211/2020
* Guangdong/20SF201/2020
* Guangzhou/20SF206/2020
* National Reference Center for Viruses of Respiratory Infections, Institut Pasteur, Paris
* France/IDF0372-isl/2020
* France/IDF0372/2020
* France/IDF0373/2020
* France/IDF0515-isl/2020
* France/IDF0515/2020
* France/IDF0626/2020
* Laboratoire Virpath, CIRI U111, UCBL1, INSERM, CNRS, ENS Lyon
* France/IDF0386-islP1/2020
* France/IDF0386-islP3/2020
* France/IDF0571/2020
* CNR Virus des Infections Respiratoires - France SUD
* France/RA739/2020
* Fujian Center for Disease Control and Prevention
* Fujian/13/2020
* Fujian/8/2020
* Charite Universitatsmedizin Berlin, Institute of Virology
* Germany/Baden-Wuerttemberg-1/2020
* Germany/BavPat1/2020
* Department of Microbiology, Guangdong Provincial Center for Diseases Control and Prevention
* Guangdong/20SF012/2020
* Guangdong/20SF013/2020
* Guangdong/20SF014/2020
* Guangdong/20SF025/2020
* Guangdong/20SF028/2020
* Guangdong/20SF040/2020
* Guangdong Provincial Center for Disease Control and Prevention
* Guangdong/20SF174/2020
* Hangzhou Center for Disease and Control Microbiology Lab
* Hangzhou/HZ-1/2020
* Hangzhou Center for Disease Control and Prevention
* Hangzhou/HZCDC0001/2020
* Second Hospital of Anhui Medical University
* Hefei/2/2020
* National Institute for Viral Disease Control & Prevention, China CDC
* Henan/IVDC-HeN-002/2020
* School of Public Health, The University of Hon g Kong
* HongKong/VB20026565/2020
* HongKong/VM20001061/2020
* The University of Hong Kong
* HongKong/VM20001988/2020
* Nepal/61/2020
* Virology Laboratory, Scientific Department, Army Medical Center
* Italy/CDG1/2020
* Italy/SPL1/2020
* Laboratory of Virology, INMI Lazzaro Spallanzani IRCCS
* Italy/INMI1-cs/2020
* Italy/INMI1-isl/2020
* Pathogen Genomics Center, National Institute of Infectious Diseases
* Japan/AI/I-004/2020
* Japan/KY-V-029/2020
* Japan/NA-20-05-1/2020
* Japan/OS-20-07-1/2020
* Japan/TY-WK-012/2020
* Japan/TY-WK-501/2020
* Japan/TY-WK-521/2020
* Takayuki Hishiki Kanagawa Prefectural Institute of Public Health, Department of Microbiology
* Japan/Hu_DP_Kng_19-020/2020
* Japan/Hu_DP_Kng_19-027/2020
* Jiangsu Provincial Center for Disease Control & Prevention
* Jiangsu/JS01/2020
* Jiangsu/JS02/2020
* Jiangsu/JS03/2020
* Hubei Provincial Center for Disease Control and Prevention
* Jingzhou/HBCDC-HB-01/2020
* Tianmen/HBCDC-HB-07/2020
* Wuhan/HBCDC-HB-01/2019
* Wuhan/HBCDC-HB-02/2019
* Wuhan/HBCDC-HB-02/2020
* Wuhan/HBCDC-HB-03/2019
* Wuhan/HBCDC-HB-03/2020
* Wuhan/HBCDC-HB-04/2019
* Wuhan/HBCDC-HB-04/2020
* Wuhan/HBCDC-HB-05/2020
* Wuhan/HBCDC-HB-06/2020
* Division of Viral Diseases, Center for Laboratory Control of Infectious Diseases, Korea Centers for Diseases Control and Prevention
* Korea/KCDC05/2020
* Korea/KCDC06/2020
* Korea/KCDC07/2020
* Korea/KCDC12/2020
* Korea/KCDC24/2020
* Instituto de Diagnostico y Referencia Epidemiologicos (INDRE)
* Mexico/CDMX/InDRE_01/2020
* 1. Department of Medical Sciences, Ministry of Public Health, Thailand 2. Thai Red Cross Emerging Infectious Diseases - Health Science Centre 3. Department of Disease Control, Ministry of Public Health, Thailand
* Nonthaburi/61/2020
* Nonthaburi/74/2020
* Li Ka Shing Faculty of Medicine, The University of Hong Kong
* Shenzhen/HKU-SZ-002/2020
* Shenzhen/HKU-SZ-005/2020
* Shenzhen Key Laboratory of Pathogen and Immunity, National Clinical Research Center for Infectious Disease, Shenzhen Third People's Hospital
* Shenzhen/SZTH-001/2020
* Shenzhen/SZTH-002/2020
* Shenzhen/SZTH-003/2020
* Shenzhen/SZTH-004/2020
* National Public Health Laboratory
* Singapore/1/2020
* Singapore/11/2020
* National Centre for Infectious Diseases, National Centre for Infectious Diseases
* Singapore/10/2020
* Programme in Emerging Infectious Diseases, Duke-NUS Medical School
* Singapore/2/2020
* Singapore/3/2020
* Singapore/4/2020
* Singapore/5/2020
* Singapore/6/2020
* National Public Health Laboratory, National Centre for Infectious Diseases
* Singapore/7/2020
* Singapore/8/2020
* Singapore/9/2020
* Korea Centers for Disease Control & Prevention (KCDC) Center for Laboratory Control of Infectious Diseases Division of Viral Diseases
* SouthKorea/KCDC03/2020
* Department of Clinical Diagnostics
* SouthKorea/SNU01/2020
* Unit for Laboratory Development and Technology Transfer, Public Health Agency of Sweden
* Sweden/01/2020
* NSW Health Pathology - Institute of Clinical Pathology and Medical Research; Centre for Infectious Diseases and Microbiology Laboratory Services; Westmead Hospital; University of Sydney
* Sydney/3/2020
* Centers for Disease Control, R.O.C. (Taiwan)
* Taiwan/2/2020
* Taiwan Centers for Disease Control
* Taiwan/3/2020
* Taiwan/4/2020
* Department of Laboratory Medicine, Lin-Kou Chang Gung Memorial Hospital, Taoyuan, Taiwan.
* Taiwan/CGMH-CGU-01/2020
* Microbial Genomics Core Lab, National Taiwan University Centers of Genomic and Precision Medicine
* Taiwan/NTU01/2020
* Taiwan/NTU02/2020
* Pathogen Discovery, Respiratory Viruses Branch, Division of Viral Diseases, Centers for Disease Control and Prevention
* USA/AZ1/2020
* USA/CA1/2020
* USA/CA2/2020
* USA/CA3/2020
* USA/CA4/2020
* USA/CA5/2020
* USA/CA6/2020
* USA/CA7/2020
* USA/CA8/2020
* USA/CA9/2020
* USA/IL1/2020
* USA/IL2/2020
* USA/MA1/2020
* USA/TX1/2020
* USA/WA1-A12/2020
* USA/WA1-F6/2020
* USA/WI1/2020
* Division of Viral Diseases, Centers for Disease Control and Prevention
* USA/WA1/2020
* Seattle Flu Study
* USA/WA2/2020
* National Influenza Center - National Institute of Hygiene and Epidemiology (NIHE)
* Vietnam/VR03-38142/2020
* National Institute for Communicable Disease Control and Prevention (ICDC) Chinese Center for Disease Control and Prevention (China CDC)
* Wuhan-Hu-1/2019
* Institute of Pathogen Biology, Chinese Academy of Medical Sciences & Peking Union Medical College
* Wuhan/IPBCAMS-WH-01/2019
* Wuhan/IPBCAMS-WH-02/2019
* Wuhan/IPBCAMS-WH-03/2019
* Wuhan/IPBCAMS-WH-04/2019
* Wuhan/IPBCAMS-WH-05/2020
* National Institute for Viral Disease Control and Prevention, China CDC
* Wuhan/IVDC-HB-01/2019
* Wuhan/IVDC-HB-04/2020
* Wuhan/IVDC-HB-05/2019
* Institute of Viral Disease Control and Prevention, China CDC
* Wuhan/IVDC-HB-envF13-20/2020
* Wuhan/IVDC-HB-envF13-21/2020
* Wuhan/IVDC-HB-envF13/2020
* Wuhan/IVDC-HB-envF54/2020
* BGI & Institute of Microbiology, Chinese Academy of Sciences & Shandong First Medical University & Shandong Academy of Medical Sciences & General Hospital of Central Theater Command of People's Liberation Army of China
* Wuhan/WH01/2019
* Wuhan/WH02/2019
* Wuhan/WH03/2020
* Wuhan/WH04/2020
* Beijing Genomics Institute (BGI)
* Wuhan/WH05/2020
* Wuhan Institute of Virology, Chinese Academy of Sciences
* Wuhan/WIV02/2019
* Wuhan/WIV04/2019
* Wuhan/WIV05/2019
* Wuhan/WIV06/2019
* Wuhan/WIV07/2019
* Department of Microbiology, Zhejiang Provincial Center for Disease Control and Prevention
* Zhejiang/WZ-01/2020
* Zhejiang/WZ-02/2020