| title | authors | authorLinks | affiliations | date | dataset | abstract |
|---|---|---|---|---|---|---|
Análisis genómico de la propagación del nCoV. Informe de la situación hasta el 2020-03-05. |
Trevor Bedford, Richard Neher, James Hadfield, Emma Hodcroft, Misja Ilcisin, Nicola Müller, Julián Villabona-Arenas |
Fred Hutch, Seattle, USA; Biozentrum, Basel, Switzerland and CMMID-LSHTM, London, UK |
2020 March 05 |
Este informe utiliza los datos genómicos del nuevo coronavirus (nCoV) compartidos públicamente en GISAID y en Genbank. Los datos se usan para estimar las tasas y los patrones de propagación de la epidemia viral. Planeamos emitir informes de la situación que se actualizaran en la medida que se produzcan y compartan nuevos datos. Este sitio web está optimizado para su visualización en navegadores web de escritorio. |
## Resumen Ejecutivo
Hemos examinados la diversidad genética de 169</tag> genomas públicos del nuevo coronavirus (nCoV) para caracterizar la propagación del virus en diferentes áreas y estimar la fecha del ancestro común más reciente.
Principales hallazgos:
* El COVID-19 ha sido introducido en Italia al menos dos veces y se ha documentado transmisión comunitaria([Enlace a esto](https://nextstrain.org/narratives/ncov/sit-rep/es/2020-03-05?n=7)).
* Esto incluye un grupo de secuencias de 6 países diferentes donde los casos parecen haber sido importados desde Italia ([Enlace a esto](https://nextstrain.org/narratives/ncov/sit-rep/es/2020-03-05?n=9)).
* Los datos de secuencias genéticas apoyan la hipótesis de una propagación no detectada (críptica) y sostenida del COVID-19 en el área metropolitana de Seattle desde mediados de enero ([Enlace a esto](https://nextstrain.org/narratives/ncov/sit-rep/es/2020-03-05?n=10)).
* Todas las secuencias incluidas en este análisis comparten un ancestro común que existió en algún momento entre mediados de noviembre y mediados de diciembre de 2019([Enlace a esto](https://nextstrain.org/narratives/ncov/sit-rep/es/2020-03-05?n=11)).
- Resumen del brote de COVID-19 en Wikipedia 2020-03-07
- Material proporcionado por los Centros para el Control y la Prevención de Enfermedades de los Estados Unidos en US CDC 2020-02-14
## Recursos de información relacionados con el COVID-19
A continuación, hemos preparado algunos recursos (actualmente en inglés) que vale la pena leer para familiarizarse con el COVID-19 y el virus que lo causa, SARS-CoV-2.
Esta información facilitará la interpretación de los datos que presentamos en esta narrativa.
<div>
<a href="https://nextstrain.org/help/coronavirus/human-CoV"><img alt="Imagen por microscopio de los coronavirus" width="100" src="https://nextstrain.org/static/ncov_narrative-76cfd610d11ef708d213a3170de9519f.png"/> Antecedentes sobre los coronavirus </a>
<a href="https://nextstrain.org/help/coronavirus/SARS-CoV-2"><img alt="Ilustración de un coronavirus" width="100" src="http://data.nextstrain.org/img_nCoV-CDC.jpg"/> Antecedentes recientes del brote de COVID-19 </a>
<a href="https://nextstrain.org/help/general/how-to-read-a-tree"><img alt="Ilustración de un árbol filogenético" width="100" src="http://data.nextstrain.org/img_toy_alignment_mini.png"/> Cómo leer filogenias</a>
</div>
## Narrativas Nextstrain
Las siguientes páginas contienen análisis realizados con [Nextstrain](https://nextstrain.org).
El texto aparecerá al desplazarse con la barra lateral izquierda y la visualización de los datos genómicos correspondiente aparecerá en el lado derecho.
La obtención (secuenciación) rápida de genomas completos de un nuevo virus de ARN es un logro muy notable.
Estos análisis han sido posibles gracias al intercambio rápido y abierto de los datos genómicos y a las interpretaciones por parte de científicos de todo el mundo (la diapositiva final incluye los autores de cada uno de los diferentes genomas).
-
"No crea las teorías de conspiración que escucha sobre el coronavirus y el VIH" artículo 2020-01-31
-
"Teorías de conspiración sin fundamento afirman que el nuevo coronavirus fue producto de la bioingeniería" artículo 2020-02-07
-
"No, el coronavirus de Wuhan no fue diseñado genéticamente para insertar trozos del VIH en él" artículo 2020-02-02
-
"Rompiendo los mitos del coronavirus" Verificación de información por la AFP 2020-02-19
## Preguntas frecuentes y conceptos erróneos
### Preguntas frecuentes
Sabemos que mucha gente tiene preguntas sobre el COVID-19.
Hemos creado una guía para tratar de responder algunas de las preguntas más frecuentes [aquí](https://nextstrain.org/help/coronavirus/FAQ):
<div>
<a href="https://nextstrain.org/help/coronavirus/FAQ"><img alt="Imagen de un signo de interrogación" width="100" src="http://data.nextstrain.org/img_question-mark.jpg"/> Preguntas frecuentes sobre el COVID-19 (en inglés) </a>
</div>
### Conceptos erróneos
Se han circulado varios conceptos erróneos sobre los orígenes del nuevo coronavirus.
Durante brotes como este, la difusión de información que se sabe que es incorrecta puede generar más pánico y hacer que las personas no confíen en los científicos y los gobiernos, lo que significa que es menos probable que sigan las advertencias y tomen las precauciones adecuadas.
En un esfuerzo por tratar de explicar por qué estos puntos de vista son incorrectos, los científicos han abordado estas teorías en las siguientes páginas (en inglés):
<div>
<a href="http://virological.org/t/ncovs-relationship-to-bat-coronaviruses-recombination-signals-no-snakes-no-evidence-the-2019-ncov-lineage-is-recombinant/331"><img alt="Foto de una serpiente" width="100" src="http://data.nextstrain.org/img_snake-freeToUse.jpg"/> Orígen a partir de 'serpientes' del SARS-CoV-2 (informe técnico) </a>
<a href="https://twitter.com/trvrb/status/1223666856923291648"><img alt="Ilustración del VIH" width="100" src="http://data.nextstrain.org/img_HIV-wiki.jpg"/> Ideas sobre la 'bioingeniería del VIH' (hilo en Twitter)</a>
</div>
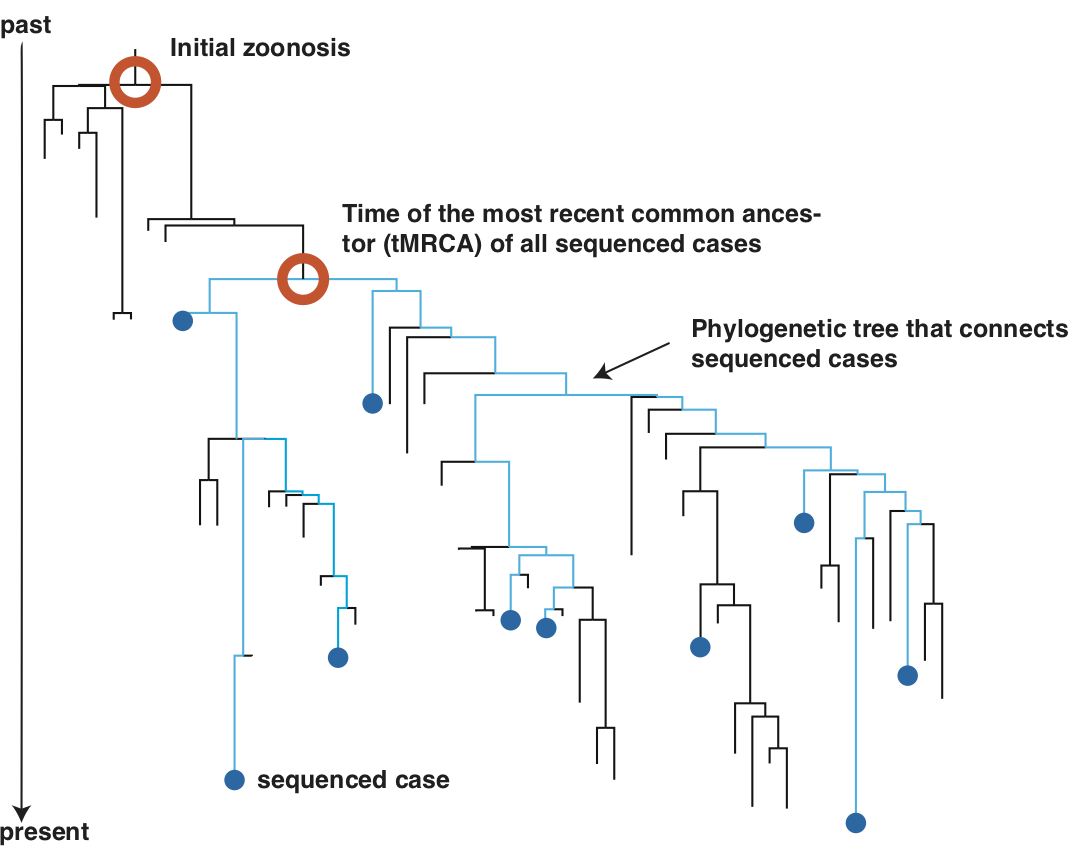
Aquí presentamos una filogenia de 169 cepas del SARS-CoV-2 (el virus que causa COVID-19) que se han compartido públicamente. La información sobre cómo se realizó el análisis está disponible en este repositorio de GitHub.
Los colores representan la región del país o el estado de los EE. UU. donde se produjo el aislamiento. El eje x representa la fecha de aislamiento de las muestras. El eje y muestra cómo están conectadas las secuencias, y no tiene ninguna unidad de medida.
Las fechas de aislamiento son útiles, pero no siempre muestran exactamente cómo se relacionan genéticamente dos secuencias: por ejemplo, dos secuencias que son idénticas podrían tener fechas de aislamiento diferentes y, por lo tanto, se ven muy separadas usando la opción actual de visualización.
Podemos cambiar la opción de visualización para que el eje x ahora muestre 'divergencia' en lugar de la fecha de aislamiento.
Presta atención a cuántas secuencias que se veían diferentes antes, ahora aparecen verticalmente en una línea. Puedes desplazarte entre la diapositiva anterior y esta, para que veas cómo cambia el árbol.
La divergencia se mide como el número de cambios (mutaciones) en el genoma. Algunas secuencias pueden tener cero mutaciones, lo que significa que todas son idénticas a la raíz (centro) del árbol. Otros virus tienen entre una y once mutaciones.
La secuenciación del genoma de un virus nuevo de ARN durante un brote es un desafío enorme. Algunas de las diferencias observadas en estas secuencias pueden ser errores de secuenciación en lugar de mutaciones reales. Las inserciones, las deleciones y las diferencias observadas en los extremos del genoma tienen una mayor probabilidad de ser errores, por lo que los filtramos durante los presentes análisis.
A veces mostraremos el árbol con la opción de visualización de 'tiempo', y a veces con la opción de visualización 'divergencia', dependiendo de lo que estemos tratando de resaltar.
Actualmente tenemos secuencias de cepas colectadas en cinco continentes diferentes. Si bien todos los primeros casos estaban directamente relacionados con los casos en Wuhan, asociados con el brote del mercado de mariscos, ahora observamos varios casos que evidencian una propagación comunitaria o casos que fueron importados de lugares fuera de China.
En general, se observa una diversidad genética significativa cuando ocurren múltiples introducciones desde un reservorio animal (como ha sido el caso de Lassa, Ebola, MERS-CoV y la gripe aviar). El hecho de que ocurra una agrupación tan próxima de las infecciones humanas por el nCoV puede indicar que el brote se inició a partir de un único evento de zoonosis seguido de la propagación epidémica de persona a persona.
Actualmente tenemos 3 secuencias de Italia, dos de las cuales son del área de Roma y una de Lombardía en el norte de Italia.
Estas 3 secuencias comparten un ancestro común al comienzo de la epidemia (cerca de la base del árbol, a la izquierda), lo que sugiere que hubo al menos dos introducciones que llevaron a la propagación comunitaria en Italia.
El Dr. Nuno Faria y colaboradores hacen un excelente análisis aquí (en inglés) de cómo las secuencias brasileñas junto con otras secuencias del mundo evidencian que "el brote en el norte de Italia fue probablemente el resultado de múltiples introducciones en la región y no el resultado de una única introducción".
Las dos secuencias de Roma (29 de enero de 2020) están directamente conectadas entre sí y ambas tienen un historial de viaje a China.
Hemos cambiado a la visualización de 'divergencia' para mostrar que las dos secuencias italianas son idénticas, mientras que otras secuencias cercanas (de Inglaterra, Brasil, Suiza, EE. UU. Y China) en realidad están separadas de las secuencias italianas por 2-4 mutaciones.
Sin embargo, la secuencia brasileña (SPBR-02) tiene un historial de viajes a Milán, en Lombardía, y se cree que la secuencia suiza en este grupo también viajó recientemente a Italia. No sabemos nada sobre el historial de viajes de la muestra de EE. UU. La secuencia inglesa (England/09c) representa una importación directa de China.
La secuencia obtenida en Inglaterra (importada desde China) aparece entre las secuencias italianas que fueron colectadas más temprano (es decir, las cepas aisladas en Roma) y aquellas secuencias obtenidas de individuos con antecedentes de viaje confirmados (cepas aisladas en Brasil y Suiza) o posible (cepa aislada en los EE. UU.) a Italia. Esto significa que no debemos suponer que las secuencias italianas más antiguas y aquellas secuencias más nuevas con un vínculo a Italia están directamente relacionadas. Las secuencias más recientes podrían provenir de una segunda introducción a Italia.
La secuencia de Lombardía (Italy/CDG1/2020) se agrupa junto con secuencias obtenidas de individuos con antecedentes de viajes a Italia y que probablemente se infectaron allí: estas secuencias fueron obtenidas en México, Alemania, Brasil y Finlandia.
La secuencia alemana "BavPat1" hace parte de una introducción mucho más temprana en la epidemia y desde China. Su similitud con la otra secuencia en este grupo (están separadas por una sola mutación), podría indicar una transmisión no detectada ("críptica") en Europa a partir de este temprano grupo alemán.
También podría ser el resultado de dos introducciones distintas en Europa: una secuencia que no fue muestreada de otro lugar podría 'caer' entre 'BavPat1' y el resto del grupo. Por el momento, no podemos decir con certeza cuál escenario es el correcto.
Ahora hay varios casos de COVID-19 reportados en el área metropolitana de Seattle y los Estados Unidos en general. Los casos recientemente aislados y secuenciados están genéticamente relacionados con un caso aislado a mediados de enero en la misma área.
Hay dos posibles explicaciones para esto. El virus podría haberse introducido al menos dos veces en el área metropolitana de Seattle desde una fuente común en China. Sin embargo, otra explicación es que el virus estuvo circulando sin ser detectado en el área por un tiempo.
Trevor Bedford (cofundador de Nextstrain) ha escrito una gran publicación en su blog sobre estas posibilidades, que puedes leer aquí (en inglés).
Las secuencias restantes recientes de Washington nos muestran algo más: las secuencias del área metropolitana de Seattle se agrupan juntas. Esto sugiere propagación comunitaria, y que el virus SARS-CoV-2 ha estado circulando en el área desde hace algún tiempo.
El tiempo del ancestro común más reciente (o tMRCA por sus siglas en inglés) de un conjunto de secuencias nos indica cuándo estos casos compartieron un ancestro común por última vez. Esta fecha puede ser tan temprano como el momento en que el virus ingresó por primera vez a la población humana, pero también puede ser sustancialmente más tarde, como se muestra en la figura a continuación.
## Fecha del ancestro común de los virus del brote
Varios grupos de investigación y personas han estimado el tiempo del antepasado común más reciente - ver [este post de A Rambaut (en inglés)](http://virological.org/t/phylodynamic-analysis-of-sars-cov-2-update-2020-03-06/420) o [esta publicación de T Stadler (en inglés)](http://virological.org/t/phylodynamic-analysis-of-sars-cov-2-update-2020-03-06/420).
El antepasado común de todas las secuencias existió probablemente entre mediados de noviembre y mediados de diciembre.
Esto sería consistente con todos los casos secuenciados actualmente que descienden del [grupo inicial de casos en el mercado de mariscos de Wuhan](http://virological.org/t/phylodynamic-analysis-129-genomes-24-feb-2020).
<div>
<img alt="estimación de tMRCA utilizando filogenética bayesiana" width="500" src="https://raw.githubusercontent.com/nicfel/nCov-Nicola/master/figures/beast_coal-tmrca_2020303.png"/>
</div>
Nos gustaría reconocer el trabajo increíble y oportuno realizado por todos los científicos involucrados en este brote, pero particularmente aquellos que trabajan en China. Solo mediante el intercambio rápido de datos genómicos y metadatos se pueden realizar análisis como estos.
También agradecemos a GISAID por proporcionar la plataforma a través de la cual estos datos se pueden depositar y compartir.
Agradecemos los datos generados por estos laboratorios:
* Centre for Infectious Diseases and Microbiology Laboratory Services
* Pathology Queensland
* Monash Medical Centre
* National Institute for Viral Disease Control and Prevention, China CDC
* KU Leuven, Clinical and Epidemiological Virology
* Hospital Israelita Albert Einstein
* Virology Unit, Institut Pasteur du Cambodge.
* BCCDC Public Health Laboratory
* Yongchuan District Center for Disease Control and Prevention
* Zhongxian Center for Disease Control and Prevention
* Respiratory Virus Unit, Microbiology Services Colindale, Public Health England
* Lapland Central Hospital
* HUS Diagnostiikkakeskus, Hallinto
* Guangdong Provincial Center for Diseases Control and Prevention; Guangdong Provincial Public Health
* Department of Infectious and Tropical Diseases, Bichat Claude Bernard Hospital, Paris
* Sorbonne Universite, Inserm et Assistance Publique-Hopitaux de Paris (Pitie Salpetriere)
* CNR Virus des Infections Respiratoires - France SUD
* Fujian Center for Disease Control and Prevention
* State Health Office Baden-Wuerttemberg
* Charite Universitatsmedizin Berlin, Institute of Virology; Institut fur Mikrobiologie der Bundeswehr, Munich
* Guangdong Provincial Center for Diseases Control and Prevention; Guangdong Provinical Public Health
* Guangdong Provincial Center for Diseases Control and Prevention;Guangdong Provincial Institute of Public Health
* Hangzhou Center for Disease and Control Microbiology Lab
* Hangzhou Center for Disease Control and Prevention
* Second Hospital of Anhui Medical University
* Hong Kong Department of Health
* Department of Infectious Diseases, Istituto Superiore di Sanita, Roma , Italy
* INMI Lazzaro Spallanzani IRCCS
* Department of Infectious Diseases, Istituto Superiore di Sanita, Rome, Italy
* Department of Virology III, National Institute of Infectious Diseases
* Dept. of Virology III, National Institute of Infectious Diseases
* Dept. of Pathology, National Institute of Infectious Diseases
* NHC Key laboratory of Enteric Pathogenic Microbiology, Institute of Pathogenic Microbiology
* Jingzhou Center for Disease Control and Prevention
* Division of Viral Diseases, Center for Laboratory Control of Infectious Diseases, Korea Centers for Diseases Control and Prevention
* Instituto Nacional de Enfermedades Respiratorias
* National Influenza Centre, National Public Health Laboratory, Kathmandu, Nepal
* Bamrasnaradura Hospital
* The University of Hong Kong - Shenzhen Hospital
* Shenzhen Third People's Hospital
* Shenzhen Key Laboratory of Pathogen and Immunity, National Clinical Research Center for Infectious Disease, Shenzhen Third People's Hospital
* Singapore General Hospital
* National Public Health Laboratory, National Centre for Infectious Diseases
* National Public Health Laboratory
* National Centre for Infectious Diseases
* Singapore General Hospital, Molecular Laboratory, Division of Pathology
* Korea Centers for Disease Control & Prevention (KCDC) Center for Laboratory Control of Infectious Diseases Division of Viral Diseases
* Serology, Virology and OTDS Laboratories (SAViD), NSW Health Pathology Randwick
* Centers for Disease Control, R.O.C. (Taiwan)
* Taiwan Centers for Disease Control
* Laboratory Medicine
* Department of Laboratory Medicine, National Taiwan University Hospital
* Tianmen Center for Disease Control and Prevention
* Arizona Department of Health Services
* California Department of Public Health
* California Department of Health
* IL Department of Public Health Chicago Laboratory
* Massachusetts Department of Public Health
* Texas Department of State Health Services
* WA State Department of Health
* Washington State Department of Health
* Providence Regional Medical Center
* Wisconsin Department of Health Services
* National Influenza Center - National Institute of Hygiene and Epidemiology (NIHE)
* Wuhan Jinyintan Hospital
* The Central Hospital Of Wuhan
* Union Hospital of Tongji Medical College, Huazhong University of Science and Technology
* CR & Wisco General Hospital
* Wuhan Lung Hospital
* Institute of Pathogen Biology, Chinese Academy of Medical Sciences & Peking Union Medical College
* Institute of Viral Disease Control and Prevention, China CDC
* General Hospital of Central Theater Command of People's Liberation Army of China
* Wuhan Fourth Hospital
* Zhejiang Provincial Center for Disease Control and Prevention
* Wuhan Institute of Virology, Chinese Academy of Sciences
* Shandong First Medical University & Shandong Academy of Medical Sciences
* South China Agricultural University
* Beijing Institute of Microbiology and Epidemiology
Estos datos se compartieron a través de GISAID. Agradecemos sus contribuciones.
A la derecha especifícamos las secuencias compartidas por cada laboratorio.
Los genomas del SARS-CoV-2 fueron generosamente compartidos por los científicos en estos laboratorios:
* NSW Health Pathology - Institute of Clinical Pathology and Medical Research; Westmead Hospital; University of Sydney
* Australia/NSW01/2020
* Australia/NSW05/2020
* Sydney/2/2020
* Public Health Virology Laboratory
* Australia/QLD01/2020
* Australia/QLD02/2020
* Australia/QLD03/2020
* Australia/QLD04/2020
* Collaboration between the University of Melbourne at The Peter Doherty Institute for Infection and Immunity, and the Victorian Infectious Disease Reference Laboratory
* Australia/VIC01/2020
* National Institute for Viral Disease Control & Prevention, CCDC
* Beijing/IVDC-BJ-005/2020
* Chongqing/IVDC-CQ-001/2020
* Jiangsu/IVDC-JS-001/2020
* Jiangxi/IVDC-JX-002/2020
* Shandong/IVDC-SD-001/2020
* Shanghai/IVDC-SH-001/2020
* Sichuan/IVDC-SC-001/2020
* Yunnan/IVDC-YN-003/2020
* KU Leuven, Clinical and Epidemiological Virology
* Belgium/GHB-03021/2020
* Instituto Adolfo Lutz Interdisciplinary Procedures Center Strategic Laboratory
* Brazil/SPBR-01/2020
* Virology Unit, Institut Pasteur du Cambodge (Sequencing done by: Jessica E Manning/Jennifer A Bohl at Malaria and Vector Research Research Laboratory, National Institute of Allergy and Infectious Diseases and Vida Ahyong from Chan-Zuckerberg Biohub)
* Cambodia/0012/2020
* BCCDC Public Health Laboratory
* Canada/BC_37_0-2/2020
* Technology Centre, Guangzhou Customs
* China/IQTC01/2020
* China/IQTC02/2020
* Key Laboratory of Human Diseases, Comparative Medicine, Institute of Laboratory Animal Science
* China/WH-09/2020
* State Key Laboratory of Virology, Wuhan University
* China/WHU01/2020
* China/WHU02/2020
* Chongqing Municipal Center for Disease Control and Prevention
* Chongqing/YC01/2020
* Chongqing/ZX01/2020
* Respiratory Virus Unit, Microbiology Services Colindale, Public Health England
* England/01/2020
* England/02/2020
* England/09c/2020
* Department of Virology, University of Helsinki and Helsinki University Hospital, Helsinki, Finland
* Finland/1/2020
* Department of Virology Faculty of Medicine, Medicum University of Helsinki
* Finland/FIN-25/2020
* Guangdong Provincial Center for Diseases Control and Prevention
* Foshan/20SF207/2020
* Foshan/20SF210/2020
* Foshan/20SF211/2020
* Guangdong/20SF201/2020
* Guangzhou/20SF206/2020
* National Reference Center for Viruses of Respiratory Infections, Institut Pasteur, Paris
* France/IDF0372-isl/2020
* France/IDF0372/2020
* France/IDF0373/2020
* France/IDF0515-isl/2020
* France/IDF0515/2020
* France/IDF0626/2020
* Laboratoire Virpath, CIRI U111, UCBL1, INSERM, CNRS, ENS Lyon
* France/IDF0386-islP1/2020
* France/IDF0386-islP3/2020
* France/IDF0571/2020
* CNR Virus des Infections Respiratoires - France SUD
* France/RA739/2020
* Fujian Center for Disease Control and Prevention
* Fujian/13/2020
* Fujian/8/2020
* Charite Universitatsmedizin Berlin, Institute of Virology
* Germany/Baden-Wuerttemberg-1/2020
* Germany/BavPat1/2020
* Department of Microbiology, Guangdong Provincial Center for Diseases Control and Prevention
* Guangdong/20SF012/2020
* Guangdong/20SF013/2020
* Guangdong/20SF014/2020
* Guangdong/20SF025/2020
* Guangdong/20SF028/2020
* Guangdong/20SF040/2020
* Guangdong Provincial Center for Disease Control and Prevention
* Guangdong/20SF174/2020
* Hangzhou Center for Disease and Control Microbiology Lab
* Hangzhou/HZ-1/2020
* Hangzhou Center for Disease Control and Prevention
* Hangzhou/HZCDC0001/2020
* Second Hospital of Anhui Medical University
* Hefei/2/2020
* National Institute for Viral Disease Control & Prevention, China CDC
* Henan/IVDC-HeN-002/2020
* School of Public Health, The University of Hon g Kong
* HongKong/VB20026565/2020
* HongKong/VM20001061/2020
* The University of Hong Kong
* HongKong/VM20001988/2020
* Nepal/61/2020
* Virology Laboratory, Scientific Department, Army Medical Center
* Italy/CDG1/2020
* Italy/SPL1/2020
* Laboratory of Virology, INMI Lazzaro Spallanzani IRCCS
* Italy/INMI1-cs/2020
* Italy/INMI1-isl/2020
* Pathogen Genomics Center, National Institute of Infectious Diseases
* Japan/AI/I-004/2020
* Japan/KY-V-029/2020
* Japan/NA-20-05-1/2020
* Japan/OS-20-07-1/2020
* Japan/TY-WK-012/2020
* Japan/TY-WK-501/2020
* Japan/TY-WK-521/2020
* Takayuki Hishiki Kanagawa Prefectural Institute of Public Health, Department of Microbiology
* Japan/Hu_DP_Kng_19-020/2020
* Japan/Hu_DP_Kng_19-027/2020
* Jiangsu Provincial Center for Disease Control & Prevention
* Jiangsu/JS01/2020
* Jiangsu/JS02/2020
* Jiangsu/JS03/2020
* Hubei Provincial Center for Disease Control and Prevention
* Jingzhou/HBCDC-HB-01/2020
* Tianmen/HBCDC-HB-07/2020
* Wuhan/HBCDC-HB-01/2019
* Wuhan/HBCDC-HB-02/2019
* Wuhan/HBCDC-HB-02/2020
* Wuhan/HBCDC-HB-03/2019
* Wuhan/HBCDC-HB-03/2020
* Wuhan/HBCDC-HB-04/2019
* Wuhan/HBCDC-HB-04/2020
* Wuhan/HBCDC-HB-05/2020
* Wuhan/HBCDC-HB-06/2020
* Division of Viral Diseases, Center for Laboratory Control of Infectious Diseases, Korea Centers for Diseases Control and Prevention
* Korea/KCDC05/2020
* Korea/KCDC06/2020
* Korea/KCDC07/2020
* Korea/KCDC12/2020
* Korea/KCDC24/2020
* Instituto de Diagnostico y Referencia Epidemiologicos (INDRE)
* Mexico/CDMX/InDRE_01/2020
* 1. Department of Medical Sciences, Ministry of Public Health, Thailand 2. Thai Red Cross Emerging Infectious Diseases - Health Science Centre 3. Department of Disease Control, Ministry of Public Health, Thailand
* Nonthaburi/61/2020
* Nonthaburi/74/2020
* Li Ka Shing Faculty of Medicine, The University of Hong Kong
* Shenzhen/HKU-SZ-002/2020
* Shenzhen/HKU-SZ-005/2020
* Shenzhen Key Laboratory of Pathogen and Immunity, National Clinical Research Center for Infectious Disease, Shenzhen Third People's Hospital
* Shenzhen/SZTH-001/2020
* Shenzhen/SZTH-002/2020
* Shenzhen/SZTH-003/2020
* Shenzhen/SZTH-004/2020
* National Public Health Laboratory
* Singapore/1/2020
* Singapore/11/2020
* National Centre for Infectious Diseases, National Centre for Infectious Diseases
* Singapore/10/2020
* Programme in Emerging Infectious Diseases, Duke-NUS Medical School
* Singapore/2/2020
* Singapore/3/2020
* Singapore/4/2020
* Singapore/5/2020
* Singapore/6/2020
* National Public Health Laboratory, National Centre for Infectious Diseases
* Singapore/7/2020
* Singapore/8/2020
* Singapore/9/2020
* Korea Centers for Disease Control & Prevention (KCDC) Center for Laboratory Control of Infectious Diseases Division of Viral Diseases
* SouthKorea/KCDC03/2020
* Department of Clinical Diagnostics
* SouthKorea/SNU01/2020
* Unit for Laboratory Development and Technology Transfer, Public Health Agency of Sweden
* Sweden/01/2020
* NSW Health Pathology - Institute of Clinical Pathology and Medical Research; Centre for Infectious Diseases and Microbiology Laboratory Services; Westmead Hospital; University of Sydney
* Sydney/3/2020
* Centers for Disease Control, R.O.C. (Taiwan)
* Taiwan/2/2020
* Taiwan Centers for Disease Control
* Taiwan/3/2020
* Taiwan/4/2020
* Department of Laboratory Medicine, Lin-Kou Chang Gung Memorial Hospital, Taoyuan, Taiwan.
* Taiwan/CGMH-CGU-01/2020
* Microbial Genomics Core Lab, National Taiwan University Centers of Genomic and Precision Medicine
* Taiwan/NTU01/2020
* Taiwan/NTU02/2020
* Pathogen Discovery, Respiratory Viruses Branch, Division of Viral Diseases, Centers for Disease Control and Prevention
* USA/AZ1/2020
* USA/CA1/2020
* USA/CA2/2020
* USA/CA3/2020
* USA/CA4/2020
* USA/CA5/2020
* USA/CA6/2020
* USA/CA7/2020
* USA/CA8/2020
* USA/CA9/2020
* USA/IL1/2020
* USA/IL2/2020
* USA/MA1/2020
* USA/TX1/2020
* USA/WA1-A12/2020
* USA/WA1-F6/2020
* USA/WI1/2020
* Division of Viral Diseases, Centers for Disease Control and Prevention
* USA/WA1/2020
* Seattle Flu Study
* USA/WA2/2020
* National Influenza Center - National Institute of Hygiene and Epidemiology (NIHE)
* Vietnam/VR03-38142/2020
* National Institute for Communicable Disease Control and Prevention (ICDC) Chinese Center for Disease Control and Prevention (China CDC)
* Wuhan-Hu-1/2019
* Institute of Pathogen Biology, Chinese Academy of Medical Sciences & Peking Union Medical College
* Wuhan/IPBCAMS-WH-01/2019
* Wuhan/IPBCAMS-WH-02/2019
* Wuhan/IPBCAMS-WH-03/2019
* Wuhan/IPBCAMS-WH-04/2019
* Wuhan/IPBCAMS-WH-05/2020
* National Institute for Viral Disease Control and Prevention, China CDC
* Wuhan/IVDC-HB-01/2019
* Wuhan/IVDC-HB-04/2020
* Wuhan/IVDC-HB-05/2019
* Institute of Viral Disease Control and Prevention, China CDC
* Wuhan/IVDC-HB-envF13-20/2020
* Wuhan/IVDC-HB-envF13-21/2020
* Wuhan/IVDC-HB-envF13/2020
* Wuhan/IVDC-HB-envF54/2020
* BGI & Institute of Microbiology, Chinese Academy of Sciences & Shandong First Medical University & Shandong Academy of Medical Sciences & General Hospital of Central Theater Command of People's Liberation Army of China
* Wuhan/WH01/2019
* Wuhan/WH02/2019
* Wuhan/WH03/2020
* Wuhan/WH04/2020
* Beijing Genomics Institute (BGI)
* Wuhan/WH05/2020
* Wuhan Institute of Virology, Chinese Academy of Sciences
* Wuhan/WIV02/2019
* Wuhan/WIV04/2019
* Wuhan/WIV05/2019
* Wuhan/WIV06/2019
* Wuhan/WIV07/2019
* Department of Microbiology, Zhejiang Provincial Center for Disease Control and Prevention
* Zhejiang/WZ-01/2020
* Zhejiang/WZ-02/2020