| title | authors | authorLinks | affiliations | date | dataset | abstract |
|---|---|---|---|---|---|---|
Analyse von Virus-Genomen zur Ausbreitung von nCoV. Lagebericht 2020-03-05. |
Trevor Bedford, Richard Neher, James Hadfield, Emma Hodcroft, Misja Ilcisin, Nicola Müller |
Fred Hutch, Seattle, USA and Biozentrum, Basel, Switzerland |
2020 March 05 |
Dieser Bericht verwendet öffentlich zugängliche Genome des neuartigen Coronavirus (nCoV) aus GISAID oder Genbank, um die Rate und das Muster der Verbreitung des Ausbruchs abzuschätzen. Wir planen, aktualisierte Lageberichte herauszugeben, sobald neue Daten verfügbar sind. Diese Website ist für Desktop-Browser optimiert und nur eingeschränkt auf mobilen Endgeräten nutzbar. |
## Zusammenfassung
Wir haben 169</tag> öffentlich zugängliche Genome des neuartigen Coronavirus (nCoV) untersucht, um auf das Datum des gemeinsamen Vorfahren und die Verbreitungsrate zurückzuschliessen.
Wir finden:
* COVID-19 wurde mindestens zwei mal nach Italien eingeführt und vermutlich lokal verbreitet ([siehe hier](https://nextstrain.org/narratives/ncov/sit-rep/de/2020-03-05?n=7)).
* Einer dieser lokalen Cluster beinhalted Sequenzen von 6 verschieden Ländern welche direkt aus Italien exportiert zu sein scheinen ([siehe hier](https://nextstrain.org/narratives/ncov/sit-rep/de/2020-03-05?n=9)).
* Genetische Sequenzen stützen die Hypothese, dass COVID-19 seit Mitte Januar in der Region um Seattle präsent ist. ([siehe hier](https://nextstrain.org/narratives/ncov/sit-rep/de/2020-03-05?n=10)).
* Alle Sequenzen in dieser Analyse haben einen geminsame Vorfahren irgendwann zwischen Mitte November und Mitte Dezember ([siehe hier](https://nextstrain.org/narratives/ncov/sit-rep/de/2020-03-05?n=11)).
## COVID-19 Ressourcen
Hier haben wir mehrere lesenswerte Resourcen (auf Englisch) zusammengestellt um sich mit COVID-19 (der Krankeit) um dem Virus welche sie verursacht, SARS-CoV-2, bekannt zu machen.
Diese Informationen werden es einfacher machen die nachfolgenden Daten zu verstehen.
<div>
<a href="https://nextstrain.org/help/coronavirus/human-CoV"><img alt="microscopy image of coronaviruses" width="100" src="https://nextstrain.org/static/ncov_narrative-76cfd610d11ef708d213a3170de9519f.png"/> Background on Coronaviruses </a>
<a href="https://nextstrain.org/help/coronavirus/SARS-CoV-2"><img alt="illustration of a coronavirus" width="100" src="http://data.nextstrain.org/img_nCoV-CDC.jpg"/> Recent COVID-19 Outbreak Background </a>
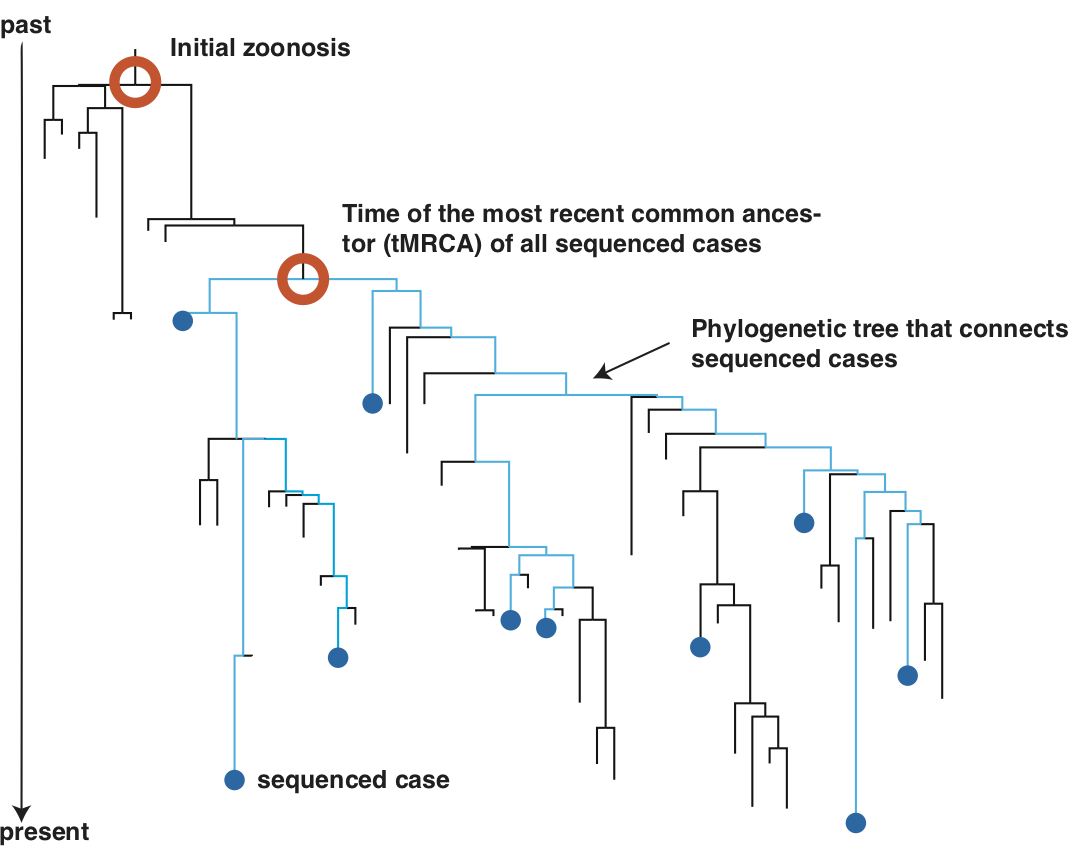
<a href="https://nextstrain.org/help/general/how-to-read-a-tree"><img alt="cartoon of a phylogenetic tree" width="100" src="http://data.nextstrain.org/img_toy_alignment_mini.png"/> How to Read Phylogenies</a>
</div>
## Nextstrain Narrative
Die folgenden Seiten enthalten Analysen, die mit [Nextstrain](https://nextstrain.org) durchgeführt wurden.
Wenn Sie durch die linke Leiste blättern, werden Textabschnitte mit einer entsprechenden Visualisierung der genomischen Daten auf der rechten Seite angezeigt.
Das vollständige Genom eines neuartigen und grossen RNA-Virus schnell zu entschlüsseln ist eine bemerkenswerte Leistung.
Diese Analysen wurden durch den schnellen und offenen Austausch von genomischen Daten und Interpretationen durch Wissenschaftler auf der ganzen Welt ermöglicht (siehe die letzte Folie mit einer Liste der beitragenden Institute).
-
"Don't believe the conspiracy theories you hear about coronavirus & HIV" article 2020-01-31
-
"Baseless Conspiracy Theories Claim New Coronavirus Was Bioengineered" article 2020-02-07
-
"No, The Wuhan Coronavirus Was Not Genetically Engineered To Put Pieces Of HIV In It" article 2020-02-02
-
"Busting coronavirus myths" AFP Fact Check 2020-02-19
## Weiterführende Informationen und Missverständnisse
### Weiterführende Informationen
Wir sind uns bewusst, dass viele Personen offene Fragen bezüglich COVID-19 haben.
Wir habe mehrere dieser zusammegetragen und [here](https://nextstrain.org/help/coronavirus/FAQ) versucht zu beantworten:
<div>
<a href="https://nextstrain.org/help/coronavirus/FAQ"><img alt="picture of a question mark" width="100" src="http://data.nextstrain.org/img_question-mark.jpg"/> COVID-19 (in English) FAQ </a>
</div>
### Missverständnisse
Momentan zirkulieren mehrere Missverständnisse über den Ursprung dieses neurtigen Coronaviruses.
Solche Falsch-Informationen führen zu verstärkter Unsicherheit und Misstrauen in die Wissenschaft und Behörden, was wiederum die Wahrscheinlichkeit verringert, dass geeignete Vorsichtsmassnahmen getroffen werden.
Wissenschaftler bemühen sich darum zu erklären, wieso diese Behauptungen nicht der Wahrheit entsprechen und wir haben ein paar davon hier (in Englisch) zusammengetragen :
<div>
<a href="http://virological.org/t/ncovs-relationship-to-bat-coronaviruses-recombination-signals-no-snakes-no-evidence-the-2019-ncov-lineage-is-recombinant/331"><img alt="picture of a snake" width="100" src="http://data.nextstrain.org/img_snake-freeToUse.jpg"/> 'Snake' Origins of SARS-CoV-2 (Technical) </a>
<a href="https://twitter.com/trvrb/status/1223666856923291648"><img alt="illustration of HIV" width="100" src="http://data.nextstrain.org/img_HIV-wiki.jpg"/> 'HIV Engineering' Idea (Twitter thread)</a>
</div>
Zur Rechten sehen Sie eine Phylogenie von 169 SARS-CoV-2-Stämmen (dem Virus welches COVID-19 verursacht), die öffentlich zugänglich sind. Informationen darüber, wie die Analyse durchgeführt wurde, finden Sie in diesem GitHub-Repository.
Die Farben repräsentieren die Region eines Landes wie zum Beispiel einem US-Staat der Isolation der Probe, wobei die x-Achse die Zeit darstellt.
Wann ein Virus isoliert wurde kann hilfreich sein, sagt aber nicht genau aus wie zwei Sequenzen miteinander verwandt sind. Zwei Sequenzen welche identisch sind und am gleichen Tag isoliert wurden können dennoch in dieser Ansicht nur entfernt miteinander verwandt sein.
Wir können die Ansicht nun so ändern, dass auf der x-Achse nun die 'Divergenz' angezeit wird.
Viele Sequenzen welche bis jetzt unterschiedlich ausgesehen haben sitzen nun auf der gleichen vertikalen Linie. Sie können nun herauf und herunter scrollen um zwischen den einzelnen Seiten hin und her zu wechseln um zu sehen, wie sich die Phylogenie verändert.
Divergenz wird in der Anzahl Änderungen (Mutationen) im Genom gemessen Gewisse Sequenzen haben unter Umständen keine Mutation, sprich sie sind alle identisch mit dem Ursprung der Phylogenie. Andere Viren wiederum haben zwischen einer und elf Mutationen.
Die Sequenzierung des Genoms eines grossen neuartigen RNA-Virus in einer Ausbruchssituation ist eine grosse Herausforderung. Einige der in diesen Sequenzen beobachteten Unterschiede können eher auf Sequenzierungsfehler als auf tatsächliche Mutationen zurückzuführen sein. Einfügungen, Deletionen und Unterschiede an den Enden des Genoms sind mit grösserer Wahrscheinlichkeit Fehler und wurden deshalb in dieser Analyse weggelassen.
Machmal werden wir die Phylogeie mit der x-Achse als Zeit und machmal als Divergenz darstellen, je nach dem was wir versuchen zu betonen.
Wir habe momentan Sequenzen von fünf verschiedenen Kontinenten Während die ersten Fälle alle direkt mit Wuhan verbunden waren, welche wiederum mit einem Ausbruch an einem Meeresfrüchte Markt verbunden sind, beobachten wir nun mehrere Fälle von lokaler Verbreitung ausserhalb von China.
Generell weisen wiederholte Einführungen von Viren aus einem tierischen Reservoir starke Diversität der genetischen Sequenzen, dies ist zum Beispiel bei Lassa, Ebola, MERS-CoV und der Vogelgrippe der Fall.
Die Beobachtung von so geringer Diversität bei den Infektionen kann durch eine sogenannte Zoonose erklärt werden, welche danach von Mensch zu Mensch verbreitet wurde.
Wir haben im Moment 3 Sequenzen aus Italien, zwei davon aus Rom und eine aus der Lombardei.
Diese 3 Sequenzen haben einen gemeinsamen Vorfahren früh in der Epidemie (in der Nähe des Ursprungs am linken Rand der Phylogenie). Dies stützt die Vermutung, dass das Virus mindestens zweimal nach Italien eingeführt wurde.
Dr Nuno Faria et al. geben eine gute Erklärung dazu (in English) wie ein brasilianische und andere weltweit gesammelte Sequenzen zeigen, dass der Ausbruch in Italien vermutlich von mehreren Quellen und nicht nur einer verursacht wurde.
Zwei Sequenzen aus Rom vom 29ten Januar 2020 sind direkt mit einander Verbunden und haben ein Reisegeschichte nach China.
Wir haben hier zur 'Divergenz' Ansicht gewechselt um zu zeigen, dass die beiden Sequenzen aus Rom identisch sind, während andere Sequenzen (aus Brasilien, der Schweiz, den USA und China) sich von den italienischen durch 2-4 Mutationen unterscheiden.
Die Sequenz aus Brasilien (SPBR-02) hat eine Reisegeschichte nach Mailand in der Lombardei und es wird angenommen, dass das gleiche auch für die Sequenz aus der Schweiz gilt. Wir haben keine genauere Informationen zur Reisegeschichte der Sequenz aus den USA. Die Sequenz England/09c wurde jedoch von China importiert nach England.
Die Sequenz aus England (importiert von China) sitzt zwischen der beiden Sequenzen aus Rom und denjenigen mit Reisegeschichte nach Mailand (Schweiz und Brasilien) und möglicher Reisegeschichte nach Italien (US Sequenz). Wir können darum nicht davon ausgehen, dass die beiden Sequenzen aus Italien mit den späteren Sequenzen assoziiert sind. Die neueren Sequenzen könnten von einer weiteren unabhängigen Einführung stammen.
Die Sequenz aus der Lombardei (Italy/CDG1/2020) ist nahe mit anderen Sequenzen verwandt, für welche wir wissen, dass sie eine Reisegeschichte nach Italien haben, sprich sie haben sich sehr wahrscheindlich dort angesteckt:
Die Sequenz aus Deutschland "BavPat1" ist Teil einer Einführung von China viel früher während der Epidemie. Die nahe Verwandschaft (sie unterscheidet sich von den anderen Sequenzen nur durch eine Mutation), könnte unter Umständen auf eine versteckte Verbreitung des Viruses in Europa hindeuten, welche eventuell auf diese Sequenz zurückgeht.
Sie könnte aber genauso gut das Resultat einer seperaten Einführung nach Europa sein. Eine bis dato unsequenzierte Probe könnte zwischen 'BavPat1' und den anderen Sequenzen fallen. Im Moment können wir zwischen den beiden Szenarien nicht unterscheiden.
Es gibt bis dato mehrere Fälle von COVID-19 in der Region um Seattle und den USA im Allgemeinen. Diese neu isolierten und sequenzierten Fälle sind genetisch eng verwandt mit einem Fall welcher Mitte Januar in der gleichen Region aufgetreten ist.
Es gibt hierführ zwei unterschiedliche Erklärungen. Einerseits könnte das Virus im Minimum zweimal in die Region von einer gemeinsamen Quelle in China eingeführt worden sein. Die andere Erklärung ist, dass das Virus bereits seit längerem in der Region zirkuliert.
Trevor Bedford (Co-Founder von Nextstrain) hat einen guten Blog post zu diesen unterschiedlichen Möglichkeiten verfasst, siehe hier (in Englisch).
Die weiteren Sequenzen aus Washington State erzählen zusätzlich eine weitere Geschichte. Diese Sequenzen sind eng miteinander verwandt, was sehr stark darauf hindeutet, dass SARS-CoV-2 bereits seit mehreren Wochen in der Region um Seattle zirkuliert.
Die Zeit des letzten geimensamen Vorfahrens (oder tMRCA vom Englischen) von Sequenzen ist die Zeit in welcher alle diese Sequenzen zuletzt einen gemeinsamen Vorfahren hatten. Dies kann die Zeit sein als ein Virus zum ersten Mal auf Menschen übertragen wurde, kann aber unter Umständen auch signifikant später sein, wie wir hier zeigen.
## Zeit des gemeinsamen Vorfahrens
Mehrere Forschungsgruppen haben die Zeit des letzten geimensame Vorfahren geschätzt, siehe zum Beispiel [dieser Eintrag von A. Rambaut (in Englisch)](http://virological.org/t/phylodynamic-analysis-of-sars-cov-2-update-2020-03-06/420) oder [dieser Eintrag von T Stadler (in Englisch)](http://virological.org/t/evolutionary-epidemiological-analysis-of-93-genomes).
Der letzte gemeinsame Vorfahre aller Sequenzen war sehr wahrscheindlich zwischen Mitte November und Mitte Dezember.
Dies wäre mit der Hypothese kompatibel, dass alle bis jetzt sequenzierten Fälle auf eine ursprüngliche Verbreitung in einem [Meeresfrüchte Markt in Wuhan zurückzuführen sind](http://virological.org/t/phylodynamic-analysis-of-sars-cov-2-update-2020-03-06/420).
Gewissheit darüber besteht aber nicht.
<div>
<img alt="estimate of the tMRCA using Bayesian phylogenetics" width="500" src="https://raw.githubusercontent.com/nicfel/nCov-Nicola/master/figures/beast_coal-tmrca_2020303.png"/>
</div>
Wir möchten uns für die beeindruckende, schnelle und transparente Arbeit aller an diesem Ausbruch beteiligten Wissenschaftler, insbesondere aber jener in China tätigen, bedanken. Nur durch den raschen Austausch von genomischen Daten und Metadaten sind solche Analysen möglich.
Wir möchten uns auch bei GISAID bedanken, dass sie eine Platform bieten um diese Daten heraufzuladen und zu teilen.
We are grateful for the data gathered by these originating labs:
* Centre for Infectious Diseases and Microbiology Laboratory Services
* Pathology Queensland
* Monash Medical Centre
* National Institute for Viral Disease Control and Prevention, China CDC
* KU Leuven, Clinical and Epidemiological Virology
* Hospital Israelita Albert Einstein
* Virology Unit, Institut Pasteur du Cambodge.
* BCCDC Public Health Laboratory
* Yongchuan District Center for Disease Control and Prevention
* Zhongxian Center for Disease Control and Prevention
* Respiratory Virus Unit, Microbiology Services Colindale, Public Health England
* Lapland Central Hospital
* HUS Diagnostiikkakeskus, Hallinto
* Guangdong Provincial Center for Diseases Control and Prevention; Guangdong Provincial Public Health
* Department of Infectious and Tropical Diseases, Bichat Claude Bernard Hospital, Paris
* Sorbonne Universite, Inserm et Assistance Publique-Hopitaux de Paris (Pitie Salpetriere)
* CNR Virus des Infections Respiratoires - France SUD
* Fujian Center for Disease Control and Prevention
* State Health Office Baden-Wuerttemberg
* Charite Universitatsmedizin Berlin, Institute of Virology; Institut fur Mikrobiologie der Bundeswehr, Munich
* Guangdong Provincial Center for Diseases Control and Prevention; Guangdong Provinical Public Health
* Guangdong Provincial Center for Diseases Control and Prevention;Guangdong Provincial Institute of Public Health
* Hangzhou Center for Disease and Control Microbiology Lab
* Hangzhou Center for Disease Control and Prevention
* Second Hospital of Anhui Medical University
* Hong Kong Department of Health
* Department of Infectious Diseases, Istituto Superiore di Sanita, Roma , Italy
* INMI Lazzaro Spallanzani IRCCS
* Department of Infectious Diseases, Istituto Superiore di Sanita, Rome, Italy
* Department of Virology III, National Institute of Infectious Diseases
* Dept. of Virology III, National Institute of Infectious Diseases
* Dept. of Pathology, National Institute of Infectious Diseases
* NHC Key laboratory of Enteric Pathogenic Microbiology, Institute of Pathogenic Microbiology
* Jingzhou Center for Disease Control and Prevention
* Division of Viral Diseases, Center for Laboratory Control of Infectious Diseases, Korea Centers for Diseases Control and Prevention
* Instituto Nacional de Enfermedades Respiratorias
* National Influenza Centre, National Public Health Laboratory, Kathmandu, Nepal
* Bamrasnaradura Hospital
* The University of Hong Kong - Shenzhen Hospital
* Shenzhen Third People's Hospital
* Shenzhen Key Laboratory of Pathogen and Immunity, National Clinical Research Center for Infectious Disease, Shenzhen Third People's Hospital
* Singapore General Hospital
* National Public Health Laboratory, National Centre for Infectious Diseases
* National Public Health Laboratory
* National Centre for Infectious Diseases
* Singapore General Hospital, Molecular Laboratory, Division of Pathology
* Korea Centers for Disease Control & Prevention (KCDC) Center for Laboratory Control of Infectious Diseases Division of Viral Diseases
* Serology, Virology and OTDS Laboratories (SAViD), NSW Health Pathology Randwick
* Centers for Disease Control, R.O.C. (Taiwan)
* Taiwan Centers for Disease Control
* Laboratory Medicine
* Department of Laboratory Medicine, National Taiwan University Hospital
* Tianmen Center for Disease Control and Prevention
* Arizona Department of Health Services
* California Department of Public Health
* California Department of Health
* IL Department of Public Health Chicago Laboratory
* Massachusetts Department of Public Health
* Texas Department of State Health Services
* WA State Department of Health
* Washington State Department of Health
* Providence Regional Medical Center
* Wisconsin Department of Health Services
* National Influenza Center - National Institute of Hygiene and Epidemiology (NIHE)
* Wuhan Jinyintan Hospital
* The Central Hospital Of Wuhan
* Union Hospital of Tongji Medical College, Huazhong University of Science and Technology
* CR & Wisco General Hospital
* Wuhan Lung Hospital
* Institute of Pathogen Biology, Chinese Academy of Medical Sciences & Peking Union Medical College
* Institute of Viral Disease Control and Prevention, China CDC
* General Hospital of Central Theater Command of People's Liberation Army of China
* Wuhan Fourth Hospital
* Zhejiang Provincial Center for Disease Control and Prevention
* Wuhan Institute of Virology, Chinese Academy of Sciences
* Shandong First Medical University & Shandong Academy of Medical Sciences
* South China Agricultural University
* Beijing Institute of Microbiology and Epidemiology
Diese Daten wurden via GISAID zur Verfügung gestellt.
Auf der rechten Seite finden Sie eine detaillierte Auflistung der Sequenzen und ihrer Urheber.
The SARS-CoV-2 genomes were generously shared by scientists at these submitting labs:
* NSW Health Pathology - Institute of Clinical Pathology and Medical Research; Westmead Hospital; University of Sydney
* Australia/NSW01/2020
* Australia/NSW05/2020
* Sydney/2/2020
* Public Health Virology Laboratory
* Australia/QLD01/2020
* Australia/QLD02/2020
* Australia/QLD03/2020
* Australia/QLD04/2020
* Collaboration between the University of Melbourne at The Peter Doherty Institute for Infection and Immunity, and the Victorian Infectious Disease Reference Laboratory
* Australia/VIC01/2020
* National Institute for Viral Disease Control & Prevention, CCDC
* Beijing/IVDC-BJ-005/2020
* Chongqing/IVDC-CQ-001/2020
* Jiangsu/IVDC-JS-001/2020
* Jiangxi/IVDC-JX-002/2020
* Shandong/IVDC-SD-001/2020
* Shanghai/IVDC-SH-001/2020
* Sichuan/IVDC-SC-001/2020
* Yunnan/IVDC-YN-003/2020
* KU Leuven, Clinical and Epidemiological Virology
* Belgium/GHB-03021/2020
* Instituto Adolfo Lutz Interdisciplinary Procedures Center Strategic Laboratory
* Brazil/SPBR-01/2020
* Virology Unit, Institut Pasteur du Cambodge (Sequencing done by: Jessica E Manning/Jennifer A Bohl at Malaria and Vector Research Research Laboratory, National Institute of Allergy and Infectious Diseases and Vida Ahyong from Chan-Zuckerberg Biohub)
* Cambodia/0012/2020
* BCCDC Public Health Laboratory
* Canada/BC_37_0-2/2020
* Technology Centre, Guangzhou Customs
* China/IQTC01/2020
* China/IQTC02/2020
* Key Laboratory of Human Diseases, Comparative Medicine, Institute of Laboratory Animal Science
* China/WH-09/2020
* State Key Laboratory of Virology, Wuhan University
* China/WHU01/2020
* China/WHU02/2020
* Chongqing Municipal Center for Disease Control and Prevention
* Chongqing/YC01/2020
* Chongqing/ZX01/2020
* Respiratory Virus Unit, Microbiology Services Colindale, Public Health England
* England/01/2020
* England/02/2020
* England/09c/2020
* Department of Virology, University of Helsinki and Helsinki University Hospital, Helsinki, Finland
* Finland/1/2020
* Department of Virology Faculty of Medicine, Medicum University of Helsinki
* Finland/FIN-25/2020
* Guangdong Provincial Center for Diseases Control and Prevention
* Foshan/20SF207/2020
* Foshan/20SF210/2020
* Foshan/20SF211/2020
* Guangdong/20SF201/2020
* Guangzhou/20SF206/2020
* National Reference Center for Viruses of Respiratory Infections, Institut Pasteur, Paris
* France/IDF0372-isl/2020
* France/IDF0372/2020
* France/IDF0373/2020
* France/IDF0515-isl/2020
* France/IDF0515/2020
* France/IDF0626/2020
* Laboratoire Virpath, CIRI U111, UCBL1, INSERM, CNRS, ENS Lyon
* France/IDF0386-islP1/2020
* France/IDF0386-islP3/2020
* France/IDF0571/2020
* CNR Virus des Infections Respiratoires - France SUD
* France/RA739/2020
* Fujian Center for Disease Control and Prevention
* Fujian/13/2020
* Fujian/8/2020
* Charite Universitatsmedizin Berlin, Institute of Virology
* Germany/Baden-Wuerttemberg-1/2020
* Germany/BavPat1/2020
* Department of Microbiology, Guangdong Provincial Center for Diseases Control and Prevention
* Guangdong/20SF012/2020
* Guangdong/20SF013/2020
* Guangdong/20SF014/2020
* Guangdong/20SF025/2020
* Guangdong/20SF028/2020
* Guangdong/20SF040/2020
* Guangdong Provincial Center for Disease Control and Prevention
* Guangdong/20SF174/2020
* Hangzhou Center for Disease and Control Microbiology Lab
* Hangzhou/HZ-1/2020
* Hangzhou Center for Disease Control and Prevention
* Hangzhou/HZCDC0001/2020
* Second Hospital of Anhui Medical University
* Hefei/2/2020
* National Institute for Viral Disease Control & Prevention, China CDC
* Henan/IVDC-HeN-002/2020
* School of Public Health, The University of Hon g Kong
* HongKong/VB20026565/2020
* HongKong/VM20001061/2020
* The University of Hong Kong
* HongKong/VM20001988/2020
* Nepal/61/2020
* Virology Laboratory, Scientific Department, Army Medical Center
* Italy/CDG1/2020
* Italy/SPL1/2020
* Laboratory of Virology, INMI Lazzaro Spallanzani IRCCS
* Italy/INMI1-cs/2020
* Italy/INMI1-isl/2020
* Pathogen Genomics Center, National Institute of Infectious Diseases
* Japan/AI/I-004/2020
* Japan/KY-V-029/2020
* Japan/NA-20-05-1/2020
* Japan/OS-20-07-1/2020
* Japan/TY-WK-012/2020
* Japan/TY-WK-501/2020
* Japan/TY-WK-521/2020
* Takayuki Hishiki Kanagawa Prefectural Institute of Public Health, Department of Microbiology
* Japan/Hu_DP_Kng_19-020/2020
* Japan/Hu_DP_Kng_19-027/2020
* Jiangsu Provincial Center for Disease Control & Prevention
* Jiangsu/JS01/2020
* Jiangsu/JS02/2020
* Jiangsu/JS03/2020
* Hubei Provincial Center for Disease Control and Prevention
* Jingzhou/HBCDC-HB-01/2020
* Tianmen/HBCDC-HB-07/2020
* Wuhan/HBCDC-HB-01/2019
* Wuhan/HBCDC-HB-02/2019
* Wuhan/HBCDC-HB-02/2020
* Wuhan/HBCDC-HB-03/2019
* Wuhan/HBCDC-HB-03/2020
* Wuhan/HBCDC-HB-04/2019
* Wuhan/HBCDC-HB-04/2020
* Wuhan/HBCDC-HB-05/2020
* Wuhan/HBCDC-HB-06/2020
* Division of Viral Diseases, Center for Laboratory Control of Infectious Diseases, Korea Centers for Diseases Control and Prevention
* Korea/KCDC05/2020
* Korea/KCDC06/2020
* Korea/KCDC07/2020
* Korea/KCDC12/2020
* Korea/KCDC24/2020
* Instituto de Diagnostico y Referencia Epidemiologicos (INDRE)
* Mexico/CDMX/InDRE_01/2020
* 1. Department of Medical Sciences, Ministry of Public Health, Thailand 2. Thai Red Cross Emerging Infectious Diseases - Health Science Centre 3. Department of Disease Control, Ministry of Public Health, Thailand
* Nonthaburi/61/2020
* Nonthaburi/74/2020
* Li Ka Shing Faculty of Medicine, The University of Hong Kong
* Shenzhen/HKU-SZ-002/2020
* Shenzhen/HKU-SZ-005/2020
* Shenzhen Key Laboratory of Pathogen and Immunity, National Clinical Research Center for Infectious Disease, Shenzhen Third People's Hospital
* Shenzhen/SZTH-001/2020
* Shenzhen/SZTH-002/2020
* Shenzhen/SZTH-003/2020
* Shenzhen/SZTH-004/2020
* National Public Health Laboratory
* Singapore/1/2020
* Singapore/11/2020
* National Centre for Infectious Diseases, National Centre for Infectious Diseases
* Singapore/10/2020
* Programme in Emerging Infectious Diseases, Duke-NUS Medical School
* Singapore/2/2020
* Singapore/3/2020
* Singapore/4/2020
* Singapore/5/2020
* Singapore/6/2020
* National Public Health Laboratory, National Centre for Infectious Diseases
* Singapore/7/2020
* Singapore/8/2020
* Singapore/9/2020
* Korea Centers for Disease Control & Prevention (KCDC) Center for Laboratory Control of Infectious Diseases Division of Viral Diseases
* SouthKorea/KCDC03/2020
* Department of Clinical Diagnostics
* SouthKorea/SNU01/2020
* Unit for Laboratory Development and Technology Transfer, Public Health Agency of Sweden
* Sweden/01/2020
* NSW Health Pathology - Institute of Clinical Pathology and Medical Research; Centre for Infectious Diseases and Microbiology Laboratory Services; Westmead Hospital; University of Sydney
* Sydney/3/2020
* Centers for Disease Control, R.O.C. (Taiwan)
* Taiwan/2/2020
* Taiwan Centers for Disease Control
* Taiwan/3/2020
* Taiwan/4/2020
* Department of Laboratory Medicine, Lin-Kou Chang Gung Memorial Hospital, Taoyuan, Taiwan.
* Taiwan/CGMH-CGU-01/2020
* Microbial Genomics Core Lab, National Taiwan University Centers of Genomic and Precision Medicine
* Taiwan/NTU01/2020
* Taiwan/NTU02/2020
* Pathogen Discovery, Respiratory Viruses Branch, Division of Viral Diseases, Centers for Disease Control and Prevention
* USA/AZ1/2020
* USA/CA1/2020
* USA/CA2/2020
* USA/CA3/2020
* USA/CA4/2020
* USA/CA5/2020
* USA/CA6/2020
* USA/CA7/2020
* USA/CA8/2020
* USA/CA9/2020
* USA/IL1/2020
* USA/IL2/2020
* USA/MA1/2020
* USA/TX1/2020
* USA/WA1-A12/2020
* USA/WA1-F6/2020
* USA/WI1/2020
* Division of Viral Diseases, Centers for Disease Control and Prevention
* USA/WA1/2020
* Seattle Flu Study
* USA/WA2/2020
* National Influenza Center - National Institute of Hygiene and Epidemiology (NIHE)
* Vietnam/VR03-38142/2020
* National Institute for Communicable Disease Control and Prevention (ICDC) Chinese Center for Disease Control and Prevention (China CDC)
* Wuhan-Hu-1/2019
* Institute of Pathogen Biology, Chinese Academy of Medical Sciences & Peking Union Medical College
* Wuhan/IPBCAMS-WH-01/2019
* Wuhan/IPBCAMS-WH-02/2019
* Wuhan/IPBCAMS-WH-03/2019
* Wuhan/IPBCAMS-WH-04/2019
* Wuhan/IPBCAMS-WH-05/2020
* National Institute for Viral Disease Control and Prevention, China CDC
* Wuhan/IVDC-HB-01/2019
* Wuhan/IVDC-HB-04/2020
* Wuhan/IVDC-HB-05/2019
* Institute of Viral Disease Control and Prevention, China CDC
* Wuhan/IVDC-HB-envF13-20/2020
* Wuhan/IVDC-HB-envF13-21/2020
* Wuhan/IVDC-HB-envF13/2020
* Wuhan/IVDC-HB-envF54/2020
* BGI & Institute of Microbiology, Chinese Academy of Sciences & Shandong First Medical University & Shandong Academy of Medical Sciences & General Hospital of Central Theater Command of People's Liberation Army of China
* Wuhan/WH01/2019
* Wuhan/WH02/2019
* Wuhan/WH03/2020
* Wuhan/WH04/2020
* Beijing Genomics Institute (BGI)
* Wuhan/WH05/2020
* Wuhan Institute of Virology, Chinese Academy of Sciences
* Wuhan/WIV02/2019
* Wuhan/WIV04/2019
* Wuhan/WIV05/2019
* Wuhan/WIV06/2019
* Wuhan/WIV07/2019
* Department of Microbiology, Zhejiang Provincial Center for Disease Control and Prevention
* Zhejiang/WZ-01/2020
* Zhejiang/WZ-02/2020