diff --git a/book/es-419/02_Software_Tools_Techniques/01_Loading_Raster_Data_from_GeoTIFF_Files.md b/book/es-419/02_Software_Tools_Techniques/01_Loading_Raster_Data_from_GeoTIFF_Files.md
new file mode 100644
index 0000000..31ecea2
--- /dev/null
+++ b/book/es-419/02_Software_Tools_Techniques/01_Loading_Raster_Data_from_GeoTIFF_Files.md
@@ -0,0 +1,343 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Carga de datos ráster desde archivos GeoTIFF
+
+
+
+Dado que la mayoría de los datos geoespaciales con los que trabajaremos en este tutorial están almacenados en archivos GeoTIFF, debemos saber cómo trabajar con esos archivos. La solución más sencilla es utilizar [rioxarray](https://corteva.github.io/rioxarray/html/index.html). Esta solución se encarga de muchos detalles complicados de forma transparente. También podemos utilizar [Rasterio](https://rasterio.readthedocs.io/en/stable) como herramienta para leer datos o metadatos de archivos GeoTIFF. Un uso adecuado de Rasterio puede marcar una gran diferencia a la hora de trabajar con archivos remotos en la nube.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import numpy as np
+import rasterio
+import rioxarray as rio
+from pathlib import Path
+
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+```
+
+***
+
+## [rioxarray](https://corteva.github.io/rioxarray/html/index.html)
+
+
+
+`rioxarray` es un paquete que _extiende_ el paquete Xarray (hablaremos al respecto más adelante). Las principales funciones de `rioxarray` que utilizaremos en este tutorial son:
+
+- `rioxarray.open_rasterio` para cargar archivos GeoTIFF directamente en estructuras Xarray `DataArray`, y
+- `xarray.DataArray.rio` para proporcionar usos útiles (por ejemplo, para especificar información CRS).
+
+Para acostumbrarnos a trabajar con archivos GeoTIFF, utilizaremos algunos ejemplos específicos en este cuaderno computacional y en otros posteriores. Más adelante explicaremos qué tipo de datos contiene el archivo, por el momento, solo queremos acostumbrarnos a cargar datos.
+
+
+
+### Carga de archivos en un DataArray
+
+
+
+Observa en primer lugar que `open_rasterio` funciona con direcciones de archivos locales y URL remotas.
+
+- Como era de esperarse, el acceso local es más rápido que el remoto.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif')
+data = rio.open_rasterio(LOCAL_PATH)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+REMOTE_URL ='https://opera-provisional-products.s3.us-west-2.amazonaws.com/DIST/DIST_HLS/WG/DIST-ALERT/McKinney_Wildfire/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif'
+data_remote = rio.open_rasterio(REMOTE_URL)
+```
+
+
+
+La siguiente operación compara elementos de un Xarray `DataArray` elemento a elemento (el uso del método `.all` es similar a lo que haríamos para comparar arrays NumPy). Por lo general, esta no es una forma recomendable de comparar matrices, series, dataframes u otras estructuras de datos grandes que contengan datos de punto flotante. Sin embargo, en este caso concreto, como las dos estructuras de datos se leyeron del mismo archivo almacenado en dos ubicaciones diferentes, la comparación elemento a elemento tiene sentido. Confirma que los datos cargados en la memoria desde dos fuentes distintas son idénticos en cada bit.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+(data_remote == data).all() # Verify that the data is identical from both sources
+```
+
+***
+
+## [rasterio](https://rasterio.readthedocs.io/en/stable)
+
+
+
+Esta sección puede omitirse si `rioxarray` funciona adecuadamente para nuestros análisis, es decir, si la carga de datos en la memoria no es prohibitiva. Cuando _no_ sea el caso, `rasterio` proporciona estrategias alternativas para explorar los archivos GeoTIFF. Es decir, el paquete `rasterio` ofrece formas de bajo nivel para cargar datos que `rioxarray` cuando sea necesario.
+
+De la [documentación de Rasterio](https://rasterio.readthedocs.io/en/stable):
+
+> Antes de Rasterio había una opción en Python para acceder a los diferentes tipos de archivos de datos ráster utilizados en el campo de los SIG: los enlaces de Python distribuidos con la [Biblioteca de Abstracción de Datos Geoespaciales](http://gdal.org/) (GDAL, por sus siglas en inglés de _Geospatial Data Abstraction Library_). Estos enlaces extienden Python, pero proporcionan poca abstracción para la Interface de programación de aplicaciones C (API C, por sus siglas en inglés de _Application Programming Interface_) de GDAL. Esto significa que los programas Python que los utilizan tienden a leerse y ejecutarse como programas de C. Por ejemplo, los enlaces a Python de GDAL obligan a los usuarios a tener cuidado con los punteros de C incorrectos, que pueden bloquear los programas. Esto es malo: entre otras consideraciones hemos elegido Python en vez de C para evitar problemas con los punteros.
+>
+> ¿Cómo sería tener una abstracción de datos geoespaciales en la biblioteca estándar de Python? ¿Una que utilizara características y modismos modernos del lenguaje Python? ¿Una que liberara a los usuarios de la preocupación por los punteros incorrectos y otras trampas de la programación en C? El objetivo de Rasterio es ser este tipo de biblioteca de datos ráster, que exprese el modelo de datos de GDAL utilizando menos clases de extensión no idiomáticas y tipos y protocolos de Python más idiomáticos, a la vez que funciona tan rápido como los enlaces de Python de GDAL.
+>
+> Alto rendimiento, menor carga cognitiva, código más limpio y transparente. Eso es Rasterio.
+
+
+
+***
+
+### Abrir archivos con rasterio.open
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Show rasterio.open works using context manager
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif')
+print(LOCAL_PATH)
+```
+
+
+
+Dada una fuente de datos (por ejemplo, un archivo GeoTIFF en el contexto actual), podemos abrir un objeto `DatasetReader` asociado utilizando `rasterio.open`. Técnicamente, debemos recordar cerrar el objeto después. Es decir, nuestro código quedaría así:
+
+```{code-cell} python
+ds = rasterio.open(LOCAL_PATH)
+# ..
+# do some computation
+# ...
+ds.close()
+```
+
+Al igual que con el manejo de archivos en Python, podemos utilizar un _administrador de contexto_ (es decir, una cláusula `with`) en su lugar.
+
+```python
+with rasterio.open(LOCAL_PATH) as ds:
+ # ...
+ # do some computation
+ # ...
+
+# more code outside the scope of the with block.
+```
+
+El conjunto de datos se cerrará automáticamente fuera del bloque `with`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ print(f'{type(ds)=}')
+ assert not ds.closed
+
+# outside the scope of the with block
+assert ds.closed
+```
+
+
+
+La principal ventaja al utilizar `rasterio.open` en vez de `rioxarray.open_rasterio` para abrir un archivo es que este último método abre el archivo y carga inmediatamente su contenido en un `DataDarray` en la memoria.
+
+Por el contrario, al utilizar `rasterio.open` se abre el archivo en su lugar y su contenido _no_ se carga inmediatamente en la memoria. Los datos del archivo _pueden_ leerse, pero esto debe hacerse explícitamente. Esto representa una gran diferencia cuando se trabaja con datos remotos. Transferir todo el contenido a través de una red de datos implica ciertos costos. Por ejemplo, si examinamos los metadatos, que suelen ser mucho más pequeños y pueden transferirse rápidamente, podemos descubrir, por ejemplo, que no está justificado mover todo un _array_ de datos a través de la red.
+
+
+
+***
+
+### Análisis de los atributos DatasetReader
+
+
+
+Cuando se abre un archivo utilizando `rasterio.open`, el objeto instanciado es de la clase `DatasetReader`. Esta clase tiene una serie de atributos y métodos de interés para nosotros:
+
+| | | |
+| --------- | ----------- | -------- |
+| `profile` | `height` | `width` |
+| `shape` | `count` | `nodata` |
+| `crs` | `transform` | `bounds` |
+| `xy` | `index` | `read` |
+
+En primer lugar, dado un `DatasetReader` `ds` asociado a una fuente de datos, el análisis de `ds.profile` devuelve cierta información de diagnóstico.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ print(f'{ds.profile=}')
+```
+
+
+
+Los atributos `ds.height`, `ds.width`, `ds.shape`, `ds.count`, `ds.nodata` y `ds.transform` se incluyen en la salida de `ds.profile`, pero también son accesibles individualmente.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ print(f'{ds.height=}')
+ print(f'{ds.width=}')
+ print(f'{ds.shape=}')
+ print(f'{ds.count=}')
+ print(f'{ds.nodata=}')
+ print(f'{ds.crs=}')
+ print(f'{ds.transform=}')
+```
+
+***
+
+### Lectura de datos en la memoria
+
+
+
+El método `ds.read` carga un _array_ del archivo de datos en la memoria. Ten en cuenta que esto se puede hacer en archivos locales o remotos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+with rasterio.open(LOCAL_PATH) as ds:
+ array = ds.read()
+ print(f'{array.shape=}')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+%%time
+with rasterio.open(REMOTE_URL) as ds:
+ array = ds.read()
+ print(f'{array.shape=}')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(f'{type(array)=}')
+```
+
+
+
+El _array_ cargado en la memoria con `ds.read` es una matriz NumPy. Este puede ser encapsulado por un Xarray `DataArray` si proporcionamos código adicional para especificar las etiquetas de las coordenadas y demás.
+
+
+
+***
+
+### Mapeo de coordenadas
+
+
+
+Anteriormente, cargamos los datos de un archivo local en un `DataArray` llamado `da` utilizando `rioxarray.open_rasterio`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+da = rio.open_rasterio(LOCAL_PATH)
+da
+```
+
+
+
+De este modo se simplificó la carga de datos ráster de un archivo GeoTIFF en un Xarray `DataArray` a la vez que cargaban los metadatos automáticamente. En particular, las coordenadas asociadas a los píxeles se almacenaron en `da.coords` (los ejes de coordenadas predeterminados son `band`, `x` y `y` para este _array_ tridimensional).
+
+Si ignoramos la dimensión extra de `band`, los píxeles de los datos ráster se asocian con coordenadas de píxel (enteros) y coordenadas espaciales (valores reales, típicamente un punto en el centro de cada píxel).
+
+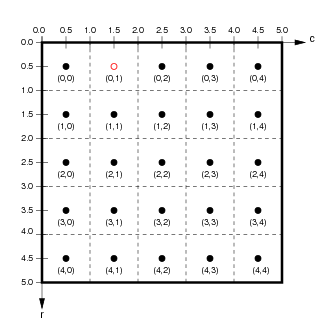
+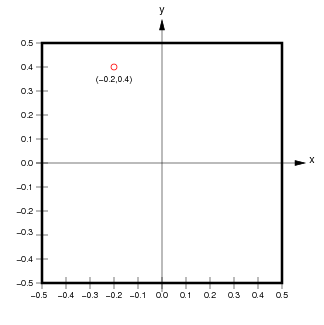
+(de `http://ioam.github.io/topographica`)
+
+Los accesores `da.isel` y `da.sel` nos permiten extraer porciones del _array_ utilizando coordenadas de píxel o coordenadas espaciales, respectivamente.
+
+
+
+
+
+Si utilizamos `rasterio.open` para abrir un archivo, el atributo `transform` de `DatasetReader` proporciona los detalles sobre cómo realizar la conversión entre coordenadas de píxel y espaciales. Utilizaremos esta propiedad en algunos de los casos prácticos más adelante.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ print(f'{ds.transform=}')
+ print(f'{np.abs(ds.transform[0])=}')
+ print(f'{np.abs(ds.transform[4])=}')
+```
+
+
+
+El atributo `ds.transform` es un objeto que describe una [_transformación afín_](https://es.wikipedia.org/wiki/Transformaci%C3%B3n_af%C3%ADn) (representada anteriormente como una matriz $2\times3$). Observa que los valores absolutos de las entradas diagonales de la matriz `ds.transform` dan las dimensiones espaciales de los píxeles ($30\mathrm{m}\times30\mathrm{m}$ en este caso).
+
+También podemos utilizar este objeto para convertir las coordenadas de los píxeles en las coordenadas espaciales correspondientes.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ print(f'{ds.transform * (0,0)=}') # top-left pixel
+ print(f'{ds.transform * (0,3660)=}') # bottom-left pixel
+ print(f'{ds.transform * (3660,0)=}') # top-right pixel
+ print(f'{ds.transform * (3660,3660)=}') # bottom-right pixel
+```
+
+
+
+El atributo `ds.bounds` muestra los límites de la región espacial (izquierda, abajo, derecha, arriba).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ print(f'coordinate bounds: {ds.bounds=}')
+```
+
+
+
+El método `ds.xy` también convierte coordenadas de índice entero en coordenadas continuas. Observa que `ds.xy` asigna enteros al centro de los píxeles. Los bucles siguientes imprimen la primera esquina superior izquierda de las coordenadas en coordenadas de píxel y, después, las coordenadas espaciales correspondientes.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ for k in range(3):
+ for l in range(4):
+ print(f'({k:2d},{l:2d})','\t', end='')
+ print()
+ print()
+ for k in range(3):
+ for l in range(4):
+ e,n = ds.xy(k,l)
+ print(f'({e},{n})','\t', end='')
+ print()
+ print()
+```
+
+
+
+`ds.index` hace lo contrario: dadas las coordenadas espaciales `(x,y)`, devuelve los índices enteros del píxel que contiene ese punto.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ print(ds.index(500000, 4700015))
+```
+
+
+
+Estas conversiones pueden ser complicadas, sobre todo porque las coordenadas de píxel corresponden a los centros de los píxeles y también porque la segunda coordenada espacial `y` _disminuye_ a medida que la segunda coordenada de píxel _aumenta_. Hacer un seguimiento de detalles tediosos como este es en parte la razón por la que resulta útil cargar desde `rioxarray`, es decir, que nosotros lo hagamos. Pero vale la pena saber que podemos reconstruir este mapeo si es necesario a partir de los metadatos en el archivo GeoTIFF (utilizaremos este hecho más adelante).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+with rasterio.open(LOCAL_PATH) as ds:
+ print(ds.bounds)
+ print(ds.transform * (0.5,0.5)) # Maps to centre of top left pixel
+ print(ds.xy(0,0)) # Same as above
+ print(ds.transform * (0,0)) # Maps to top left corner of top left pixel
+ print(ds.xy(-0.5,-0.5)) # Same as above
+ print(ds.transform[0], ds.transform[4])
+```
+
+***
diff --git a/book/es-419/02_Software_Tools_Techniques/02_Array_Manipulation_with_Xarray.md b/book/es-419/02_Software_Tools_Techniques/02_Array_Manipulation_with_Xarray.md
new file mode 100644
index 0000000..d99c045
--- /dev/null
+++ b/book/es-419/02_Software_Tools_Techniques/02_Array_Manipulation_with_Xarray.md
@@ -0,0 +1,317 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Manipulación de arreglos con [Xarray](https://docs.xarray.dev/en/stable/index.html)
+
+
+
+Hay numerosas formas de trabajar con datos geoespaciales, así que elegir una herramienta puede ser difícil. La principal librería que utilizaremos es [_Xarray_](https://docs.xarray.dev/en/stable/index.html) por sus estructuras de datos `DataArray` y `Dataset`, y sus utilidades asociadas, así como [NumPy](https://numpy.org) y [Pandas](https://pandas.pydata.org) para manipular arreglos numéricos homogéneos y datos tabulares, respectivamente.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+from warnings import filterwarnings
+filterwarnings('ignore')
+from pathlib import Path
+import numpy as np, pandas as pd, xarray as xr
+import rioxarray as rio
+
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+```
+
+***
+
+
+
+ +
+La principal estructura de datos de Xarray es [`DataArray`](https://docs.xarray.dev/en/stable/user-guide/data-structures.html), que ofrece soporte para arreglos multidimensionales etiquetados. El [Projecto Pythia](https://foundations.projectpythia.org/core/xarray.html) proporciona una amplia introducción a este paquete. Nos enfocaremos principalmente en las partes específicas del API Xarray que utilizaremos para nuestros análisis geoespaciales particulares.
+
+Vamos a cargar una estructura de datos `xarray.DataArray` de ejemplo desde un archivo cuya ubicación viene determinada por `LOCAL_PATH`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif')
+data = rio.open_rasterio(LOCAL_PATH)
+```
+
+***
+
+## Análisis de la `repr` enriquecida de `DataArray`
+
+
+
+Cuando se utiliza un cuaderno computacional de Jupyter, los datos Xarray `DataArray` `data` se pueden analizar de forma interactiva.
+
+- La celda de salida contiene un cuaderno computacional Jupyter `repr` enriquecido para la clase `DataArray`.
+- Los triángulos situados junto a los encabezados "Coordinates", "Indexes" y "Attributes" pueden pulsarse con el mouse para mostrar una vista ampliada.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(f'{type(data)=}\n')
+data
+```
+
+***
+
+## Análisis de los atributos de `DataArray` mediante programación
+
+
+
+Por supuesto, aunque esta vista gráfica es práctica, también es posible acceder a varios atributos de `DataArray` mediante programación. Esto nos permite escribir una lógica programatica para manipular los `DataArray` condicionalmente según sea necesario. Por ejemplo:
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(data.coords)
+```
+
+
+
+Las dimensiones `data.dims` son las cadenas/etiquetas asociadas a los ejes del `DataArray`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.dims
+```
+
+
+
+Podemos extraer las coordenadas como arreglos NumPy unidimensionales (homogéneas) utilizando los atributos `coords` y `.values`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(data.coords['x'].values)
+```
+
+
+
+`data.attrs` es un diccionario que contiene otros metadatos analizados a partir de las etiquetas GeoTIFF (los "Atributos" en la vista gráfica). Una vez más, esta es la razón por la que `rioxarray` es útil. Es posible escribir código que cargue datos de varios formatos de archivo en Xarray `DataArray`, pero este paquete encapsula mucho del código desordenado que, por ejemplo, rellenaría `data.attrs`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.attrs
+```
+
+***
+
+## Uso del método de acceso `rio` de `DataArray`
+
+
+
+Tal como se mencionó, `rioxarray` extiende la clase `xarray.DataArray` con un método de acceso llamado `rio`. El método de acceso `rio` agrega efectivamente un espacio de nombres con una variedad de atributos. Podemos usar una lista de comprensión de Python para mostrar los que no empiezan con guión bajo (los llamados métodos/atributos "private" y "dunder").
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+[name for name in dir(data.rio) if not name.startswith('_')]
+```
+
+
+
+El atributo `data.rio.crs` es importante para nuestros propósitos. Proporciona acceso al sistema de referencia de coordenadas asociado a este conjunto de datos ráster.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(type(data.rio.crs))
+print(data.rio.crs)
+```
+
+
+
+El atributo `.rio.crs` es una estructura de datos de la clase `CRS` del proyecto [pyproj](https://pyproj4.github.io/pyproj/stable/index.html). La `repr` de Python para esta clase devuelve una cadena como `EPSG:32610`. Este número se refiere al [conjunto de datos de parámetros geodésicos _European Petroleum Survey Group_ (EPGS)](https://en.wikipedia.org/wiki/EPSG_Geodetic_Parameter_Dataset) (en español, Grupo Europeo de Estudio sobre el Petróleo).
+
+De [Wikipedia](https://en.wikipedia.org/wiki/EPSG_Geodetic_Parameter_Dataset):
+
+> El [EPSG Geodetic Parameter Dataset (también conocido como registro EPSG)](https://en.wikipedia.org/wiki/EPSG_Geodetic_Parameter_Dataset) es un registro público de [datums geodésicos](https://es.wikipedia.org/wiki/Sistema_de_referencia_geod%C3%A9sico), [sistemas de referencia espacial](https://es.wikipedia.org/wiki/Sistema_de_referencia_espacial), [elipsoides terrestres](https://es.wikipedia.org/wiki/Elipsoide_de_referencia), transformaciones de coordenadas y [unidades de medida](https://es.wikipedia.org/wiki/Unidad_de_medida) relacionadas, originados por un miembro del [EPGS](https://en.wikipedia.org/wiki/European_Petroleum_Survey_Group) en 1985. A cada entidad se le asigna un código EPSG comprendido entre 1024 y 32767, junto con una representación estándar de [texto conocido (WKT)](https://en.wikipedia.org/wiki/Well-known_text_representation_of_coordinate_reference_systems) legible por máquina. El mantenimiento del conjunto de datos corre a cargo del Comité de Geomática [IOGP](https://en.wikipedia.org/wiki/International_Association_of_Oil_%26_Gas_Producers).
+
+
+
+***
+
+## Manipulación de los datos en un `DataArray`
+
+
+
+Estos datos se almacenan utilizando un CRS [ sistema de coordenadas universal transversal de Mercator (UTM)](https://en.wikipedia.org/wiki/Universal_Transverse_Mercator_coordinate_system) (por sus siglas en inglés de _Mercator transversal universal_) particular. Las etiquetas de las coordenadas serían convencionalmente _este_ y _norte_. Sin embargo, a la hora de hacer el trazo, será conveniente utilizar _longitud_ y _latitud_ en su lugar. Reetiquetaremos las coordenadas para reflejar esto, es decir, la coordenada llamada `x` se reetiquetará como `longitude` y la coordenada llamada `y` se reetiquetará como `latitude`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data = data.rename({'x':'longitude', 'y':'latitude'})
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(data.coords)
+```
+
+
+
+Una vez más, aunque los valores numéricos almacenados en los arreglos de coordenadas no tienen sentido estrictamente como valores (longitud, latitud), aplicaremos estas etiquetas ahora para simplificar el trazado más adelante.
+
+Los objetos Xarray `DataArray` permiten estraer subconjuntos de forma muy similar a las listas de Python. Las dos celdas siguientes extraen ambas el mismo subarreglo mediante dos llamadas a métodos diferentes.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.isel(longitude=slice(0,2))
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.sel(longitude=[499_995, 500_025])
+```
+
+
+
+En vez de utilizar paréntesis para cortar secciones de arreglos (como en NumPy), para `DataArray`, podemos utilizar los métodos `sel` o `isel` para seleccionar subconjuntos por valores de coordenadas continuas o por posiciones enteras (es decir, coordenadas de "píxel") respectivamente. Esto es similar al uso de `.loc` and `.iloc` en Pandas para extraer entradas de una Pandas `Series` o `DataFrame`.
+
+Si tomamos un subconjunto en 2D de los `DataArray` `data` 3D, podemos graficarlo usando el método de acceso `.plot` (hablaremos al respecto más adelante).
+
+
+
+```python jupyter={"source_hidden": false}
+data.isel(band=0).plot();
+```
+
+
+
+Este gráfico tarda un poco en procesarse porque el arreglo representado tiene $3,600\times3,600$ píxeles. Podemos utilizar la función `slice` de Python para extraer, por ejemplo, cada 100 píxeles en cualquier dirección para trazar una imagen de menor resolución mucho más rápido.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 100
+subset = slice(0,None,steps)
+view = data.isel(longitude=subset, latitude=subset, band=0)
+view.plot();
+```
+
+
+
+El gráfico producido es bastante oscuro (lo que refleja que la mayoría de las entradas son cero según la leyenda). Observa que los ejes se etiquetan automáticamente utilizando las `coords` que renombramos antes.
+
+
+
+***
+
+## Extracción de datos `DataArray` a NumPy, Pandas
+
+
+
+Observa que un `DataArray` encapsula de un arreglo NumPy. Ese arreglo NumPy se puede recuperar usando el atributo `.values`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+array = data.values
+print(f'{type(array)=}')
+print(f'{array.shape=}')
+print(f'{array.dtype=}')
+print(f'{array.nbytes=}')
+```
+
+
+
+Estos datos ráster se almacenan como datos enteros sin signo de 8 bits, es decir, un byte por cada píxel. Un entero de 8 bits sin signo puede representar valores enteros entre 0 y 255. En un arreglo con algo más de trece millones de elementos, eso significa que hay muchos valores repetidos. Podemos verlo poniendo los valores de los píxeles en una Pandas `Series` y usando el método `.value_counts`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+s_flat = pd.Series(array.flatten()).value_counts()
+s_flat.sort_index()
+```
+
+
+
+La mayoría de las entradas de este arreglo ráster son cero. Los valores numéricos varían entre 0 y 100 con la excepción de unos 1,700 píxeles con el valor 255. Esto tendrá más sentido cuando hablemos de la especificación del producto de datos DIST.
+
+
+
+***
+
+## Acumulación y concatenación de una secuencia de `DataArrays`
+
+
+
+A menudo es conveniente apilar múltiples arreglos bidimensionales de datos ráster en un único arreglo tridimensional. En NumPy, esto se hace típicamente con la función [`numpy.concatenate`](https://numpy.org/doc/stable/reference/generated/numpy.concatenate.html). Hay una funcionalidad similar en Xarray—[`xarray.concat`](https://docs.xarray.dev/en/stable/generated/xarray.concat.html) (que es similar en diseño a la función [`pandas.concat`](https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.concat.html)). La principal diferencia entre `numpy.concatenate` y `xarray.concat` es que esta última función debe tener en cuenta las _coordenadas etiquetadas_, mientras que la primera no. Esto es importante cuando, por ejemplo, los ejes de coordenadas de dos rásters se superponen pero no están perfectamente alineados.
+
+Para ver cómo funciona el apilamiento de rásteres, empezaremos haciendo una lista de tres archivos GeoTIFF (almacenados localmente), inicializando una lista de `stack` vacía, y después construyendo una lista de `DataArrays` en un bucle.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+RASTER_FILES = list((Path(FILE_STEM, 'assets').glob('OPERA*VEG*.tif')))
+
+stack = []
+for path in RASTER_FILES:
+ print(f"Stacking {path.name}..")
+ data = rio.open_rasterio(path).rename(dict(x='longitude', y='latitude'))
+ band_name = path.stem.split('_')[-1]
+ data.coords.update({'band': [band_name]})
+ data.attrs = dict(description=f"OPERA DIST product", units=None)
+ stack.append(data)
+```
+
+
+
+He aquí algunas observaciones importantes sobre el bucle de código anterior:
+
+- El uso de `rioxarray.open_rasterio` para cargar un Xarray `DataArray` en memoria hace mucho trabajo por nosotros. En particular, se asegura de que las coordenadas continuas están alineadas con las coordenadas de píxeles subyacentes.
+- De manera predeterminada, `data.coords` tiene las claves `x` y `y` que elegimos reetiquetar como `longitude` y `latitude` respectivamente. Técnicamente, los valores de las coordenadas continuas que se cargaron desde este archivo GeoTIFF en particular se expresan en coordenadas UTM (es decir, este y norte), pero, posteriormente, al trazar, las etiquetas `longitude` y `latitude` serán más convenientes.
+- `data.coords['band']`, tal como se cargó desde el archivo, tiene el valor `1`. Elegimos sobrescribir ese valor con el nombre de la banda (que extraemos del nombre del archivo como `band_name`).
+- De manera predeterminada, `rioxarray.open_rasterio` completa `data.attrs` con pares clave-valor extraídos de las etiquetas TIFF. Para diferentes bandas/capas, estos diccionarios de atributos podrían tener claves o valores conflictivos. Puede ser aconsejable conservar estos metadatos en algunas circunstancias. Simplemente elegimos descartarlos en este contexto para evitar posibles conflictos. El diccionario mínimo de atributos de la estructura de datos final tendrá como únicas claves `description` y `units`.
+
+Dado que construimos una lista de `DataArray` en la lista `stack`, podemos ensamblar un `DataArray` tridimensional utilizando `xarray.concat`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack = xr.concat(stack, dim='band')
+```
+
+
+
+La función `xarray.concat` acepta una secuencia de objetos `xarray.DataArray` con dimensiones conformes y los _concatena_ a lo largo de una dimensión especificada. Para este ejemplo, apilamos rásteres bidimensionales que corresponden a diferentes bandas o capas. Por eso utilizamos la opción `dim='band'` en la llamada a `xarray.concat`. Más adelante, en cambio, apilaremos rásteres bidimensionales a lo largo de un eje _temporal_ (esto implica un código ligeramente diferente para garantizar el etiquetado y la alineación correctos).
+
+Examinemos `stack` mediante su `repr`en este cuaderno computacional Jupyter.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack
+```
+
+
+
+Observa que `stack` tiene un CRS asociado que fue analizado por `rioxarray.open_rasterio`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack.rio.crs
+```
+
+
+
+Este proceso es muy útil para el análisis (suponiendo que haya suficiente memoria disponible para almacenar toda la colección de rásteres). Más adelante, utilizaremos este enfoque varias veces para manipular colecciones de rásteres de dimensiones conformes. El apilamiento se puede utilizar para producir una visualización dinámica con un control deslizante o, alternativamente, para producir un gráfico estático.
+
+
+
+***
diff --git a/book/es-419/02_Software_Tools_Techniques/03_Data_Visualization_with_GeoViews_HvPlot.md b/book/es-419/02_Software_Tools_Techniques/03_Data_Visualization_with_GeoViews_HvPlot.md
new file mode 100644
index 0000000..e156d8b
--- /dev/null
+++ b/book/es-419/02_Software_Tools_Techniques/03_Data_Visualization_with_GeoViews_HvPlot.md
@@ -0,0 +1,486 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Visualización de datos con GeoViews y HvPlot
+
+
+
+Las principales herramientas que utilizaremos para la visualización de datos provienen de la familia [Holoviz](https://holoviz.org/) de librerías Python, principalmente [GeoViews](https://geoviews.org/) y [hvPlot](https://hvplot.holoviz.org/). Estas están construidas en gran parte sobre [HoloViews](https://holoviews.org/) y soportan múltiples _backends_ para la representación de gráficos ([Bokeh](http://bokeh.pydata.org/) para visualización interactiva y [Matplotlib](http://matplotlib.org/) para gráficos estáticos con calidad de publicación.
+
+
+
+***
+
+## [GeoViews](https://geoviews.org/)
+
+
+
+
+
+La principal estructura de datos de Xarray es [`DataArray`](https://docs.xarray.dev/en/stable/user-guide/data-structures.html), que ofrece soporte para arreglos multidimensionales etiquetados. El [Projecto Pythia](https://foundations.projectpythia.org/core/xarray.html) proporciona una amplia introducción a este paquete. Nos enfocaremos principalmente en las partes específicas del API Xarray que utilizaremos para nuestros análisis geoespaciales particulares.
+
+Vamos a cargar una estructura de datos `xarray.DataArray` de ejemplo desde un archivo cuya ubicación viene determinada por `LOCAL_PATH`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif')
+data = rio.open_rasterio(LOCAL_PATH)
+```
+
+***
+
+## Análisis de la `repr` enriquecida de `DataArray`
+
+
+
+Cuando se utiliza un cuaderno computacional de Jupyter, los datos Xarray `DataArray` `data` se pueden analizar de forma interactiva.
+
+- La celda de salida contiene un cuaderno computacional Jupyter `repr` enriquecido para la clase `DataArray`.
+- Los triángulos situados junto a los encabezados "Coordinates", "Indexes" y "Attributes" pueden pulsarse con el mouse para mostrar una vista ampliada.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(f'{type(data)=}\n')
+data
+```
+
+***
+
+## Análisis de los atributos de `DataArray` mediante programación
+
+
+
+Por supuesto, aunque esta vista gráfica es práctica, también es posible acceder a varios atributos de `DataArray` mediante programación. Esto nos permite escribir una lógica programatica para manipular los `DataArray` condicionalmente según sea necesario. Por ejemplo:
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(data.coords)
+```
+
+
+
+Las dimensiones `data.dims` son las cadenas/etiquetas asociadas a los ejes del `DataArray`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.dims
+```
+
+
+
+Podemos extraer las coordenadas como arreglos NumPy unidimensionales (homogéneas) utilizando los atributos `coords` y `.values`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(data.coords['x'].values)
+```
+
+
+
+`data.attrs` es un diccionario que contiene otros metadatos analizados a partir de las etiquetas GeoTIFF (los "Atributos" en la vista gráfica). Una vez más, esta es la razón por la que `rioxarray` es útil. Es posible escribir código que cargue datos de varios formatos de archivo en Xarray `DataArray`, pero este paquete encapsula mucho del código desordenado que, por ejemplo, rellenaría `data.attrs`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.attrs
+```
+
+***
+
+## Uso del método de acceso `rio` de `DataArray`
+
+
+
+Tal como se mencionó, `rioxarray` extiende la clase `xarray.DataArray` con un método de acceso llamado `rio`. El método de acceso `rio` agrega efectivamente un espacio de nombres con una variedad de atributos. Podemos usar una lista de comprensión de Python para mostrar los que no empiezan con guión bajo (los llamados métodos/atributos "private" y "dunder").
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+[name for name in dir(data.rio) if not name.startswith('_')]
+```
+
+
+
+El atributo `data.rio.crs` es importante para nuestros propósitos. Proporciona acceso al sistema de referencia de coordenadas asociado a este conjunto de datos ráster.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(type(data.rio.crs))
+print(data.rio.crs)
+```
+
+
+
+El atributo `.rio.crs` es una estructura de datos de la clase `CRS` del proyecto [pyproj](https://pyproj4.github.io/pyproj/stable/index.html). La `repr` de Python para esta clase devuelve una cadena como `EPSG:32610`. Este número se refiere al [conjunto de datos de parámetros geodésicos _European Petroleum Survey Group_ (EPGS)](https://en.wikipedia.org/wiki/EPSG_Geodetic_Parameter_Dataset) (en español, Grupo Europeo de Estudio sobre el Petróleo).
+
+De [Wikipedia](https://en.wikipedia.org/wiki/EPSG_Geodetic_Parameter_Dataset):
+
+> El [EPSG Geodetic Parameter Dataset (también conocido como registro EPSG)](https://en.wikipedia.org/wiki/EPSG_Geodetic_Parameter_Dataset) es un registro público de [datums geodésicos](https://es.wikipedia.org/wiki/Sistema_de_referencia_geod%C3%A9sico), [sistemas de referencia espacial](https://es.wikipedia.org/wiki/Sistema_de_referencia_espacial), [elipsoides terrestres](https://es.wikipedia.org/wiki/Elipsoide_de_referencia), transformaciones de coordenadas y [unidades de medida](https://es.wikipedia.org/wiki/Unidad_de_medida) relacionadas, originados por un miembro del [EPGS](https://en.wikipedia.org/wiki/European_Petroleum_Survey_Group) en 1985. A cada entidad se le asigna un código EPSG comprendido entre 1024 y 32767, junto con una representación estándar de [texto conocido (WKT)](https://en.wikipedia.org/wiki/Well-known_text_representation_of_coordinate_reference_systems) legible por máquina. El mantenimiento del conjunto de datos corre a cargo del Comité de Geomática [IOGP](https://en.wikipedia.org/wiki/International_Association_of_Oil_%26_Gas_Producers).
+
+
+
+***
+
+## Manipulación de los datos en un `DataArray`
+
+
+
+Estos datos se almacenan utilizando un CRS [ sistema de coordenadas universal transversal de Mercator (UTM)](https://en.wikipedia.org/wiki/Universal_Transverse_Mercator_coordinate_system) (por sus siglas en inglés de _Mercator transversal universal_) particular. Las etiquetas de las coordenadas serían convencionalmente _este_ y _norte_. Sin embargo, a la hora de hacer el trazo, será conveniente utilizar _longitud_ y _latitud_ en su lugar. Reetiquetaremos las coordenadas para reflejar esto, es decir, la coordenada llamada `x` se reetiquetará como `longitude` y la coordenada llamada `y` se reetiquetará como `latitude`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data = data.rename({'x':'longitude', 'y':'latitude'})
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(data.coords)
+```
+
+
+
+Una vez más, aunque los valores numéricos almacenados en los arreglos de coordenadas no tienen sentido estrictamente como valores (longitud, latitud), aplicaremos estas etiquetas ahora para simplificar el trazado más adelante.
+
+Los objetos Xarray `DataArray` permiten estraer subconjuntos de forma muy similar a las listas de Python. Las dos celdas siguientes extraen ambas el mismo subarreglo mediante dos llamadas a métodos diferentes.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.isel(longitude=slice(0,2))
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.sel(longitude=[499_995, 500_025])
+```
+
+
+
+En vez de utilizar paréntesis para cortar secciones de arreglos (como en NumPy), para `DataArray`, podemos utilizar los métodos `sel` o `isel` para seleccionar subconjuntos por valores de coordenadas continuas o por posiciones enteras (es decir, coordenadas de "píxel") respectivamente. Esto es similar al uso de `.loc` and `.iloc` en Pandas para extraer entradas de una Pandas `Series` o `DataFrame`.
+
+Si tomamos un subconjunto en 2D de los `DataArray` `data` 3D, podemos graficarlo usando el método de acceso `.plot` (hablaremos al respecto más adelante).
+
+
+
+```python jupyter={"source_hidden": false}
+data.isel(band=0).plot();
+```
+
+
+
+Este gráfico tarda un poco en procesarse porque el arreglo representado tiene $3,600\times3,600$ píxeles. Podemos utilizar la función `slice` de Python para extraer, por ejemplo, cada 100 píxeles en cualquier dirección para trazar una imagen de menor resolución mucho más rápido.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 100
+subset = slice(0,None,steps)
+view = data.isel(longitude=subset, latitude=subset, band=0)
+view.plot();
+```
+
+
+
+El gráfico producido es bastante oscuro (lo que refleja que la mayoría de las entradas son cero según la leyenda). Observa que los ejes se etiquetan automáticamente utilizando las `coords` que renombramos antes.
+
+
+
+***
+
+## Extracción de datos `DataArray` a NumPy, Pandas
+
+
+
+Observa que un `DataArray` encapsula de un arreglo NumPy. Ese arreglo NumPy se puede recuperar usando el atributo `.values`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+array = data.values
+print(f'{type(array)=}')
+print(f'{array.shape=}')
+print(f'{array.dtype=}')
+print(f'{array.nbytes=}')
+```
+
+
+
+Estos datos ráster se almacenan como datos enteros sin signo de 8 bits, es decir, un byte por cada píxel. Un entero de 8 bits sin signo puede representar valores enteros entre 0 y 255. En un arreglo con algo más de trece millones de elementos, eso significa que hay muchos valores repetidos. Podemos verlo poniendo los valores de los píxeles en una Pandas `Series` y usando el método `.value_counts`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+s_flat = pd.Series(array.flatten()).value_counts()
+s_flat.sort_index()
+```
+
+
+
+La mayoría de las entradas de este arreglo ráster son cero. Los valores numéricos varían entre 0 y 100 con la excepción de unos 1,700 píxeles con el valor 255. Esto tendrá más sentido cuando hablemos de la especificación del producto de datos DIST.
+
+
+
+***
+
+## Acumulación y concatenación de una secuencia de `DataArrays`
+
+
+
+A menudo es conveniente apilar múltiples arreglos bidimensionales de datos ráster en un único arreglo tridimensional. En NumPy, esto se hace típicamente con la función [`numpy.concatenate`](https://numpy.org/doc/stable/reference/generated/numpy.concatenate.html). Hay una funcionalidad similar en Xarray—[`xarray.concat`](https://docs.xarray.dev/en/stable/generated/xarray.concat.html) (que es similar en diseño a la función [`pandas.concat`](https://pandas.pydata.org/pandas-docs/stable/reference/api/pandas.concat.html)). La principal diferencia entre `numpy.concatenate` y `xarray.concat` es que esta última función debe tener en cuenta las _coordenadas etiquetadas_, mientras que la primera no. Esto es importante cuando, por ejemplo, los ejes de coordenadas de dos rásters se superponen pero no están perfectamente alineados.
+
+Para ver cómo funciona el apilamiento de rásteres, empezaremos haciendo una lista de tres archivos GeoTIFF (almacenados localmente), inicializando una lista de `stack` vacía, y después construyendo una lista de `DataArrays` en un bucle.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+RASTER_FILES = list((Path(FILE_STEM, 'assets').glob('OPERA*VEG*.tif')))
+
+stack = []
+for path in RASTER_FILES:
+ print(f"Stacking {path.name}..")
+ data = rio.open_rasterio(path).rename(dict(x='longitude', y='latitude'))
+ band_name = path.stem.split('_')[-1]
+ data.coords.update({'band': [band_name]})
+ data.attrs = dict(description=f"OPERA DIST product", units=None)
+ stack.append(data)
+```
+
+
+
+He aquí algunas observaciones importantes sobre el bucle de código anterior:
+
+- El uso de `rioxarray.open_rasterio` para cargar un Xarray `DataArray` en memoria hace mucho trabajo por nosotros. En particular, se asegura de que las coordenadas continuas están alineadas con las coordenadas de píxeles subyacentes.
+- De manera predeterminada, `data.coords` tiene las claves `x` y `y` que elegimos reetiquetar como `longitude` y `latitude` respectivamente. Técnicamente, los valores de las coordenadas continuas que se cargaron desde este archivo GeoTIFF en particular se expresan en coordenadas UTM (es decir, este y norte), pero, posteriormente, al trazar, las etiquetas `longitude` y `latitude` serán más convenientes.
+- `data.coords['band']`, tal como se cargó desde el archivo, tiene el valor `1`. Elegimos sobrescribir ese valor con el nombre de la banda (que extraemos del nombre del archivo como `band_name`).
+- De manera predeterminada, `rioxarray.open_rasterio` completa `data.attrs` con pares clave-valor extraídos de las etiquetas TIFF. Para diferentes bandas/capas, estos diccionarios de atributos podrían tener claves o valores conflictivos. Puede ser aconsejable conservar estos metadatos en algunas circunstancias. Simplemente elegimos descartarlos en este contexto para evitar posibles conflictos. El diccionario mínimo de atributos de la estructura de datos final tendrá como únicas claves `description` y `units`.
+
+Dado que construimos una lista de `DataArray` en la lista `stack`, podemos ensamblar un `DataArray` tridimensional utilizando `xarray.concat`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack = xr.concat(stack, dim='band')
+```
+
+
+
+La función `xarray.concat` acepta una secuencia de objetos `xarray.DataArray` con dimensiones conformes y los _concatena_ a lo largo de una dimensión especificada. Para este ejemplo, apilamos rásteres bidimensionales que corresponden a diferentes bandas o capas. Por eso utilizamos la opción `dim='band'` en la llamada a `xarray.concat`. Más adelante, en cambio, apilaremos rásteres bidimensionales a lo largo de un eje _temporal_ (esto implica un código ligeramente diferente para garantizar el etiquetado y la alineación correctos).
+
+Examinemos `stack` mediante su `repr`en este cuaderno computacional Jupyter.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack
+```
+
+
+
+Observa que `stack` tiene un CRS asociado que fue analizado por `rioxarray.open_rasterio`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack.rio.crs
+```
+
+
+
+Este proceso es muy útil para el análisis (suponiendo que haya suficiente memoria disponible para almacenar toda la colección de rásteres). Más adelante, utilizaremos este enfoque varias veces para manipular colecciones de rásteres de dimensiones conformes. El apilamiento se puede utilizar para producir una visualización dinámica con un control deslizante o, alternativamente, para producir un gráfico estático.
+
+
+
+***
diff --git a/book/es-419/02_Software_Tools_Techniques/03_Data_Visualization_with_GeoViews_HvPlot.md b/book/es-419/02_Software_Tools_Techniques/03_Data_Visualization_with_GeoViews_HvPlot.md
new file mode 100644
index 0000000..e156d8b
--- /dev/null
+++ b/book/es-419/02_Software_Tools_Techniques/03_Data_Visualization_with_GeoViews_HvPlot.md
@@ -0,0 +1,486 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Visualización de datos con GeoViews y HvPlot
+
+
+
+Las principales herramientas que utilizaremos para la visualización de datos provienen de la familia [Holoviz](https://holoviz.org/) de librerías Python, principalmente [GeoViews](https://geoviews.org/) y [hvPlot](https://hvplot.holoviz.org/). Estas están construidas en gran parte sobre [HoloViews](https://holoviews.org/) y soportan múltiples _backends_ para la representación de gráficos ([Bokeh](http://bokeh.pydata.org/) para visualización interactiva y [Matplotlib](http://matplotlib.org/) para gráficos estáticos con calidad de publicación.
+
+
+
+***
+
+## [GeoViews](https://geoviews.org/)
+
+
+
+ +
+De la [documentación de GeoViews](https://geoviews.org/index.html):
+
+> GeoViews es una librería de [Python](http://python.org/) que facilita la exploración y visualización de conjuntos de datos geográficos, meteorológicos y oceanográficos, como los que se utilizan en la investigación meteorológica, climática y de teledetección.
+>
+> GeoViews se basa en la biblioteca [HoloViews](http://holoviews.org/) y permite crear visualizaciones flexibles de datos multidimensionales. GeoViews agrega una familia de tipos de gráficos geográficos basados en la librería [Cartopy](http://scitools.org.uk/cartopy), trazados con los paquetes [Matplotlib](http://matplotlib.org/) o [Bokeh](http://bokeh.pydata.org/). Con GeoViews, puedes trabajar de forma fácil y natural con grandes conjuntos de datos geográficos multidimensionales, visualizando al instante cualquier subconjunto o combinación de ellos. Al mismo tiempo, podrás acceder siempre a los datos crudos subyacentes a cualquier gráfico.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import warnings
+warnings.filterwarnings('ignore')
+from pathlib import Path
+from pprint import pprint
+
+import geoviews as gv
+gv.extension('bokeh')
+from geoviews import opts
+
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+```
+
+***
+
+### Visualización de un mapa base
+
+
+
+Un _mapa base_ o _capa de mosaico_ es útil cuando se muestran datos vectoriales o ráster porque nos permite superponer los datos geoespaciales relevantes sobre un mapa geográfico conocido como fondo. La principal funcionalidad que utilizaremos es `gv.tile_sources`. Podemos utilizar el método `opts` para especificar parámetros de configuración adicionales. A continuación, utilizaremos el servicio de mapas web _Open Street Map (OSM)_ (en español, Mapas de Calles Abiertos) para crear el objeto `basemap`. Cuando mostramos la representación de este objeto en la celda del cuaderno computacional, el menú de Bokeh que está a la derecha permite la exploración interactiva.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+basemap = gv.tile_sources.OSM.opts(width=600, height=400)
+basemap # When displayed, this basemap can be zoomed & panned using the menu at the right
+```
+
+***
+
+### Gráficos de puntos
+
+
+
+Para empezar, vamos a definir una tupla regular en Python para las coordenadas de longitud y latitud de Tokio, Japón.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+tokyo_lonlat = (139.692222, 35.689722)
+print(tokyo_lonlat)
+```
+
+
+
+La clase `geoviews.Points` acepta una lista de tuplas (cada una de la forma `(x, y)`) y construye un objeto `Points` que puede ser visualizado. Podemos superponer el punto creado en los mosaicos OpenStreetMap de `basemap` utilizando el operador `*` en Holoviews. También podemos utilizar `geoviews.opts` para establecer varias preferencias de visualización para estos puntos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+tokyo_point = gv.Points([tokyo_lonlat])
+point_opts = opts.Points(
+ size=48,
+ alpha=0.5,
+ color='red'
+ )
+print(type(tokyo_point))
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Use Holoviews * operator to overlay plot on basemap
+# Note: zoom out to see basemap (starts zoomed "all the way in")
+(basemap * tokyo_point).opts(point_opts)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# to avoid starting zoomed all the way in, this zooms "all the way out"
+(basemap * tokyo_point).opts(point_opts, opts.Overlay(global_extent=True))
+```
+
+***
+
+### Gráficos de rectángulos
+
+
+
+- Forma estándar de representar un rectángulo (también llamado caja delimitadora) con vértices $$(x_{\mathrm{min}},y_{\mathrm{min}}), (x_{\mathrm{min}},y_{\mathrm{max}}), (x_{\mathrm{max}},y_{\mathrm{min}}), (x_{\mathrm{max}},y_{\mathrm{max}})$$
+ (suponiendo que $x_{\mathrm{max}}>x_{\mathrm{min}}$ & $y_{\mathrm{max}}>y_{\mathrm{min}}$) es como única cuadrupla
+ $$(x_{\mathrm{min}},y_{\mathrm{min}},x_{\mathrm{max}},y_{\mathrm{max}}),$$
+ es decir, las coordenadas de la esquina inferior izquierda seguidas de las coordenadas de la esquina superior derecha.
+
+ Vamos a crear una función sencilla para generar un rectángulo de un ancho y altura dados, según la coordenada central.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# simple utility to make a rectangle centered at pt of width dx & height dy
+def make_bbox(pt,dx,dy):
+ '''Returns bounding box represented as tuple (x_lo, y_lo, x_hi, y_hi)
+ given inputs pt=(x, y), width & height dx & dy respectively,
+ where x_lo = x-dx/2, x_hi=x+dx/2, y_lo = y-dy/2, y_hi = y+dy/2.
+ '''
+ return tuple(coord+sgn*delta for sgn in (-1,+1) for coord,delta in zip(pt, (dx/2,dy/2)))
+```
+
+
+
+Podemos probar la función anterior utilizando las coordenadas de longitud y latitud de Marruecos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Verify that the function bounds works as intended
+marrakesh_lonlat = (-7.93, 31.67)
+dlon, dlat = 0.5, 0.25
+marrakesh_bbox = make_bbox(marrakesh_lonlat, dlon, dlat)
+print(marrakesh_bbox)
+```
+
+
+
+La función `geoviews.Rectangles` acepta una lista de cajas delimitadoras (cada uno descrito por una tupla de la forma `(x_min, y_min, x_max, y_max)`) para el trazado. También podemos utilizar `geoviews.opts` para adaptar el rectángulo a nuestras necesidades.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+rectangle = gv.Rectangles([marrakesh_bbox])
+rect_opts = opts.Rectangles(
+ line_width=0,
+ alpha=0.1,
+ color='red'
+ )
+```
+
+
+
+Podemos graficar un punto para Marruecos al igual que antes utilizando `geoviews.Points` (personalizado utilizando `geoviews.opts`).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+marrakesh_point = gv.Points([marrakesh_lonlat])
+point_opts = opts.Points(
+ size=48,
+ alpha=0.25,
+ color='blue'
+ )
+```
+
+
+
+Por último, podemos superponer todas estas características en el mapa base con las opciones aplicadas.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+(basemap * rectangle * marrakesh_point).opts( rect_opts, point_opts )
+```
+
+
+
+Utilizaremos el método anterior para visualizar _(AOIs)_ al construir consultas de búsqueda para los productos EarthData de la NASA. En particular, la convención de representar una caja delimitadora por ordenadas (izquierda, inferior, derecha, superior) también se utiliza en la API [PySTAC](https://pystac.readthedocs.io/en/stable/).
+
+
+
+***
+
+## [hvPlot](https://hvplot.holoviz.org/)
+
+
+
+
+
+De la [documentación de GeoViews](https://geoviews.org/index.html):
+
+> GeoViews es una librería de [Python](http://python.org/) que facilita la exploración y visualización de conjuntos de datos geográficos, meteorológicos y oceanográficos, como los que se utilizan en la investigación meteorológica, climática y de teledetección.
+>
+> GeoViews se basa en la biblioteca [HoloViews](http://holoviews.org/) y permite crear visualizaciones flexibles de datos multidimensionales. GeoViews agrega una familia de tipos de gráficos geográficos basados en la librería [Cartopy](http://scitools.org.uk/cartopy), trazados con los paquetes [Matplotlib](http://matplotlib.org/) o [Bokeh](http://bokeh.pydata.org/). Con GeoViews, puedes trabajar de forma fácil y natural con grandes conjuntos de datos geográficos multidimensionales, visualizando al instante cualquier subconjunto o combinación de ellos. Al mismo tiempo, podrás acceder siempre a los datos crudos subyacentes a cualquier gráfico.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import warnings
+warnings.filterwarnings('ignore')
+from pathlib import Path
+from pprint import pprint
+
+import geoviews as gv
+gv.extension('bokeh')
+from geoviews import opts
+
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+```
+
+***
+
+### Visualización de un mapa base
+
+
+
+Un _mapa base_ o _capa de mosaico_ es útil cuando se muestran datos vectoriales o ráster porque nos permite superponer los datos geoespaciales relevantes sobre un mapa geográfico conocido como fondo. La principal funcionalidad que utilizaremos es `gv.tile_sources`. Podemos utilizar el método `opts` para especificar parámetros de configuración adicionales. A continuación, utilizaremos el servicio de mapas web _Open Street Map (OSM)_ (en español, Mapas de Calles Abiertos) para crear el objeto `basemap`. Cuando mostramos la representación de este objeto en la celda del cuaderno computacional, el menú de Bokeh que está a la derecha permite la exploración interactiva.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+basemap = gv.tile_sources.OSM.opts(width=600, height=400)
+basemap # When displayed, this basemap can be zoomed & panned using the menu at the right
+```
+
+***
+
+### Gráficos de puntos
+
+
+
+Para empezar, vamos a definir una tupla regular en Python para las coordenadas de longitud y latitud de Tokio, Japón.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+tokyo_lonlat = (139.692222, 35.689722)
+print(tokyo_lonlat)
+```
+
+
+
+La clase `geoviews.Points` acepta una lista de tuplas (cada una de la forma `(x, y)`) y construye un objeto `Points` que puede ser visualizado. Podemos superponer el punto creado en los mosaicos OpenStreetMap de `basemap` utilizando el operador `*` en Holoviews. También podemos utilizar `geoviews.opts` para establecer varias preferencias de visualización para estos puntos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+tokyo_point = gv.Points([tokyo_lonlat])
+point_opts = opts.Points(
+ size=48,
+ alpha=0.5,
+ color='red'
+ )
+print(type(tokyo_point))
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Use Holoviews * operator to overlay plot on basemap
+# Note: zoom out to see basemap (starts zoomed "all the way in")
+(basemap * tokyo_point).opts(point_opts)
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# to avoid starting zoomed all the way in, this zooms "all the way out"
+(basemap * tokyo_point).opts(point_opts, opts.Overlay(global_extent=True))
+```
+
+***
+
+### Gráficos de rectángulos
+
+
+
+- Forma estándar de representar un rectángulo (también llamado caja delimitadora) con vértices $$(x_{\mathrm{min}},y_{\mathrm{min}}), (x_{\mathrm{min}},y_{\mathrm{max}}), (x_{\mathrm{max}},y_{\mathrm{min}}), (x_{\mathrm{max}},y_{\mathrm{max}})$$
+ (suponiendo que $x_{\mathrm{max}}>x_{\mathrm{min}}$ & $y_{\mathrm{max}}>y_{\mathrm{min}}$) es como única cuadrupla
+ $$(x_{\mathrm{min}},y_{\mathrm{min}},x_{\mathrm{max}},y_{\mathrm{max}}),$$
+ es decir, las coordenadas de la esquina inferior izquierda seguidas de las coordenadas de la esquina superior derecha.
+
+ Vamos a crear una función sencilla para generar un rectángulo de un ancho y altura dados, según la coordenada central.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# simple utility to make a rectangle centered at pt of width dx & height dy
+def make_bbox(pt,dx,dy):
+ '''Returns bounding box represented as tuple (x_lo, y_lo, x_hi, y_hi)
+ given inputs pt=(x, y), width & height dx & dy respectively,
+ where x_lo = x-dx/2, x_hi=x+dx/2, y_lo = y-dy/2, y_hi = y+dy/2.
+ '''
+ return tuple(coord+sgn*delta for sgn in (-1,+1) for coord,delta in zip(pt, (dx/2,dy/2)))
+```
+
+
+
+Podemos probar la función anterior utilizando las coordenadas de longitud y latitud de Marruecos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Verify that the function bounds works as intended
+marrakesh_lonlat = (-7.93, 31.67)
+dlon, dlat = 0.5, 0.25
+marrakesh_bbox = make_bbox(marrakesh_lonlat, dlon, dlat)
+print(marrakesh_bbox)
+```
+
+
+
+La función `geoviews.Rectangles` acepta una lista de cajas delimitadoras (cada uno descrito por una tupla de la forma `(x_min, y_min, x_max, y_max)`) para el trazado. También podemos utilizar `geoviews.opts` para adaptar el rectángulo a nuestras necesidades.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+rectangle = gv.Rectangles([marrakesh_bbox])
+rect_opts = opts.Rectangles(
+ line_width=0,
+ alpha=0.1,
+ color='red'
+ )
+```
+
+
+
+Podemos graficar un punto para Marruecos al igual que antes utilizando `geoviews.Points` (personalizado utilizando `geoviews.opts`).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+marrakesh_point = gv.Points([marrakesh_lonlat])
+point_opts = opts.Points(
+ size=48,
+ alpha=0.25,
+ color='blue'
+ )
+```
+
+
+
+Por último, podemos superponer todas estas características en el mapa base con las opciones aplicadas.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+(basemap * rectangle * marrakesh_point).opts( rect_opts, point_opts )
+```
+
+
+
+Utilizaremos el método anterior para visualizar _(AOIs)_ al construir consultas de búsqueda para los productos EarthData de la NASA. En particular, la convención de representar una caja delimitadora por ordenadas (izquierda, inferior, derecha, superior) también se utiliza en la API [PySTAC](https://pystac.readthedocs.io/en/stable/).
+
+
+
+***
+
+## [hvPlot](https://hvplot.holoviz.org/)
+
+
+
+ +
+- [hvPlot](https://hvplot.holoviz.org/) está diseñado para extender la API `.plot` de `DataFrames` de Pandas.
+- Funciona para `DataFrames` de Pandas y `DataArrays`/`Datasets` de Xarray.
+
+
+
+***
+
+### Graficar desde un DataFrame con hvplot.pandas
+
+
+
+El código siguiente carga un `DataFrame` de Pandas con datos de temperatura.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import pandas as pd, numpy as np
+from pathlib import Path
+LOCAL_PATH = Path(FILE_STEM, 'assets/temperature.csv')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+df = pd.read_csv(LOCAL_PATH, index_col=0, parse_dates=[0])
+df.head()
+```
+
+***
+
+#### Revisando la API de `DataFrame.plot` de Pandas
+
+
+
+Vamos a extraer un subconjunto de columnas de este `DataFrame` y generar un gráfico.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+west_coast = df[['Vancouver', 'Portland', 'San Francisco', 'Seattle', 'Los Angeles']]
+west_coast.head()
+```
+
+
+
+La API de `.plot` de `DataFrame` de Pandas proporciona acceso a varios métodos de visualización. Aquí usaremos `.plot.line`, pero hay otras opciones disponibles (por ejemplo, `.plot.area`, `.plot.bar`, `.plot.nb`, `.plot.scatter`, etc.). Esta API se ha repetido en varias librerías debido a su conveniencia.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+west_coast.plot.line(); # This produces a static Matplotlib plot
+```
+
+***
+
+#### Usando la API de hvPlot `DataFrame.hvplot`
+
+
+
+Importando `hvplot.pandas`, se puede generar un gráfico interactivo similar. La API para `.hvplot` imita esto para `.plot`. Por ejemplo, podemos generar la gráfica de línea anterior usando `.hvplot.line`. En este caso, el _backend_ para los gráficos por defecto es Bokeh, así que el gráfico es _interactivo_.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import hvplot.pandas
+west_coast.hvplot.line() # This produces an interactive Bokeh plot
+```
+
+
+
+La API `.plot` de DataFrame de Pandas proporciona acceso a una serie de métodos de graficación.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+west_coast.hvplot.line(width=600, height=300, grid=True)
+```
+
+
+
+La API `hvplot` también funciona cuando está enlazada junto con otras llamadas del método `DataFrame`. Por ejemplo, podemos muestrear los datos de temperatura y calcular la media para suavizarlos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+smoothed = west_coast.resample('2d').mean()
+smoothed.hvplot.line(width=600, height=300, grid=True)
+```
+
+***
+
+### Graficar desde un `DataArray` con `hvplot.xarray`
+
+
+
+La API `.plot` de Pandas también se extendió a Xarray, es decir, para `DataArray`. de Xarray
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import xarray as xr
+import hvplot.xarray
+import rioxarray as rio
+```
+
+
+
+Para empezar, carga un archivo GeoTIFF local usando `rioxarray` en una estructura Zarray de `DataArray`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+data = rio.open_rasterio(LOCAL_PATH)
+data
+```
+
+
+
+Hacemos algunos cambios menores al `DataArray`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data = data.squeeze() # to reduce 3D array with singleton dimension to 2D array
+data = data.rename({'x':'easting', 'y':'northing'})
+data
+```
+
+***
+
+#### Revisando la API `DataFrame.plot` de Pandas
+
+
+
+La API `DataArray.plot` por defecto usa el `pcolormesh` de Matplotlib para mostrar un arreglo de 2D almacenado dentro de un `DataArray`. La renderización de esta imagen moderadamente de alta resolución lleva un poco de tiempo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.plot(); # by default, uses pcolormesh
+```
+
+***
+
+#### Usando la API de hvPlot `DataFrame.hvplot`
+
+
+
+De nuevo, la API `DataArray.hvplot` imita la API `DataArray.plot`; de forma predeterminada, utiliza una subclase derivada de `holoviews.element.raster.Image`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+plot = data.hvplot() # by default uses Image class
+print(f'{type(plot)=}')
+plot
+```
+
+
+
+El resultado anterior es una visualización interactiva, procesada usando Bokeh. Esto es un poco lento, pero podemos añadir algunas opciones para acelerar la renderización. También se requiere una manipulación de la misma; por ejemplo, la imagen no es cuadrada, el mapa de colores no resalta características útiles, los ejes son transpuestos, etc.
+
+
+
+***
+
+#### Creando opciones para mejorar los gráficos de manera incremental
+
+
+
+Añadamos opciones para mejorar la imagen. Para hacer esto, iniciaremos un diccionario de Python `image_opts` para usar dentro de la llamada al método `image`. Creando opciones para mejorar los gráficos de manera incremental.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict(rasterize=True, dynamic=True)
+pprint(image_opts)
+```
+
+
+
+Para empezar, hagamos la llamada explícita a `hvplot.image` y especifiquemos la secuencia de ejes. Y apliquemos las opciones del diccionario `image_opts`. Utilizaremos la operación `dict-unpacking` `**image_opts` cada vez que invoquemos a `data.hvplot.image`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+plot = data.hvplot.image(x='easting', y='northing', **image_opts)
+plot
+```
+
+
+
+A continuación, vamos a corregir el ratio y las dimensiones de la imagen.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts.update(frame_width=500, frame_height=500, aspect='equal')
+pprint(image_opts)
+plot = data.hvplot.image(x='easting', y='northing', **image_opts)
+plot
+```
+
+
+
+A continuación, vamos a corregir el ratio y las dimensiones de la imagen.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts.update( cmap='hot_r', clim=(0,100), alpha=0.8 )
+pprint(image_opts)
+plot = data.hvplot.image(x='easting', y='northing', **image_opts)
+plot
+```
+
+
+
+Antes de añadir un mapa de base, tenemos que tener en cuenta el sistema de coordenadas. Esto se almacena en el archivo GeoTIFF y, cuando se lee usando `rioxarray.open_rasterio`, se disponibilizada mediante el atributo `data.rio.crs`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+crs = data.rio.crs
+crs
+```
+
+
+
+Podemos usar el CRS recuperado arriba como un argumento opcional para `hvplot.image`. Ten en cuenta que las coordenadas han cambiado en los ejes, pero las etiquetas no son las correctas. Podemos arreglarlo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts.update(crs=crs)
+pprint(image_opts)
+plot = data.hvplot.image(x='easting', y='northing', **image_opts)
+plot
+```
+
+
+
+Ahora vamos a corregir las etiquetas. Utilizaremos el sistema Holoviews/GeoViews `opts` para especificar estas opciones.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+label_opts = dict(title='VEG_ANOM_MAX', xlabel='Longitude (degrees)', ylabel='Latitude (degrees)')
+pprint(image_opts)
+pprint(label_opts)
+plot = data.hvplot.image(x='easting', y='northing', **image_opts).opts(**label_opts)
+plot
+```
+
+
+
+Vamos a superponer la imagen en un mapa base para que podamos ver el terreno debajo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+base = gv.tile_sources.ESRI
+base * plot
+```
+
+
+
+Finalmente, como los píxeles blancos distraen vamos a filtrarlos utilizando el método `DataArray` `where`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+plot = data.where(data>0).hvplot.image(x='easting', y='northing', **image_opts).opts(**label_opts)
+plot * base
+```
+
+
+
+En este cuaderno computacional aplicamos algunas estrategias comunes para generar gráficos. Los usaremos extensamente en el resto del tutorial.
+
+
+
+***
diff --git a/book/es-419/02_Software_Tools_Techniques/04_Constructing_Advanced_Visualizations.md b/book/es-419/02_Software_Tools_Techniques/04_Constructing_Advanced_Visualizations.md
new file mode 100644
index 0000000..0506822
--- /dev/null
+++ b/book/es-419/02_Software_Tools_Techniques/04_Constructing_Advanced_Visualizations.md
@@ -0,0 +1,417 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Construyendo visualizaciones avanzadas
+
+
+
+Vamos a aplicar algunas de las herramientas que hemos visto hasta ahora para obtener algunas visualizaciones más sofisticadas. Estas incluirán el uso de datos vectoriales de un `GeoDataFrame` de _GeoPandas_, se construirán gráficos estáticos y dinámicos a partir de un arreglo 3D y se combinarán datos vectoriales y datos ráster.
+
+Como contexto, los archivos que examinaremos se basan en [el incendio McKinney que ocurrió en el 2022](https://en.wikipedia.org/wiki/McKinney_Fire), en el Bosque Nacional Klamath (al oeste del condado de Siskiyou, California). Los datos vectoriales representan una instantánea del límite de un incendio forestal. Los datos ráster corresponden a la alteración que se observó en la superficie de la vegetación (esto se explicará con mayor detalle más adelante).
+
+
+
+## Importación preliminar y direcciones de los archivos
+
+
+
+Para empezar, se necesitan algunas importaciones típicas de paquetes. También definiremos algunas direcciones a archivos locales que contienen datos geoespaciales relevantes.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+from warnings import filterwarnings
+filterwarnings('ignore')
+from pathlib import Path
+import numpy as np, pandas as pd, xarray as xr
+import geopandas as gpd
+import rioxarray as rio
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Imports for plotting
+import hvplot.pandas, hvplot.xarray
+import geoviews as gv
+from geoviews import opts
+gv.extension('bokeh')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+ASSET_PATH = Path(FILE_STEM, 'assets')
+SHAPE_FILE = ASSET_PATH / 'shapefiles' / 'mckinney' / 'McKinney_NIFC.shp'
+RASTER_FILES = list(ASSET_PATH.glob('OPERA*VEG*.tif'))
+RASTER_FILE = RASTER_FILES[0]
+```
+
+***
+
+## Graficar datos vectoriales desde un `GeoDataFrame`
+
+
+
+
+
+- [hvPlot](https://hvplot.holoviz.org/) está diseñado para extender la API `.plot` de `DataFrames` de Pandas.
+- Funciona para `DataFrames` de Pandas y `DataArrays`/`Datasets` de Xarray.
+
+
+
+***
+
+### Graficar desde un DataFrame con hvplot.pandas
+
+
+
+El código siguiente carga un `DataFrame` de Pandas con datos de temperatura.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import pandas as pd, numpy as np
+from pathlib import Path
+LOCAL_PATH = Path(FILE_STEM, 'assets/temperature.csv')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+df = pd.read_csv(LOCAL_PATH, index_col=0, parse_dates=[0])
+df.head()
+```
+
+***
+
+#### Revisando la API de `DataFrame.plot` de Pandas
+
+
+
+Vamos a extraer un subconjunto de columnas de este `DataFrame` y generar un gráfico.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+west_coast = df[['Vancouver', 'Portland', 'San Francisco', 'Seattle', 'Los Angeles']]
+west_coast.head()
+```
+
+
+
+La API de `.plot` de `DataFrame` de Pandas proporciona acceso a varios métodos de visualización. Aquí usaremos `.plot.line`, pero hay otras opciones disponibles (por ejemplo, `.plot.area`, `.plot.bar`, `.plot.nb`, `.plot.scatter`, etc.). Esta API se ha repetido en varias librerías debido a su conveniencia.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+west_coast.plot.line(); # This produces a static Matplotlib plot
+```
+
+***
+
+#### Usando la API de hvPlot `DataFrame.hvplot`
+
+
+
+Importando `hvplot.pandas`, se puede generar un gráfico interactivo similar. La API para `.hvplot` imita esto para `.plot`. Por ejemplo, podemos generar la gráfica de línea anterior usando `.hvplot.line`. En este caso, el _backend_ para los gráficos por defecto es Bokeh, así que el gráfico es _interactivo_.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import hvplot.pandas
+west_coast.hvplot.line() # This produces an interactive Bokeh plot
+```
+
+
+
+La API `.plot` de DataFrame de Pandas proporciona acceso a una serie de métodos de graficación.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+west_coast.hvplot.line(width=600, height=300, grid=True)
+```
+
+
+
+La API `hvplot` también funciona cuando está enlazada junto con otras llamadas del método `DataFrame`. Por ejemplo, podemos muestrear los datos de temperatura y calcular la media para suavizarlos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+smoothed = west_coast.resample('2d').mean()
+smoothed.hvplot.line(width=600, height=300, grid=True)
+```
+
+***
+
+### Graficar desde un `DataArray` con `hvplot.xarray`
+
+
+
+La API `.plot` de Pandas también se extendió a Xarray, es decir, para `DataArray`. de Xarray
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+import xarray as xr
+import hvplot.xarray
+import rioxarray as rio
+```
+
+
+
+Para empezar, carga un archivo GeoTIFF local usando `rioxarray` en una estructura Zarray de `DataArray`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+LOCAL_PATH = Path(FILE_STEM, 'assets/OPERA_L3_DIST-ALERT-HLS_T10TEM_20220815T185931Z_20220817T153514Z_S2A_30_v0.1_VEG-ANOM-MAX.tif')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+data = rio.open_rasterio(LOCAL_PATH)
+data
+```
+
+
+
+Hacemos algunos cambios menores al `DataArray`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data = data.squeeze() # to reduce 3D array with singleton dimension to 2D array
+data = data.rename({'x':'easting', 'y':'northing'})
+data
+```
+
+***
+
+#### Revisando la API `DataFrame.plot` de Pandas
+
+
+
+La API `DataArray.plot` por defecto usa el `pcolormesh` de Matplotlib para mostrar un arreglo de 2D almacenado dentro de un `DataArray`. La renderización de esta imagen moderadamente de alta resolución lleva un poco de tiempo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+data.plot(); # by default, uses pcolormesh
+```
+
+***
+
+#### Usando la API de hvPlot `DataFrame.hvplot`
+
+
+
+De nuevo, la API `DataArray.hvplot` imita la API `DataArray.plot`; de forma predeterminada, utiliza una subclase derivada de `holoviews.element.raster.Image`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+plot = data.hvplot() # by default uses Image class
+print(f'{type(plot)=}')
+plot
+```
+
+
+
+El resultado anterior es una visualización interactiva, procesada usando Bokeh. Esto es un poco lento, pero podemos añadir algunas opciones para acelerar la renderización. También se requiere una manipulación de la misma; por ejemplo, la imagen no es cuadrada, el mapa de colores no resalta características útiles, los ejes son transpuestos, etc.
+
+
+
+***
+
+#### Creando opciones para mejorar los gráficos de manera incremental
+
+
+
+Añadamos opciones para mejorar la imagen. Para hacer esto, iniciaremos un diccionario de Python `image_opts` para usar dentro de la llamada al método `image`. Creando opciones para mejorar los gráficos de manera incremental.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict(rasterize=True, dynamic=True)
+pprint(image_opts)
+```
+
+
+
+Para empezar, hagamos la llamada explícita a `hvplot.image` y especifiquemos la secuencia de ejes. Y apliquemos las opciones del diccionario `image_opts`. Utilizaremos la operación `dict-unpacking` `**image_opts` cada vez que invoquemos a `data.hvplot.image`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+plot = data.hvplot.image(x='easting', y='northing', **image_opts)
+plot
+```
+
+
+
+A continuación, vamos a corregir el ratio y las dimensiones de la imagen.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts.update(frame_width=500, frame_height=500, aspect='equal')
+pprint(image_opts)
+plot = data.hvplot.image(x='easting', y='northing', **image_opts)
+plot
+```
+
+
+
+A continuación, vamos a corregir el ratio y las dimensiones de la imagen.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts.update( cmap='hot_r', clim=(0,100), alpha=0.8 )
+pprint(image_opts)
+plot = data.hvplot.image(x='easting', y='northing', **image_opts)
+plot
+```
+
+
+
+Antes de añadir un mapa de base, tenemos que tener en cuenta el sistema de coordenadas. Esto se almacena en el archivo GeoTIFF y, cuando se lee usando `rioxarray.open_rasterio`, se disponibilizada mediante el atributo `data.rio.crs`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+crs = data.rio.crs
+crs
+```
+
+
+
+Podemos usar el CRS recuperado arriba como un argumento opcional para `hvplot.image`. Ten en cuenta que las coordenadas han cambiado en los ejes, pero las etiquetas no son las correctas. Podemos arreglarlo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts.update(crs=crs)
+pprint(image_opts)
+plot = data.hvplot.image(x='easting', y='northing', **image_opts)
+plot
+```
+
+
+
+Ahora vamos a corregir las etiquetas. Utilizaremos el sistema Holoviews/GeoViews `opts` para especificar estas opciones.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+label_opts = dict(title='VEG_ANOM_MAX', xlabel='Longitude (degrees)', ylabel='Latitude (degrees)')
+pprint(image_opts)
+pprint(label_opts)
+plot = data.hvplot.image(x='easting', y='northing', **image_opts).opts(**label_opts)
+plot
+```
+
+
+
+Vamos a superponer la imagen en un mapa base para que podamos ver el terreno debajo.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+base = gv.tile_sources.ESRI
+base * plot
+```
+
+
+
+Finalmente, como los píxeles blancos distraen vamos a filtrarlos utilizando el método `DataArray` `where`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+plot = data.where(data>0).hvplot.image(x='easting', y='northing', **image_opts).opts(**label_opts)
+plot * base
+```
+
+
+
+En este cuaderno computacional aplicamos algunas estrategias comunes para generar gráficos. Los usaremos extensamente en el resto del tutorial.
+
+
+
+***
diff --git a/book/es-419/02_Software_Tools_Techniques/04_Constructing_Advanced_Visualizations.md b/book/es-419/02_Software_Tools_Techniques/04_Constructing_Advanced_Visualizations.md
new file mode 100644
index 0000000..0506822
--- /dev/null
+++ b/book/es-419/02_Software_Tools_Techniques/04_Constructing_Advanced_Visualizations.md
@@ -0,0 +1,417 @@
+---
+jupyter:
+ jupytext:
+ text_representation:
+ extension: .md
+ format_name: markdown
+ format_version: "1.3"
+ jupytext_version: 1.16.2
+ kernelspec:
+ display_name: Python 3 (ipykernel)
+ language: python
+ name: python3
+---
+
+# Construyendo visualizaciones avanzadas
+
+
+
+Vamos a aplicar algunas de las herramientas que hemos visto hasta ahora para obtener algunas visualizaciones más sofisticadas. Estas incluirán el uso de datos vectoriales de un `GeoDataFrame` de _GeoPandas_, se construirán gráficos estáticos y dinámicos a partir de un arreglo 3D y se combinarán datos vectoriales y datos ráster.
+
+Como contexto, los archivos que examinaremos se basan en [el incendio McKinney que ocurrió en el 2022](https://en.wikipedia.org/wiki/McKinney_Fire), en el Bosque Nacional Klamath (al oeste del condado de Siskiyou, California). Los datos vectoriales representan una instantánea del límite de un incendio forestal. Los datos ráster corresponden a la alteración que se observó en la superficie de la vegetación (esto se explicará con mayor detalle más adelante).
+
+
+
+## Importación preliminar y direcciones de los archivos
+
+
+
+Para empezar, se necesitan algunas importaciones típicas de paquetes. También definiremos algunas direcciones a archivos locales que contienen datos geoespaciales relevantes.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+from warnings import filterwarnings
+filterwarnings('ignore')
+from pathlib import Path
+import numpy as np, pandas as pd, xarray as xr
+import geopandas as gpd
+import rioxarray as rio
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+# Imports for plotting
+import hvplot.pandas, hvplot.xarray
+import geoviews as gv
+from geoviews import opts
+gv.extension('bokeh')
+```
+
+```{code-cell} python jupyter={"source_hidden": false}
+FILE_STEM = Path.cwd().parent if 'book' == Path.cwd().parent.stem else 'book'
+ASSET_PATH = Path(FILE_STEM, 'assets')
+SHAPE_FILE = ASSET_PATH / 'shapefiles' / 'mckinney' / 'McKinney_NIFC.shp'
+RASTER_FILES = list(ASSET_PATH.glob('OPERA*VEG*.tif'))
+RASTER_FILE = RASTER_FILES[0]
+```
+
+***
+
+## Graficar datos vectoriales desde un `GeoDataFrame`
+
+
+
+ +
+El paquete [GeoPandas](https://geopandas.org/en/stable/index.html) contiene muchas herramientas útiles para trabajar con datos geoespaciales vectoriales. En particular, el [`GeoDataFrame` de GeoPandas](https://geopandas.org/en/stable/docs/reference/api/geopandas.GeoDataFrame.html) es una subclase del `DataFrame` de Pandas que está específicamente diseñado, por ejemplo, para datos vectoriales almacenados en _shapefiles_.
+
+Como ejemplo, vamos a cargar algunos datos vectoriales desde la ruta local `SHAPEFILE` (tal como se definió anteriormente).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shape_df = gpd.read_file(SHAPE_FILE)
+shape_df
+```
+
+
+
+El objeto `shape_df` es un [`GeoDataFrame`](https://geopandas.org/en/stable/docs/reference/api/geopandas.GeoDataFrame.html) que tiene más de dos docenas de columnas de metadatos en una sola fila. La columna principal que nos interesa es la de `geometry`. Esta columna almacena las coordenadas de los vértices de un `MULTIPOLYGON`, es decir, un conjunto de polígonos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shape_df.geometry
+```
+
+
+
+Los vértices de los polígonos parecen expresarse como pares de `(longitude, latitude)`. Podemos determinar qué sistema de coordenadas específico se utiliza examinando el atributo `crs` del `GeoDataFrame`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(shape_df.crs)
+```
+
+
+
+Utilicemos `hvplot` para generar un gráfico de este conjunto de datos vectoriales.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shape_df.hvplot()
+```
+
+
+
+La proyección en este gráfico es un poco extraña. La documentación de HoloViz incluye una [discusión sobre las consideraciones cuando se visualizan datos geográficos](https://hvplot.holoviz.org/user_guide/Geographic_Data.html). El punto importante a recordar en este contexto es que la opción `geo=True` es útil.
+
+Vamos a crear dos diccionarios de Python, `shapeplot_opts` y `layout_opts`, y construiremos una visualización.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shapeplot_opts= dict(geo=True)
+layout_opts = dict(xlabel='Longitude', ylabel="Latitude")
+print(f"{shapeplot_opts=}")
+print(f"{layout_opts=}")
+
+shape_df.hvplot(**shapeplot_opts).opts(**layout_opts)
+```
+
+
+
+La documentación de HoloViz incluye una [discusión sobre las consideraciones cuando se grafican datos geográficos](https://hvplot.holoviz.org/user_guide/Geographic_Data.html). El punto importante que se debe recordar en el contexto inmediato es que la opción `geo=True` es útil.
+
+- Definir `color=None` significa que los polígonos no se rellenarán.
+- Al especificar `line_width` y `line_color` se modifica la apariencia de los límites de los polígonos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shapeplot_opts.update( color=None ,
+ line_width=2,
+ line_color='red')
+print(shapeplot_opts)
+
+shape_df.hvplot(**shapeplot_opts).opts(**layout_opts)
+```
+
+
+
+Rellenemos los polígonos con color naranja utilizando la opción `color=orange` y hagamos que el área rellenada sea parcialmente transparente especificando un valor para `alpha` entre cero y uno.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shapeplot_opts.update(color='orange', alpha=0.25)
+print(shapeplot_opts)
+
+shape_df.hvplot(**shapeplot_opts).opts(**layout_opts)
+```
+
+### Agregado de un mapa base
+
+
+
+A continuación, proporcionemos un mapa base y superpongamos los polígonos trazados utilizando el operador `*`. Observa el uso de paréntesis para aplicar el método `opts` a la salida del operador `*`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+basemap = gv.tile_sources.ESRI(height=500, width=500, padding=0.1)
+
+(shape_df.hvplot(**shapeplot_opts) * basemap).opts(**layout_opts)
+```
+
+
+
+El mapa base no necesita ser superpuesto como un objeto separado, puede especificarse utilizando la palabra clave `tiles`. Por ejemplo, al establecer `tiles='OSM'` se utilizan los mosaicos de [OpenStreetMap](ttps://www.openstreetmap.org). Observa que las dimensiones de la imagen renderizada probablemente no sean las mismas que las de la imagen anterior con los mosaicos [ESRI](https://www.esri.com), ya que antes especificamos explícitamente la altura de 500, con la opción `height=500`, y el ancho de 500, con la opción `width=500`, en la definición del `basemap`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shapeplot_opts.update(tiles='OSM')
+shape_df.hvplot(**shapeplot_opts).opts(**layout_opts)
+```
+
+
+
+Vamos a eliminar la opción `tiles` de `shapeplot_opts` y a vincular el objeto del gráfico resultante al identificador `shapeplot`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+del shapeplot_opts['tiles']
+print(shapeplot_opts)
+
+shapeplot = shape_df.hvplot(**shapeplot_opts)
+shapeplot
+```
+
+### Combinación de datos vectoriales con datos ráster en un gráfico estático
+
+
+
+Combinemos estos datos vectoriales con algunos datos ráster. Cargaremos los datos raster desde un archivo local utilizando la función `rioxarray.open_rasterio`. Por practicidad, usaremos el encadenamiento de métodos para reetiquetar las coordenadas del `DataArray` cargado y usaremos el método `squeeze` para convertir el arreglo tridimensional que se cargó en uno bidimensional.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster = (
+ rio.open_rasterio(RASTER_FILE)
+ .rename(dict(x='longitude', y='latitude'))
+ .squeeze()
+ )
+raster
+```
+
+
+
+Utilizaremos un diccionario de Python `image_opts` para almacenar argumentos útiles y pasarlos a `hvplot.image`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict(
+ x='longitude',
+ y='latitude',
+ rasterize=True,
+ dynamic=True,
+ cmap='hot_r',
+ clim=(0, 100),
+ alpha=0.8,
+ project=True,
+ )
+
+raster.hvplot.image(**image_opts)
+```
+
+
+
+Podemos superponer `shapeplot` con los datos ráster graficados utilizando el operador `*`. Podemos utilizar las herramientas `Pan`, `Wheel Zoom` y `Box Zoom` en la barra de herramientas de Bokeh a la derecha del gráfico para hacer _zoom_ y comprobar que los datos vectoriales fueron trazados encima de los datos ráster.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster.hvplot.image(**image_opts) * shapeplot
+```
+
+
+
+Además, podemos superponer los datos vectoriales y ráster en mosaicos ESRI utilizando `basemap`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster.hvplot.image(**image_opts) * shapeplot * basemap
+```
+
+
+
+Observa que muchos de los píxeles blancos oscurecen el gráfico. Resulta que estos píxeles corresponden a ceros en los datos ráster, los cuales pueden ser ignorados sin problema. Podemos filtrarlos utilizando el método `where`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster = raster.where((raster!=0))
+layout_opts.update(title="McKinney 2022 Wildfires")
+
+(raster.hvplot.image(**image_opts) * shapeplot * basemap).opts(**layout_opts)
+```
+
+***
+
+## Construcción de gráficos estáticos a partir de un arreglo 3D
+
+
+
+Vamos a cargar una secuencia de archivos ráster en un arreglo tridimensional y generaremos algunos gráficos. Para empezar, construiremos un bucle para leer `DataArrays` de los archivos `RASTER_FILES` y utilizaremos `xarray.concat` para generar un único arreglo de rásters tridimensional (es decir, tres rásters de $3,600\times3,600$ apilados verticalmente). Aprenderemos las interpretaciones específicas asociadas con el conjunto de datos ráster en un cuaderno computacional posterior. Por el momento, los trataremos como datos sin procesar con los que experimentaremos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack = []
+for path in RASTER_FILES:
+ data = rio.open_rasterio(path).rename(dict(x='longitude', y='latitude'))
+ band_name = path.stem.split('_')[-1]
+ data.coords.update({'band': [band_name]})
+ data.attrs = dict(description=f"OPERA DIST product", units=None)
+ stack.append(data)
+
+stack = xr.concat(stack, dim='band')
+stack = stack.where(stack!=0)
+```
+
+
+
+Renombramos los ejes `longitude` y `latitude` y filtramos los píxeles con valor cero para simplificar la visualización posterior.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack
+```
+
+
+
+Una vez que el `stack` de `DataArray` esté construido, podemos enfocarnos en la visualización.
+
+Si queremos generar un gráfico estático con varias imágenes, podemos utilizar `hvplot.image` junto con el método `.layout`. Para ver cómo funciona, empecemos por redefinir los diccionarios `image_opts` y `layout_opts`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict( x='longitude',
+ y='latitude',
+ rasterize=True,
+ dynamic=True,
+ cmap='hot_r',
+ crs=stack.rio.crs,
+ alpha=0.8,
+ project=True,
+ aspect='equal',
+ shared_axes=False,
+ colorbar=True,
+ tiles='ESRI',
+ padding=0.1)
+layout_opts = dict(xlabel='Longitude', ylabel="Latitude")
+```
+
+
+
+Para acelerar el renderizado, construiremos inicialmente un objeto `view` que seleccione subconjuntos de píxeles. Definimos inicialmente el parámetro `steps=200` para restringir la vista a cada 200 píxeles en cualquier dirección. Si reducimos los `steps`, se tarda más en renderizar. Establecer `steps=1` o `steps=None` equivale a seleccionar todos los píxeles.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 200
+subset = slice(0, None, steps)
+layout_opts.update(frame_width=250, frame_height=250)
+
+
+view = stack.isel(latitude=subset, longitude=subset)
+view.hvplot.image(**image_opts).opts(**layout_opts).layout()
+```
+
+
+
+El método `layout` traza de manera predeterminada cada uno de los tres rásteres seleccionados a lo largo del eje `band` horizontalmente.
+
+
+
+***
+
+## Construcción de una vista dinámica a partir de un arreglo 3D
+
+
+
+Otra forma de visualizar un arreglo tridimensional es asociar un _widget_ de selección a uno de los ejes. Si llamamos a `hvplot.image` sin agregar el método `.layout` el resultado es un _mapa dinámico_. En este caso, el _widget_ de selección nos permite elegir cortes a lo largo del eje `band`.
+
+Una vez más, aumentar el valor del parámetro `steps` reduce el tiempo de renderizado. Reducirlo a `1` o a `None` renderiza a resolución completa.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 200
+subset = slice(0, None, steps)
+layout_opts.update(frame_height=400, frame_width=400)
+
+view = stack.isel(latitude=subset, longitude=subset)
+view.hvplot.image(**image_opts).opts(**layout_opts)
+```
+
+
+
+Más adelante apilaremos muchos rásters con distintas marcas temporales a lo largo de un eje `time`. Cuando haya muchos cortes, el _widget_ de selección se renderizará como un deslizador en vez de como un menú desplegable. Podemos controlar la ubicación del _widget_ utilizando una opción de palabra clave `widget_location`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+view.hvplot.image(widget_location='bottom_left', **image_opts, **layout_opts)
+```
+
+
+
+Observa que agregar la opción `widget_location` modifica ligeramente la secuencia en la que se especifican las opciones. Es decir, si invocamos algo como
+
+```python
+view.hvplot.image(widget_location='top_left', **image_opts).opts(**layout_opts)
+```
+
+se genera una excepción (de ahí la invocación en la celda de código anterior). Hay algunas dificultades sutiles en la elaboración de la secuencia correcta para aplicar opciones al personalizar objetos HoloViz/Hvplot (principalmente debido a las formas en que los espacios de los nombres se superponen con Bokeh u otros _backends_ de renderizado). La mejor estrategia es empezar con el menor número de opciones posible y experimentar añadiendo opciones de forma incremental hasta que obtengamos la visualización final que deseamos.
+
+
+
+***
+
+### Combinación de datos vectoriales con datos ráster en una vista dinámica.
+
+
+
+Por último, vamos a superponer nuestros datos vectoriales anteriores, los límites del incendio forestal, con la vista dinámica de este arreglo tridimensional de rásteres. Podemos utilizar el operador `*` para combinar la salida de `hvplot.image` con `shapeplot`, la vista renderizada de los datos vectoriales.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 200
+subset = slice(0, None, steps)
+view = stack.isel(latitude=subset, longitude=subset)
+
+image_opts.update(colorbar=False)
+layout_opts.update(frame_height=500, frame_width=500)
+(view.hvplot.image(**image_opts) * shapeplot).opts(**layout_opts)
+```
+
+
+
+Una vez más, la correcta especificación de las opciones puede requerir un poco de experimentación. Las ideas importantes que se deben tomar en cuenta aquí son:
+
+- cómo cargar conjuntos de datos relevantes con `geopandas` and `rioxarray`, y
+- cómo utilizar `hvplot` de forma interactiva e incremental para generar visualizaciones atractivas.
+
+
+
+***
+
+El paquete [GeoPandas](https://geopandas.org/en/stable/index.html) contiene muchas herramientas útiles para trabajar con datos geoespaciales vectoriales. En particular, el [`GeoDataFrame` de GeoPandas](https://geopandas.org/en/stable/docs/reference/api/geopandas.GeoDataFrame.html) es una subclase del `DataFrame` de Pandas que está específicamente diseñado, por ejemplo, para datos vectoriales almacenados en _shapefiles_.
+
+Como ejemplo, vamos a cargar algunos datos vectoriales desde la ruta local `SHAPEFILE` (tal como se definió anteriormente).
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shape_df = gpd.read_file(SHAPE_FILE)
+shape_df
+```
+
+
+
+El objeto `shape_df` es un [`GeoDataFrame`](https://geopandas.org/en/stable/docs/reference/api/geopandas.GeoDataFrame.html) que tiene más de dos docenas de columnas de metadatos en una sola fila. La columna principal que nos interesa es la de `geometry`. Esta columna almacena las coordenadas de los vértices de un `MULTIPOLYGON`, es decir, un conjunto de polígonos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shape_df.geometry
+```
+
+
+
+Los vértices de los polígonos parecen expresarse como pares de `(longitude, latitude)`. Podemos determinar qué sistema de coordenadas específico se utiliza examinando el atributo `crs` del `GeoDataFrame`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+print(shape_df.crs)
+```
+
+
+
+Utilicemos `hvplot` para generar un gráfico de este conjunto de datos vectoriales.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shape_df.hvplot()
+```
+
+
+
+La proyección en este gráfico es un poco extraña. La documentación de HoloViz incluye una [discusión sobre las consideraciones cuando se visualizan datos geográficos](https://hvplot.holoviz.org/user_guide/Geographic_Data.html). El punto importante a recordar en este contexto es que la opción `geo=True` es útil.
+
+Vamos a crear dos diccionarios de Python, `shapeplot_opts` y `layout_opts`, y construiremos una visualización.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shapeplot_opts= dict(geo=True)
+layout_opts = dict(xlabel='Longitude', ylabel="Latitude")
+print(f"{shapeplot_opts=}")
+print(f"{layout_opts=}")
+
+shape_df.hvplot(**shapeplot_opts).opts(**layout_opts)
+```
+
+
+
+La documentación de HoloViz incluye una [discusión sobre las consideraciones cuando se grafican datos geográficos](https://hvplot.holoviz.org/user_guide/Geographic_Data.html). El punto importante que se debe recordar en el contexto inmediato es que la opción `geo=True` es útil.
+
+- Definir `color=None` significa que los polígonos no se rellenarán.
+- Al especificar `line_width` y `line_color` se modifica la apariencia de los límites de los polígonos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shapeplot_opts.update( color=None ,
+ line_width=2,
+ line_color='red')
+print(shapeplot_opts)
+
+shape_df.hvplot(**shapeplot_opts).opts(**layout_opts)
+```
+
+
+
+Rellenemos los polígonos con color naranja utilizando la opción `color=orange` y hagamos que el área rellenada sea parcialmente transparente especificando un valor para `alpha` entre cero y uno.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shapeplot_opts.update(color='orange', alpha=0.25)
+print(shapeplot_opts)
+
+shape_df.hvplot(**shapeplot_opts).opts(**layout_opts)
+```
+
+### Agregado de un mapa base
+
+
+
+A continuación, proporcionemos un mapa base y superpongamos los polígonos trazados utilizando el operador `*`. Observa el uso de paréntesis para aplicar el método `opts` a la salida del operador `*`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+basemap = gv.tile_sources.ESRI(height=500, width=500, padding=0.1)
+
+(shape_df.hvplot(**shapeplot_opts) * basemap).opts(**layout_opts)
+```
+
+
+
+El mapa base no necesita ser superpuesto como un objeto separado, puede especificarse utilizando la palabra clave `tiles`. Por ejemplo, al establecer `tiles='OSM'` se utilizan los mosaicos de [OpenStreetMap](ttps://www.openstreetmap.org). Observa que las dimensiones de la imagen renderizada probablemente no sean las mismas que las de la imagen anterior con los mosaicos [ESRI](https://www.esri.com), ya que antes especificamos explícitamente la altura de 500, con la opción `height=500`, y el ancho de 500, con la opción `width=500`, en la definición del `basemap`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+shapeplot_opts.update(tiles='OSM')
+shape_df.hvplot(**shapeplot_opts).opts(**layout_opts)
+```
+
+
+
+Vamos a eliminar la opción `tiles` de `shapeplot_opts` y a vincular el objeto del gráfico resultante al identificador `shapeplot`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+del shapeplot_opts['tiles']
+print(shapeplot_opts)
+
+shapeplot = shape_df.hvplot(**shapeplot_opts)
+shapeplot
+```
+
+### Combinación de datos vectoriales con datos ráster en un gráfico estático
+
+
+
+Combinemos estos datos vectoriales con algunos datos ráster. Cargaremos los datos raster desde un archivo local utilizando la función `rioxarray.open_rasterio`. Por practicidad, usaremos el encadenamiento de métodos para reetiquetar las coordenadas del `DataArray` cargado y usaremos el método `squeeze` para convertir el arreglo tridimensional que se cargó en uno bidimensional.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster = (
+ rio.open_rasterio(RASTER_FILE)
+ .rename(dict(x='longitude', y='latitude'))
+ .squeeze()
+ )
+raster
+```
+
+
+
+Utilizaremos un diccionario de Python `image_opts` para almacenar argumentos útiles y pasarlos a `hvplot.image`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict(
+ x='longitude',
+ y='latitude',
+ rasterize=True,
+ dynamic=True,
+ cmap='hot_r',
+ clim=(0, 100),
+ alpha=0.8,
+ project=True,
+ )
+
+raster.hvplot.image(**image_opts)
+```
+
+
+
+Podemos superponer `shapeplot` con los datos ráster graficados utilizando el operador `*`. Podemos utilizar las herramientas `Pan`, `Wheel Zoom` y `Box Zoom` en la barra de herramientas de Bokeh a la derecha del gráfico para hacer _zoom_ y comprobar que los datos vectoriales fueron trazados encima de los datos ráster.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster.hvplot.image(**image_opts) * shapeplot
+```
+
+
+
+Además, podemos superponer los datos vectoriales y ráster en mosaicos ESRI utilizando `basemap`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster.hvplot.image(**image_opts) * shapeplot * basemap
+```
+
+
+
+Observa que muchos de los píxeles blancos oscurecen el gráfico. Resulta que estos píxeles corresponden a ceros en los datos ráster, los cuales pueden ser ignorados sin problema. Podemos filtrarlos utilizando el método `where`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+raster = raster.where((raster!=0))
+layout_opts.update(title="McKinney 2022 Wildfires")
+
+(raster.hvplot.image(**image_opts) * shapeplot * basemap).opts(**layout_opts)
+```
+
+***
+
+## Construcción de gráficos estáticos a partir de un arreglo 3D
+
+
+
+Vamos a cargar una secuencia de archivos ráster en un arreglo tridimensional y generaremos algunos gráficos. Para empezar, construiremos un bucle para leer `DataArrays` de los archivos `RASTER_FILES` y utilizaremos `xarray.concat` para generar un único arreglo de rásters tridimensional (es decir, tres rásters de $3,600\times3,600$ apilados verticalmente). Aprenderemos las interpretaciones específicas asociadas con el conjunto de datos ráster en un cuaderno computacional posterior. Por el momento, los trataremos como datos sin procesar con los que experimentaremos.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack = []
+for path in RASTER_FILES:
+ data = rio.open_rasterio(path).rename(dict(x='longitude', y='latitude'))
+ band_name = path.stem.split('_')[-1]
+ data.coords.update({'band': [band_name]})
+ data.attrs = dict(description=f"OPERA DIST product", units=None)
+ stack.append(data)
+
+stack = xr.concat(stack, dim='band')
+stack = stack.where(stack!=0)
+```
+
+
+
+Renombramos los ejes `longitude` y `latitude` y filtramos los píxeles con valor cero para simplificar la visualización posterior.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+stack
+```
+
+
+
+Una vez que el `stack` de `DataArray` esté construido, podemos enfocarnos en la visualización.
+
+Si queremos generar un gráfico estático con varias imágenes, podemos utilizar `hvplot.image` junto con el método `.layout`. Para ver cómo funciona, empecemos por redefinir los diccionarios `image_opts` y `layout_opts`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+image_opts = dict( x='longitude',
+ y='latitude',
+ rasterize=True,
+ dynamic=True,
+ cmap='hot_r',
+ crs=stack.rio.crs,
+ alpha=0.8,
+ project=True,
+ aspect='equal',
+ shared_axes=False,
+ colorbar=True,
+ tiles='ESRI',
+ padding=0.1)
+layout_opts = dict(xlabel='Longitude', ylabel="Latitude")
+```
+
+
+
+Para acelerar el renderizado, construiremos inicialmente un objeto `view` que seleccione subconjuntos de píxeles. Definimos inicialmente el parámetro `steps=200` para restringir la vista a cada 200 píxeles en cualquier dirección. Si reducimos los `steps`, se tarda más en renderizar. Establecer `steps=1` o `steps=None` equivale a seleccionar todos los píxeles.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 200
+subset = slice(0, None, steps)
+layout_opts.update(frame_width=250, frame_height=250)
+
+
+view = stack.isel(latitude=subset, longitude=subset)
+view.hvplot.image(**image_opts).opts(**layout_opts).layout()
+```
+
+
+
+El método `layout` traza de manera predeterminada cada uno de los tres rásteres seleccionados a lo largo del eje `band` horizontalmente.
+
+
+
+***
+
+## Construcción de una vista dinámica a partir de un arreglo 3D
+
+
+
+Otra forma de visualizar un arreglo tridimensional es asociar un _widget_ de selección a uno de los ejes. Si llamamos a `hvplot.image` sin agregar el método `.layout` el resultado es un _mapa dinámico_. En este caso, el _widget_ de selección nos permite elegir cortes a lo largo del eje `band`.
+
+Una vez más, aumentar el valor del parámetro `steps` reduce el tiempo de renderizado. Reducirlo a `1` o a `None` renderiza a resolución completa.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 200
+subset = slice(0, None, steps)
+layout_opts.update(frame_height=400, frame_width=400)
+
+view = stack.isel(latitude=subset, longitude=subset)
+view.hvplot.image(**image_opts).opts(**layout_opts)
+```
+
+
+
+Más adelante apilaremos muchos rásters con distintas marcas temporales a lo largo de un eje `time`. Cuando haya muchos cortes, el _widget_ de selección se renderizará como un deslizador en vez de como un menú desplegable. Podemos controlar la ubicación del _widget_ utilizando una opción de palabra clave `widget_location`.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+view.hvplot.image(widget_location='bottom_left', **image_opts, **layout_opts)
+```
+
+
+
+Observa que agregar la opción `widget_location` modifica ligeramente la secuencia en la que se especifican las opciones. Es decir, si invocamos algo como
+
+```python
+view.hvplot.image(widget_location='top_left', **image_opts).opts(**layout_opts)
+```
+
+se genera una excepción (de ahí la invocación en la celda de código anterior). Hay algunas dificultades sutiles en la elaboración de la secuencia correcta para aplicar opciones al personalizar objetos HoloViz/Hvplot (principalmente debido a las formas en que los espacios de los nombres se superponen con Bokeh u otros _backends_ de renderizado). La mejor estrategia es empezar con el menor número de opciones posible y experimentar añadiendo opciones de forma incremental hasta que obtengamos la visualización final que deseamos.
+
+
+
+***
+
+### Combinación de datos vectoriales con datos ráster en una vista dinámica.
+
+
+
+Por último, vamos a superponer nuestros datos vectoriales anteriores, los límites del incendio forestal, con la vista dinámica de este arreglo tridimensional de rásteres. Podemos utilizar el operador `*` para combinar la salida de `hvplot.image` con `shapeplot`, la vista renderizada de los datos vectoriales.
+
+
+
+```{code-cell} python jupyter={"source_hidden": false}
+steps = 200
+subset = slice(0, None, steps)
+view = stack.isel(latitude=subset, longitude=subset)
+
+image_opts.update(colorbar=False)
+layout_opts.update(frame_height=500, frame_width=500)
+(view.hvplot.image(**image_opts) * shapeplot).opts(**layout_opts)
+```
+
+
+
+Una vez más, la correcta especificación de las opciones puede requerir un poco de experimentación. Las ideas importantes que se deben tomar en cuenta aquí son:
+
+- cómo cargar conjuntos de datos relevantes con `geopandas` and `rioxarray`, y
+- cómo utilizar `hvplot` de forma interactiva e incremental para generar visualizaciones atractivas.
+
+
+
+***

